Im Beitrag über das Corona-Virus wurde erwähnt, dass es auch sogenannte endogene Retroviren gibt. Zur Erinnerung: Endogene Viren können ihr Genom in das Genom des Wirtes einpflanzen und über die Keimzellen an die nächste Generation vererben.
Diese endogenen Viren machen tatsächlich einen beachtlichen Anteil unseres Genoms aus.
Der DNA-Strang eines Menschen besitzt etwa 3 Milliarden Basenpaare, hat also vereinfacht ausgedrückt 3 Mrd. Buchstaben der entsprechenden Basen A, T, C und G. Nur als Vergleich: Die Bibel, welches nun sicherlich kein dünnes Buch ist, hat etwa 4,4 Millionen Zeichen – Leerzeichen & Sonderzeichen inklusive – das entspricht 0,14% der Basenpaare der DNA. Aber nur ein Bruchteil unserer DNA besteht aus Genen. Gene definieren sich dadurch, dass sie die Information für den Aufbau von Proteinen haben inklusive ihrer Regulationssequenzen – also jene Abschnitte die mitkontrollieren, wann ein Gen ein- oder ausgeschaltet wird. Das menschliche Genom hat etwa 20.000 Gene. Das klingt viel – beträgt aber nur 1,5% der Gesamt-DNA. Der Rest besteht aus sog. nicht-codierter DNA.
Wie groß ist der Anteil der endogenen Viren im menschlichen Genom? Im Rahmen des Humangenomprojekts wurden im menschlichen Genom mehrere tausend endogene Retrovieren gefunden, die zunächst in 24 verschiedene „Familien“ eingeteilt werden und etwa 8 % des menschlichen Genoms ausmachen (Lander et al. 2001, Vgl. Abb. 1).
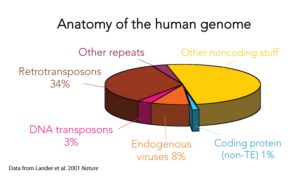
Abb. 1: menschliches Genom
Neueren Erkenntnissen zufolge sind es bereits 31 verschiedene Gruppen, die jeweils durch ein einzelnes Integrationsereignis entstanden sind (Holmes 2007, Gifford 2006).
So ein endogenes Retrovirus ist nach der Infektion erstmal nur bei einem Individuum vorhanden, und wenn es sich irgendwie schädlich bemerkbar macht, verschwindet es bald wieder durch natürliche Selektion. Wenn es keinen Schaden anrichtet, kann es sich in der ganzen Population verbreiten und schließlich fixiert werden. “Fixiert” heißt, dass dann alle Vertreter einer Art dieses endogene Retrovirus in ihrer DNA haben.
Ein Retrovirus hat mindestens die drei Gene gag, pol und env (Vgl. Abb. 2). pol kodiert unter anderem die Reverse Transkriptase, und env kodiert die Hülle. Hinzu kommen die LTR-Sequenzen, die an beiden Enden des Genoms des Provirus vorkommen. LTR steht für “long terminal repeat”. Die beiden LTRs sind zum Einfügezeitpunkt identisch aufgebaut, und das in Ableserichtung vordere von ihnen dient als Starter. Es soll also die Zelle dazu bewegen, die Gene abzulesen. Die Einfügestelle eines Retrovirus in die DNA ist weitgehend zufällig. Wenn man endogene Retroviren bei verschiedenen Tierarten an einander entsprechenden Stellen findet, kann man also davon ausgehen, dass mal ein Vorfahre beider Arten vom Virus infiziert wurde. Im menschlichen Genom dürften sich etwa 400.000 Einfügestellen finden, oft ist aber nur die LTR-Region übrig (Finlay 2006).
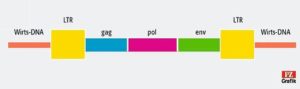
Abb. 2: Genom eines endogenen Retrovirus
2006 untersuchte ein Forscherteam 425 vollständige Retroviren des Schimpansen. 384 davon fand man auch bei Menschen an übereinstimmenden Stellen. Bei Menschen und Gorillas gibt es weniger Übereinstimmungen und bei Menschen und Rhesusäffchen noch weniger. Wenn man aus der Verteilung von endogenen Retroviren über die Affenarten einen Stammbaum konstruiert, dann entspricht er dem Stammbaum, den man auch aus anderen Merkmalen abgeleitet hat. (Polavarapu et al. 2006, Theobald 2004)
Ein anderes Forscherteam machte etwas anderes. Sie nahmen nur die endogenen Retroviren, die bei allen untersuchten Affen an der gleichen Position zu finden waren, und betrachteten dort die Mutationen an den LTRs. Aus den mit der Zeit angesammelten und weitervererbten Punktmutationen der LTRs erhielten sie für jedes einzelne Provirus, das nicht allzu schwere Beschädigungen aufwies, wieder den schon bekannten Stammbaum der Primaten (Johnson & Coffin 1999, Hughes & Coffin 2005).
Ein endogenes Retrovirus, das bei Gorillas, Bonobos und Schimpansen vorhanden ist, aber nicht bei Menschen, wurde in den wissenschaftlichen Zeitschriften analysiert (Barbulescu et al. 2001). Die Erklärung ist, dass das Provirus bei der Abspaltung des Gorillas und auch bei der Trennung von Mensch und Schimpanse noch vorhanden war und aus der menschlichen Linie verschwand, aber bei den anderen Arten fixiert wurde.
Hier also das spannende: Endogene Retroviren sind ein Beweis für die gemeinsame Abstammung.
Solche endogenen Retroviren sind oftmals funktionslos, manchmal schädlich, hin und wieder aber auch nützlich. Zum Beispiel wird das Einnisten der Eizelle in die Gebärmutter von einem viralen Gen begünstigt, das die Abstoßung verhindert, was ja für Viren und Eizellen gleichermaßen günstig ist. Wenn eine Eizelle befruchtet wird und sich auf ihren Weg in die mütterliche Gebärmutter macht, lebt sie gefährlich. Denn im Prinzip ist sie ein Fremdkörper, stammen doch zumindest die Hälfte ihrer Gene nicht von der Mutter sondern vom Vater und damit von einem anderen Menschen. Kritisch wird dieser Status vor allem dann, wenn es um die Einnistung des befruchteten Eis in die Gebärmutterschleimhaut geht.
Entscheidend für den Erfolg dieser Operation ist die Plazenta, ihre Gewebestruktur bildet sowohl nährendes Bindeglied als auch Schutz für den heranwachsenden Embryo. Erreicht wird dies durch die Ausbildung einer speziellen Grenzschicht, dem so genannten Syncytiotrophoblast. In ihm verschmelzen Plazentazellen zu einer Art Riesenzelle mit vielen Zellkernen. Eine Studie entdeckte, dass Retroviren bei der Bildung dieser Riesenzellen beteiligt sein könnten. Sie entdecken ein Protein, von ihnen Syncytin getauft, das den entscheidenden Anstoß zur Verschmelzung der Plazentazellen gibt. Doch das Protein wird nicht von einem unserer eigenen kodierenden Gene produziert, sondern von dem eines endogenen Retrovirus (Mi et al. 2000, Lavialle et al. 2013).
Transposons – Springende Gene
Es gibt aber einen weiteren Bereich unseres Genoms, der jenen der endogenen Retroviren ähnelt. Es handelt sich um die sogenannten Transposons oder springenden Gene. Das sind DNA-Sequenzen, die von einer Stelle des Genoms zu einer anderen springen können.
Diese Elemente wurden erstmals in den 1950er Jahren von der Genetikerin Barbara McClintock identifiziert (McClintock 1951). Biologen standen McClintocks Entdeckung zunächst skeptisch gegenüber. In den nächsten Jahrzehnten stellte sich jedoch heraus, dass Transposons nicht nur “springen”, sondern auch in fast allen Organismen (sowohl Prokaryoten als auch Eukaryoten) und typischerweise in großer Zahl vorkommen. Z. B. besteht 50% des menschlichen Genoms aus solchen Transposons und beim Mais sind es bis zu 90% (SanMiguel, 1996). Jedoch ist nur ein geringer Bruchteil der menschlichen Transposon-Sequenz zum „Springen“ fähig, etwa 0,05% (Mills et al. 2007).
Die Transposons im Genom kann man ebenso weiter unterteilen. Man unterscheidet Transposons, deren mobile Zwischenstufe von RNA gebildet wird (Retroelemente oder Klasse-I-Transposon), von denjenigen, deren mobile Phase DNA ist (DNA-Transposon oder Klasse-II-Transposon) (vgl. Abb. 3).
Der weitaus größte Teil des menschlichen Genoms, etwa 98%, sind Retrotransposons, die man in drei Untergruppen zusammenfasst (vgl. Abb. 4): long terminal repeats (LTRs), long interspersed nuclear elements (LINEs) und Short interspersed nuclear elements (SINEs).
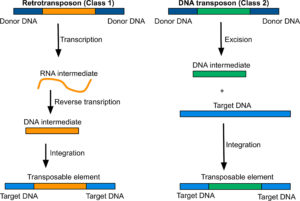
Abb. 3: DNA und RNA-Transposons
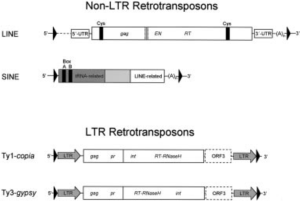
Abb. 4: Typen der RNA-Transposons
Die Frage bleibt: welche Funktionen haben diese Trabnsposons im Genom und woher stammen sie? Gerne werden sie als Beispiel für „egoistische Gene“ benutzt, da ihre eigene Funktion darin gesehen wird, sich selbst zu vermehren und dazu die Zelle als Wirt oder Vehikel zu nutzen. Dies passt in die von Richard Dawkins genutzte Matapher des egoistischen Gens, dessen Kernaussage übrigens nicht ist, dass wir Sklaven unserer Gene oder egoistisch sind (Dawkins 1976). Kernaussage von Dawkins ist, dass die Einheit der natürlichen Selektion nicht die Art oder das Individuum ist, sondern die Gene. Vielmehr stünden Genabschnitte in Konkurrenz zueinander und einzelne Individuen geben nur ihre Gene in einer zufälligen Kombination an die nächste Generation weiter. Insofern besteht eine Konkurrenz der Gene um ihre Verteilung in der nächsten Generation, an den jeweils entsprechenden Stellen im Chromosomensatz. Ich selbst bin kein Anhänger dieser Metapher, da sie mir zu vereinfachend ist und der Komplexität der Genetik und Evolution nicht gerecht wird. Aber solche Transposons entsprechen tatsächlich dem Bild egoistischer Gene: Sie vermehren sich nur um der Vermehrung wegen. Aber tatsächlich gibt es zunehmende Hinweise darauf, dass einige Transposons eine wichtige Rolle in der Evolution spielten.
Vieles, was ein Transposon tut, hängt davon ab, wo es landet. Die Landung innerhalb eines Gens kann zu einer Mutation führen. Dies ist z. B. bei Hämophilie A der Fall, bei der transposonale Elemente im Faktor VII-Gen entdeckt wurden, die die Krankheit auslösen (Kazazian et al., 1988).
Transposons müssen aber nicht immer negative Effekte haben. So ist ein Transposon-Element bei der Pigmentierung bei einer Fischart beteiligt (Koga et al., 2006).
Weitere Studien belegen, dass Transposons besonders im Gehirn aktiv sind (Reilly et al. 2013).
Das Genom ist aber auch in der Lage die Transposons zu regulieren. Einige stummgeschaltete Transposons sind inaktiv, weil sie Mutationen aufweisen, die ihre Fähigkeit beeinträchtigen, sich von einem Ort zum anderen zu bewegen. Andere sind vollkommen intakt und bewegungsfähig, werden jedoch durch epigenetische Abwehrmechanismen wie DNA-Methylierung inaktiv gehalten. Beispielsweise können in menschlichen Zellen kleine RNA-Moleküle, auch als RNA-Interferenz bekannt, die Transposition verhindern. RNA-Interferenz ist ein natürlich vorkommender Mechanismus zur Regulierung der Genaktivität (Yang and Kazazian 2006). Generell spielen Transposons wohl eine wichtige Rolle bei der Genregulation (Bourgque et al. 2018, Biemont 2010, Lisch 2011).
Eine kürzlich veröffentlichte Studie hob die entscheidende Rolle von Transposons in der Evolution der Säugetiere hervor.
Sie untersuchten nämlich die Ursprünge der Schwangerschaft bei Säugetieren und identifizierten große genetische Veränderungen, die den Übergang von der Eiablage zur Lebendgeburt kennzeichnen. Die Forscher fanden Tausende von Genen, die bei Säugetieren in der Gebärmutter ein- oder ausgeschaltet wurden, darunter viele mit kritischen Funktionen beim Aufbau der Kommunikation zwischen Mutter und Fötus und bei der Unterdrückung des Immunsystems der Mutter, damit ein Fötus nicht angegriffen wird.
Um genetische Veränderungen während der Entwicklung der Schwangerschaft bei Säugetieren zu untersuchen, wurden die Gene untersucht, die in der Gebärmutter verschiedener Arten aktiv waren. Die untersuchten Arten waren: Mensch, Affe, Maus, Hund, Kuh und Schwein. Hinzu kamen ein Beuteltier (ein Opossum), dass eierlegende Schnabeltier, ein Vogel, ein Reptil und ein Frosch. Sie rekonstruierten entsprechend dieser Daten einen evolutionären Stammbaum, der aufzeigte bei welchen Gruppen welche Gene an- oder ausgeschaltet wurden.
Die Forscher entdeckten, dass als sich die ersten Säugetiere entwickelten – und die Ressourcen für die Entwicklung des Fötus mehr von der Mutter und weniger aus dem Eigelb stammten – Hunderte von Genen, die für die zelluläre Signalübertragung, den Stoffwechsel und die Uterusentwicklung wichtig sind, in der Gebärmutter aktiviert wurden. Als die Eierschale verloren ging und sich bei dem gemeinsamen Vorfahren der Beuteltiere und Plazentasäuger eine Lebendgeburt entwickelte, wurden mehr als 1.000 Gene aktiviert, von denen viele stark mit der Kommunikation zwischen Mutter und Fötus verbunden waren. Als sich bei Säugetieren in der Plazenta eine längere Schwangerschaft entwickelte, wurden Hunderte von Genen aktiviert, die die Kommunikation zwischen Mutter und Fötus weiter stärkten und ausbauten sowie das mütterliche Immunsystem in der Gebärmutter lokal unterdrückten und so den sich entwickelnden Fötus schützten. Neben der Funktion untersuchten Lynch und seine Kollegen den Ursprung dieser Gene. Sie fanden heraus, dass die meisten bereits eine Funktion in anderen Organsystem wie dem Gehirn, dem Verdauungs- und Kreislaufsystem hatten. Während der Entwicklung der Schwangerschaft wurden diese Gene rekrutiert, um für neue Zwecke in der Gebärmutter genutzt zu werden. Das heißt diese tausende von Genen, die bei der Schwangerschaft aktiv sind, sind keine grundsätzlich neuen Gene, sondern bestehende Gene, die zusätzliche Aufgaben erhalten haben. Es gab also eine Änderung der Genregulation. Sie entwickelten regulatorische Elemente, die es ihnen ermöglichten, durch Progesteron, ein für die Fortpflanzung kritisches Hormon, aktiviert zu werden. Und das wirklich Verblüffende: diese regulierenden Sequenzen waren die Transposons. Viele der alten Säugetiertransposons besaßen Progesteronbindungsstellen, die diesen Prozess regulieren. Durch zufällige Insertion an andere Stellen im Genom scheinen Transposons diesen Aktivierungsmechanismus an nahegelegene Gene weitergegeben zu haben (Lynch et al. 2015, Abb. 5).
Abb. 5: Aktivierung und Deaktivierung von Genen bei der Schwangerschaft nach Lnych et al. 2015
Literatur:
AG Evolutionsbiologie: Evolutionsbeweis durch endogene Retroviren http://www.ag-evolutionsbiologie.net/html/2010/evolutionsbeweis-retroviren.html
Barbulescu, M. et al. (2001) A HERV-K provirus in chimpanzees, bonobos and gorillas, but not humans. Current Biology 11(10), 779-783. https://www.sciencedirect.com/science/article/pii/S0960982201002275
Biémont, C. (2010): A Brief History of the Status of Transposable Elements: From Junk DNA to Major Players in Evolution. Genetics December 1, 2010 vol. 186 no. 4 1085-1093; https://doi.org/10.1534/genetics.110.124180
Bourque, G., Burns, K.H., Gehring, M. et al. (2018): Ten things you should know about transposable elements. Genome Biol 19, 199. https://doi.org/10.1186/s13059-018-1577-z
Dawkins, R. (1976): The Selfish Gene. Oxford University Press
Finlay, G. (2006) Human genetics and the image of god. www.st-edmunds.cam.ac.uk/faraday/CIS/Finlay/lecture.htm
Gifford, R. J. (2006): Evolution at the host-retrovirus interface. In: Bioessays. Bd. 28, Nr. 12, S. 1153–1156, PMID 17117481.
Holmes, E. C. (2007) : Ancient lentiviruses leave their mark. In: Proceedings of the National Academy of Sciences. (PNAS) Bd. 104, Nr. 15, S. 6095–6096, PMID 17404211.
Hughes, J.F. & Coffin, J.M. (2005) Human Endogenous Retroviral Elements as Indicators of Ectopic Recombination Events in the Primate Genome. Genetics 171(3), 1183-1194. www.ncbi.nlm.nih.gov/pmc/articles/PMC1456821/
Johnson, W.E. & Coffin, J.M. (1999) Constructing primate phylogenies from ancient retrovirus sequences. PNAS 96(18), 10254-10260. www.ncbi.nlm.nih.gov/pmc/articles/PMC17875/
Kazazian, H. H., et al. (1988): Haemophilia A resulting from de novo insertion of L1 sequences represents a novel mechanism for mutation in man. Nature 332, 164–166 https://www.nature.com/articles/332164a0
Koga, A., et al. (2006): Vertebrate DNA transposon as a natural mutator: The medaka fish Tol2 element contributes to genetic variation without recognizable traces. Molecular Biology and Evolution 23, 1414–1419 doi:10.1093/molbev/msl003 https://academic.oup.com/mbe/article/23/7/1414/1065108
Lander, E. et al. (2001): Initial sequencing and analysis of the human genome. In: Nature. 2001, Bd. 409, S. 860–921
Lavialle, C., Cornelis, G., Dupressoir, A., Esnault, C., Heidmann, O., Vernochet, C., & Heidmann, T. (2013): Paleovirology of ‘syncytins’, retroviral env genes exapted for a role in placentation. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 368(1626), 20120507. https://doi.org/10.1098/rstb.2012.0507 https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3758191/
Lisch, D. & Bennetzen, J. L. (2011): Transposable element origins of epigenetic gene regulation, Current Opinion in Plant Biology, Volume 14, Issue 2, Pages 156-161, ISSN 1369-5266, https://doi.org/10.1016/j.pbi.2011.01.003. (http://www.sciencedirect.com/science/article/pii/S1369526611000057)
Lynch, V. et al. (2015): Ancient Transposable Elements Transformed the Uterine Regulatory Landscape and Transcriptome during the Evolution of Mammalian Pregnancy, Cell Reports Volume 10, ISSUE 4, P551-561 https://www.cell.com/cell-reports/fulltext/S2211-1247(14)01105-X#articleInformation
McClintock, B. (1951): Mutable loci in maize. Carnegie Institution of Washington Yearbook 50, 174–181
Mi, S., Lee, X., Li, X. et al. Syncytin is a captive retroviral envelope protein involved in human placental morphogenesis . Nature 403, 785–789 (2000). https://doi.org/10.1038/35001608 https://www.nature.com/articles/35001608
Mills RE, Bennett EA, Iskow RC, Devine SE (April 2007). “Which transposable elements are active in the human genome?”. Trends in Genetics. 23 (4): 183–91. doi:10.1016/j.tig.2007.02.006. PMID 17331616
Polavarapu, N. et al. (2006): Identification, characterization and comparative genomics of chimpanzee endogenous retroviruses. Genome Biol. 7(6), R51. www.ncbi.nlm.nih.gov/pmc/articles/PMC1779541/
Reilly MT, Faulkner GJ, Dubnau J, Ponomarev I, Gage FH. The role of transposable elements in health and diseases of the central nervous system. J Neurosci. 2013;33(45):17577–17586. doi:10.1523/JNEUROSCI.3369-13.2013 https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3818539/
SanMiguel, P., et al. (1996): Nested retrotransposons in the intergenic regions of the maize genome. Science 274, 765–768
Theobald, D. (2004): 29+ evidences for macroevolution. Part 4: the molecular sequence evidence. In: TalkOrigin, www.talkorigins.org/faqs/comdesc/section4.html#retroviruses
Yang, N., & Kazazian, H. H. (2006): L1 retrotransposition is suppressed by endogenously encoded small interfering RNAs in human cultured cells. Nature Structural and Molecular Biology 13, 763–771