Historisches
Der Tübinger Forscher Friedrich Miescher gilt als der Entdecker der Nukleinsäuren. Er isolierte in den Sechziger- und Siebzigerjahren des 19. Jahrhunderts aus tierischen und menschlichen Geweben eine Substanz, die er Nuklein nannte, da er diese Substanz aus dem Nukleus, also dem Zellkern, isoliert hatte.
Schon zu Beginn des 20. Jahrhunderts wurde postuliert, dass Nukleinsäuren Träger von Erbinformationen sind.
Doch erst 1928 konnte eine Forschergruppe um Frederick Griffith und Oswald Avery am Rockefeller Institute in New York nachwiesen, dass eine chemisch relativ einfache, langkettige Nukleinsäure (Desoxyribonukleinsäure, DNS, oder in der angelsächsischen Abkürzung DNA) Träger genetischer Information sein muss.
Sie überführten eine erbliche Eigenschaft eines Bakterienstammes der Art Streptococcus pneumoniae auf einen anderen, ein Prozess, der heute als Transformation bezeichnet wird. Streptococcus pneumoniae kommt in zwei Formen vor: in einer S-Form und einer R-Form. Die S-Form ist krankheitsauslösend und die Bakterien haben eine schützende Schleimkapsel, die vom Immunsystem des Wirtes nicht erkannt wird. Der R-Form fehlt diese Schutzsubstanz und sie sind nicht krankheitserregend. Mäuse wurden mit beiden Bakterienstämmen behandelt. Erhielten die Mäuse den infektiösen S-Stamm starben die Mäuse, bei einer Injektion mit dem R-Stamm überlebten sie. Erhielten die Mäuse einen durch Hitzebehandlung abgetöteten S-Stamm überlebten sie ebenfalls. In einem weiteren Versuchsablauf wurden abgetötete S-Stämme mit lebenden R-Stämmen, die nicht infektiös sind, vermengt und den Mäusen injiziert. Das Ergebnis: die Maus starb trotzdem. Was ist geschehen? Die infektiöse Eigenschaft des S-Stammes wurde von den harmlosen R-Stamm aufgenommen, ein Prozess, der heute als Transformation bezeichnet wird. Die Substanz, die die Eigenschaften von einem Bakterienstamm auf den nächsten überträgt muss hitzebeständiger sein. 1944 verfeinerten sie ihr Experiment und konnten an den Zellen des S-Stammes unter systematischem Einsatz von DNA- und Proteinextraktionen Proteine als Träger des Erbmaterials ausgeschlossen und der Nachweis erbracht werden, dass das Erbmaterial aus DNA besteht (Abb. 1).
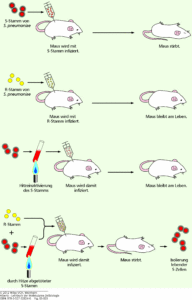
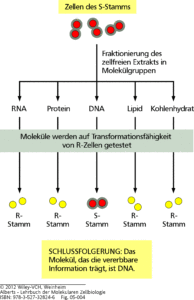
Abb. 1: Experiment von Griffith, 1928
1952 entdeckten die Forscher Alfred Day Hershey und Martha Chase, dass die genetische Information ausschließlich in DNA enthalten ist.
Die beiden Forscher arbeiteten mit dem Bakteriophagen T2, der das Darmbakterium Escherichia coli befällt. Die Bakteriophagen bestehen aus einer Proteinhülle, die man im Experiment mit einem Schwefelisotop (35S) markieren kann. Schwefelatome kommen in Nukleinsäuren nicht vor. DNA kann dagegen mit einem Phosphorisotop (32P) markiert werden. Phosphoratome fehlen wiederum in Proteinen der Bakteriophage. In zwei unabhängigen Experimenten infizierten Hershey und Chase Bakterien mit den T2-Phagen, die entweder radioaktiv markiertes Protein oder radioaktiv markierte DNA enthielten. Es stellte sich heraus, dass nur die radioaktiv markierte DNA in den neu gebildeten Phagen auftauchte, nicht aber die radioaktiv markierten Proteine (Abb. 2).
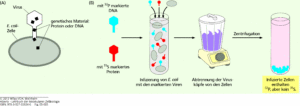
Abb. 2: Experiment von Hershey & Chase, 1944
Die Experimente von Griffith, Avery, Hershey und Chase gelten heute als die klassischen Arbeiten, deren Ergebnisse die DNA als Träger der genetischen Information identifizierten. Doch woraus besteht die DNA überhaupt?
Nukleinsäuren
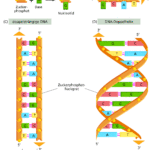
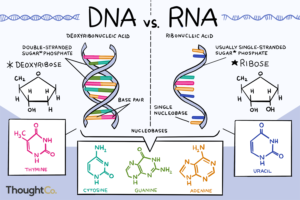
Die DNA gehört zur Stoffgruppe der Nukleinsäuren. Es handelt sich hierbei um Polymere, deren Untereinheiten (Monomere) die Nukleotide sind. Nukleotide bestehen aus drei prinzipiellen Bausteinen: Phosphaten, Zucker, Pyrimidin- oder Purin-Basen. Es gibt vier verschiedene Basen in der DNA: Cytosin und Thymin, die zu den Pyrimidin-Basen gehören sowie Guanin und Adenin, die zu den Purin-Basen gehören. Bei dem Zuckermolekül handelt es sich um die 2-Desoxyribose (Abb. 3). Das Adenin haben wir in einer anderen Verbindung, dem ATP, kennengelernt.
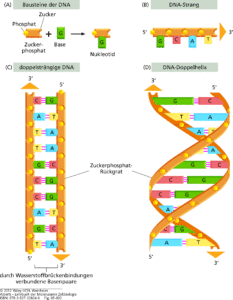
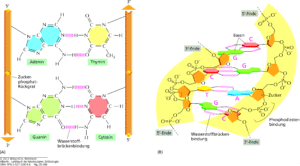
Abb. 3: Grundaufbau der DNA
Das erste C-Atom des Zuckers Desoxyribose geht eine Verbindung mit einem Stickstoffatom der Base. Für eine Verbindung mit dem Phosphatrest steht das 5 bzw. 3. C-Atom der Desoxyribose bereit.
Die Nukleotide verbinden sich miteinander, indem ein Desoxyribose-Molekül über eine Phosphat-Verbindung mit dem nächsten Desoxyribose-Molekül verknüpft wird. Der Phosphatrest bildet somit eine Bindung mit dem 5. C-Atom des einen Zuckermoleküls und dem 3. C-Atom des nächsten Zuckermoleküls usw. Bei diesen Verbindungen wird Wasser abgespalten, wie z. B. bei der Peptidbindung. Das Rückgrat dieser Nukleotid-Ketten besteht aus alternierenden Zuckermolekülen und Phosphatgruppen. Es resultieren zwei unterschiedliche Enden, welche nach der Lage des Kohlenstoffatoms der Desoxyribose entsprechend als 3´ – oder 5´-Ende bezeichnet werden.
So entsteht die Nukleotid-Kette. Dieses Phosphat-Zucker-Gerüst bildet den invariablen Teil der DNA. Variabel sind die Nukleotid-Basen, von denen es wie erwähnt vier verschiedene gibt. Solch eine Nukleotid-Kette bezeichnet man auch als Einzelstrang
Die DNA liegt normalerweise jedoch nicht als Einzelstrang, sondern Doppelstrang vor. Die Basen der NuKleotide sind in der Lage, mit anderen Basen in Wechselwirkung zu treten. Diese Basenpaarungen werden durch Wasserstoffbrücken-Bindungen vermittelt, welche sich zwischen Sauerstoff- bzw. Stickstoffatomen der gegenüberliegenden Basen ausbilden. Einer Purinbase steht in der Regel eine Pyrimidinbase gegenüber. Dabei liegen sich stets ein Cytosin und ein Guanin bzw. ein Thymin und ein Adenin gegenüber. Cytosin und Guanin werden durch drei Wasserstoffbrücken, Thymin und Adenin durch zwei miteinander verbunden. Cytosin und Guanin, sowie Thymin und Adenin sind zueinander komplementär. Die Sequenz der Nukleotidbasen eines Stranges der DNA (in 5′- nach 3′-Richtung) entspricht komplementär der Nukleotidbasensequenz (oder einfach Basensequenz) des anderen Stranges in 3′- nach 5′-Richtung. Die Spezifität der Basenpaarung ist das wichtigste strukturelle Merkmal der DNA. Die Menge von A in einem DNA-Doppelstrang entspricht der von T; ebenso korreliert die Menge von C mit der von G. Diese Beziehung ist auch als Chargaff-Regel bekannt. Der AT- bzw. GC-Gehalt variiert zwischen unterschiedlichen Organismen und wird auch als Klassifizierungsmerkmal herangezogen
1953 hatten James D. Watson und Francis H. Crick die Struktur des DNA-Moleküls herausgefunden, indem sie Daten aus Röntgenbeugungsexperimenten von Maurice Wilkins und Rosalind Franklin verwendeten. In einer am 25. April 1953 in Nature erschienenen Arbeit von einer dreiviertel Seite (A structure for deoxyribonucleic acid, Nature 171: 737–738, 1953) beschreiben Watson und Crick die Struktur der DNA als rechtsgängige Doppelhelix. Die abwechselnden Zuckermoleküle und Phosphatgruppen der Einzelstränge bilden ein außenliegendes Rückgrat, während die Basen in das Helixinnere hinein orientiert sind (Abb. 4). Da sowohl GC- als auch AT-Paarungen jeweils eine Purin- und eine Pyrimidinbase enthalten, haben sie eine sehr ähnliche Raumausfüllung.
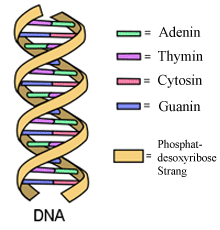
Abb. 4: DNA-Doppelhelix
Im isolierten Zustand tritt uns die DNA als relativ uniformer Faden mit einem Durchmesser von etwa 2 nm entgegen. Erhöht man die Temperatur einer DNA-haltigen Lösung, werden die Wasserstoffbrücken-Bindungen zerstört und die beiden Stränge trennen sich voneinander zu Einzelstrang-DNA. Mit zunehmender Temperatur entstehen immer mehr einzelsträngige Bereiche. Die Herstellung einzelsträngiger DNA wird auch als Denaturierung bezeichnet. Dies kann durch erhöhte Temperatur oder der Zugabe bestimmter Chemikalien wie Harnstoff oder Formamid erreicht werden.
Bei der Renaturierung, auch als Reassoziation oder Annealing bezechnet, lagern sich die komplementären DNA-Stränge wieder aneinander. Dieser Prozess kann durch die Wahl der Pufferbedingungen (pH-Wert bzw. Salzkonzentration), vor allem aber der Temperatur beeinflusst werden.
RNA
Die RNA ist ebenso wie die DNA eine Nukleinsäure. RNA besteht jedoch aus dem Zucker Ribose. Der Unterschied zur Desoxyribose besteht, dass die Ribose am 2. C-Atom eine OH-Gruppe gebunden hat, die 2-Desoxyribose – wie der Name vermuten lässt – hat nur eine Wasserstoffbindung mit dem zweiten C-Atom. Die RNA besitzt an Stelle des Thymins die Pyrimidinbase Uracil (U), die sich vom Thymin nur durch das Fehlen einer Methylgruppe in Position 5 unterscheidet (Abb. 5). Die RNA übernimmt im Zellstoffwechsel, besonders bei der Synthese von Proteinen, eine wichtige Rolle ein.
Organisation der Genome
Bei den Bakterien schwimmt die DNA frei im Zellplasma, auch als Bakterien-Chromosom bezeichnet. Dabei ist das Bakterien-Chromosom viel länger als die Bakterienzelle. Bei Coli-Bakterien ist das doppelsträngige DNA-Molekül etwa 1,6 mm lang und damit tausendmal länger als die Bakterienzelle selbst. Daher müssen die Genome kompaktiert werden. dies geschieht mittels Bindeproteinen, die in Wechselwirkung mit dem Bakteriengenom treten und dieses so schleifenförmig organisiert. Dies kann geschehen, weil die DNA negativ geladen ist, die Bindeproteine aber viele positiv geladene Aminosäuren haben (Abb. 6).
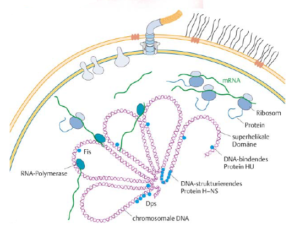
Abb. 6: Bakteriengenom
Der Großteil des Genoms eukaryotischer Zellen befindet sich im Zellkern. Auch bei Eukaryoten ist der DNA-Faden viel länger als die Zelle und auch hier helfen Bindeproteine das Genom kompakt zu halten. Die DNA und die mit ihr assoziierten Proteine werden zusammen als Chromatin bezeichnet, die kompakteste Form des Genoms als Chromosom. Die wichtigsten Bindeproteine eukaryotischer Zellen bezeichnet man als Histone. Weitere Proteine sind die Nicht-Histon-Proteine, welche für die Organisation von Funktionsdomänen im Chromosom verantwortlich sind.
Die Maximale Verkürzung von 1: 12 000 wird über vier Organisationsstufen erreicht (Abb. 7). Die unterste Verpackungsstufe besteht in der Ausbildung der Nukleosomen und führt zu einem 10-nm-Faden. Auf dieser Stufe wird ein Verkürzungsfaktor des DNA-Doppelstrangs von sechs bis sieben erzielt. Ein Nukleosom besteht aus einem bestimmten Abschnitt Kern-DNA, der um ein Aggregat aus acht Histon-Proteinen gewickelt ist. Diese Histone bezeichnet man auch als Core-Histone und das Aggregat als Histon-Oktamer. Es gib vier verschiedene Core-Histon-Moleküle. die als H2A, H2B, H3 und H4 bezeichnet werden. Alle vier Core-Histone sind im Nukleosom zweimal vertreten und bilden das Histon-Oktamer. Nukleosomen enthalten weiterhin einen außerhalb der Core-Histone liegenden DNA-Abschnitt und ein weiteres Histonmolekül, das Linker-Histon H1.
Die Histone haben zwei auffällige Eigenschaften. Sie besitzen einen relativ hohen Anteil (zusammen über 20 %) an den basischen Aminosäuren Arginin und Lysin, durch deren positive Ladungen die negativen Ladungen der DNA, die den Core-Histonen eng anliegt, neutralisiert werden. Weiterhin sind die vier Core-Histone in ihrer Molekülgröße und Aminosäurenzusammensetzung in der Evolution außerordentlich konserviert. Histon H4 unterscheidet sich z. B. beim Rind und bei der Erbse nur durch eine einzige Aminosäure.
Die Sekundärstruktur bildet der sogenannte Solenoid, ein 30 nm-Faden. Bei steigender Ionenstärke in der Lösung bilden die Nukleosomen eine Überstruktur aus. Es ist eine Superhelix mit nur kleinen Unregelmäßigkeiten, bei der das Linker-Histon H1 im Innenraum zu liegen kommt.
Die Teritärstruktur bilden sogenannte Schleifendomänen aus. Sie werden vom 30-nm-Faden ausgebildet. Sie lagern sich zu mehreren in einer Ebene an (Rosette) und sind in der Mitte über spezifische DNA-Bereiche an ein Proteingerüst gebunden. Sie bilden somit Rosetten. Die Anordnung zahlreicher Rosetten hintereinander führt zu einem 300-nm-Faden. Dieser 300-nm-Faden ist helikal aufgewunden und bildet die Quartärstruktur: die Chromatide. In jeder Chromatidenwindung sind etwa 30 Rosetten enthalten.
Im Zuge der Zellteilung (Mitose), mit der wir uns gesondert befassen werden, werden die Chromatiden verdoppelt. Zwei gleiche Chromatiden bezeichnet man als Schwesterchromatiden und diese bilden zusammen das Chromosom, die stärkste Verkürzung des DNA-Moleküls.

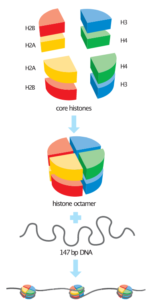
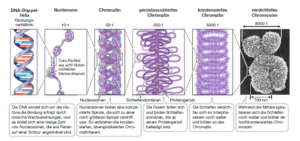
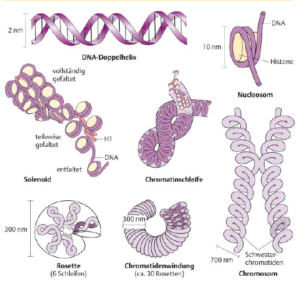
Abb. 7: Organisationsstufen des eukaryotischen Chromosoms
Chromosomen besitzen eine Reihe von Bereichen, denen spezielle Aufgaben zufallen. Man nennt sie deshalb auch Funktionselemente der Chromosomen.
Zu den wichtigsten gehören die Centromere, die für die geordnete Verteilung der Chromosomen in der Zellteilung verantwortlich sind.
Die Enden der Chromosomen bilden die Telomere, die bei der Verdoppelung der DNA eine wichtige Rolle spielen.
Mit den Centromeren und Telomeren, werden wir uns im Zusammenhang der DNA-Verdoppelung und Zellteilung näher anschauen.
Die Gesamtheit der Chromosomen bezeichnet man als Karyotyp. Jede Tierart verfügt über eine bestimmte Anzahl an Chromosomen. Wir Menschen haben 46 Chromosomen. 44 sogenannte autosomale Chromosomen und 2 Geschlechtschromosomen (Frauen: XX, Männer: XY). Tatsächlich handelt es sich bei den 46 Chromosomen um 23 Chromosomen-Paare. Das heißt: Wir haben 23 unterschiedliche Chromosomen, die jeweils doppelt vorhanden sind. Doppelte Chromosomen sind zueinander homolog (sie sind Variationen desselben Chromosomentyps). Einen doppelten Chromosomensatz bezeichnet man als diploid oder kurz: 2n. Der Karyotyp kann in einem Karyogramm dargestellt werden. Dieser diploide Chromosomensatz entsteht durch die Verschmelzung der Ei- und Spermazelle, die jeweils nur einen halben Chromosomensatz haben, man spricht von haploid (Abb. 8).
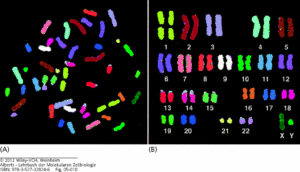
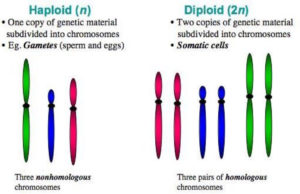
Abb. 8: Karyogramm des Menschen
Das menschliche Genom
Die Genomgröße kann bei Lebewesen unterschiedlich groß sein (Abb. 9). Betrachten wir uns aber die DNA des Menschen etwas genauer.

Der DNA-Strang eines Menschen besitzt etwa 3 Milliarden Basenpaare, hat also vereinfacht ausgedrückt 3 Mrd. Buchstaben der entsprechenden Basen A, T, C und G. Nur als Vergleich: Die Bibel, welches nun sicherlich kein dünnes Buch ist, hat etwa 4,4 Millionen Zeichen – Leerzeichen & Sonderzeichen inklusive – das entspricht 0,14% der Basenpaare der DNA.
Aber diese 3 Mrd. Basenpaare können noch weiter unterteilt werden (Abb. 10):
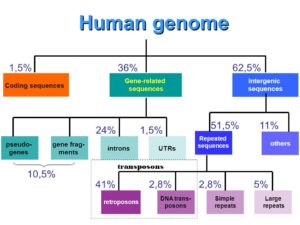
Abb. 10: menschliches Genom
Zum einen haben wir die Gene. Jeder kennt diesen Begriff bzw. hat von ihm etwas gehört. Generell wird unter Genen das verstanden, was wir biologisch an die nächste Generation vererben. In der Molekularbiologie ist die Definition des Gens eingeschränkter.
Gene definieren sich dadurch, dass sie die Information für den Aufbau von Proteinen haben inklusive ihrer Regulationssequenzen – also jene Abschnitte die mitkontrollieren, wann ein Gen ein- oder ausgeschaltet wird. Solch ein Gen liegt aber oft nicht als „ununterbrochene“ Kette vor, sondern ist „zerstückelt“ in „Introns“ und „Exons“. Exons werden die codierten Elemente genannt, die also die Information für den Aufbau der Proteine haben, die Introns sind die nicht codierenden Bereiche. Über ihre Funktion werden wir uns näher befassen, wenn wir die Proteinsynthese und Genregualtion näher behandeln. Das menschliche Genom hat etwa 20.000 Gene. Das klingt viel – beträgt aber nur 1-1,5% der Gesamt-DNA. Der Rest besteht aus sog. nicht-codierter DNA.
Hierzu gehören auch die Pseudogene. Das sind Gene, die ehemals eine Funktion hatten, aber durch Mutationen ausgeschaltet wurden und somit nicht in Proteine übersetzt werden.
Ein Großteil des menschlichen Genoms – und des Genoms aller Eukaryoten – besteht aus sogenannter repetitiver DNA. Repetitive DNA besteht aus sich vielfach wiederholenden DNA-Sequenzen. Die jeweils wiederholten Sequenzen können entweder über das gesamte Genom verstreut oder tandemartig angeordnet an bestimmten Stellen des Genoms liegen (Abb. 11).
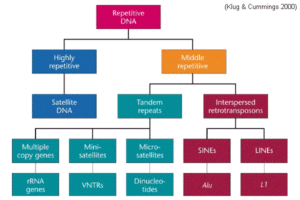
Abb. 11: Repetitive DNA
Je nach ihrer Größe gibt es klassische Satelliten-DNA, die bis zu 5 Mio. Basenpaare lang sein können und bis zu einer Mio. Wiederholungen von Sequenzen mit 5 bis 300 Nukleotiden haben können. Zu ihnen gehören die Alpha-Sequenzwiederholungen an den Centromeren der Chromosomen, die beim Menschen aus tandemartig hintereinander liegenden Wiederholungen einer Sequenz von 171 bp bestehen und Bindungsstellen für Centromer-spezifische Proteine sind. Mit einer Gesamtlänge von 100 – 20.000 Basenpaaren sind die Gruppen der Minisatelliten-DNA deutlich kleiner. Zu den Minisatelliten, die aus Einheiten von maximal 15 Basen bestehen, gehört die DNA der Telomere an den Chromosomenenden. Sie enthält bis zu 1000 tandemartige Wiederholungen einer kurzen DNA-Sequenz, die beim Menschen aus 6 Basen besteht. Sie sind Bindungsstellen für Telomer-spezifische Proteine.
Mikrosatelliten sind sehr kurze Einheiten von bis zu 6 Basenpaaren, die nur bis zu einigen hundert Mal tandemartig wiederholt werden. Mikrosatelliten stellen selbst repetitive Einheiten dar, die über alle Chromosomen eines Genoms verteilt bis zu 100 000-mal vorkommen können. Wegen der Zufälligkeit ihrer Entstehung sind Mikrosatelliten bei verschiedenen Individuen unterschiedlich lang (Längenpolymorphismus), was man sich bei der Erstellung eines „genetischen Fingerabdrucks“ zunutze macht. Durch Vergleich der Längen von Genomregionen, die Mini- und Mikrosatelliten-Loci enthalten, lassen sich Individuen eindeutig identifizieren und verwandtschaftliche Beziehungen aufklären.
Mittelrepetitive Sequenzen sind unterschiedlich lang und haben verschiedene Längeneinheiten von Wiederholungen und sind meist über das gesamte Genom verteilt. Hier unterscheidet man zwischen den SINEs (short interspersed elements) und LINEs (long interspersed elements), sie sind über das gesamte Genom verteilt.
SINEs haben kurze Sequenzen von 200 bis 400 Basenpaaren. Zu ihnen gehören die ca. 1,1 Millionen Alu-Elemente, die den größten Teil der mittelrepetitiven Sequenzen des menschlichen Genoms ausmachen.
LINEs haben eine Länge von 1.400 – 6.000 Basenpaaren und kommen mit einer Kopienzahl von 60.000 bis 100.000 im Genom vor.
Ebenfalls den LINEs zugerechnet werden die Transposons, die springenden Gene. Hinzu kommen die menschlichen endogenen Retroviren (HERV, human endogenous retroviruses). Transposons und endogene Retroviren haben wir in einem anderen Beitrag kennengelernt.
Bei Eukaryoten befindet sich die DNA nicht nur im Zellkern, sondern auch in einigen Zellorganellen, namentlich den Mitochondrien und den Chloroplasten. Eine Zelle kann mehrere hundert bis tausend Mitochondrien haben und jedes Mitochondrium hat einen kleinen DNA-Ring, der bei Menschen 16.569 Basenpaare lang ist.