Im letzten Beitrag lernten wir die ersten Stoffwechselprozesse der Protozellen kennen, die in den hydrothermalen Schloten einen natürlichen Protonengradienten ausnutzten, um den Energie- und Kohlenstoffwechsel anzutreiben.
Text als pdf
Protonengradienten
Protonengradienten zur Umwandlung von Energie in Form des ATP sind im Stoffwechsel des Lebens allgegenwärtig. In diesem Fall spricht man auch von Chemiosmose (Mitchell 1961). Unter diesem Begriff versteht man einen Vorgang, bei dem Teilchen (zum Beispiel Protonen) in Richtung eines Konzentrationsgradienten durch eine biologische Membran diffundieren und dabei ATP synthetisieren oder andere endergonische Reaktionen antreiben.
Ein solcher Protonengradient liegt vor, wenn es in einem chemischen System unterschiedliche Protonen-Konzentrationen gibt. Da die Protonen-Konzentration als pH-Wert definiert ist, werden Protonengradient und pH-Gradient synonym gebraucht, d. h. es liegt zwischen den Membranen auch ein unterschiedlicher pH-Wert vor. Aufgrund ihrer Struktur sind Zellmembranen semipermeabel. Kleine hydrophobe und gasförmige Moleküle wie CO2 und O2 können ungehindert durch die Biomembran durchdringen. Dasselbe gilt auch für kleine polare Moleküle wie z. B. Wasser. Große Moleküle, wie Aminosäuren und Glucose, sowie Ionen wie H+, also Protonen und Salze können die Biomembran nicht durchdringen. Zellmembranen sind also für Protonen nahezu undurchlässig, wodurch sich zwischen Innen und Außen ein Konzentrationsgefälle bildet (Abb. 1).
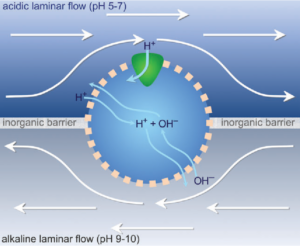
Abb. 1: Eine Zelle mit natürlichem Protonengradienten als Antriebskraft. In der Mitte des Bildes befindet sich eine Zelle, umschlossen von einer Membran, die für Protonen durchlässig ist. Die Zelle ist „eingekeilt“ in einem kleinen Spalt in einer anorganischen Barriere, die in einem Schlot mit Mikroporen als Trennwand zwischen zwei Phasen fungiert. In der oberen Phase sickert leicht saures Meerwasser durch eine längliche Pore; sein pH-Wert liegt bei 5–7 (im Modell meist als 7 angesetzt). In der unteren Phase sickern alkaline hydrothermale Flüssigkeiten durch eine mit der anderen nicht in Verbindung stehende Pore. Ihr pH-Wert liegt bei 10. Die laminare Strömung bezeugt geringe Turbulenzen und Durchmischung, was für Strömungen in engen Räumen charakteristisch ist. Protonen (H+) können direkt durch die Lipidmembran treten oder aber durch in die Membran eingebettete Proteine (dreieckige Form oben). Dabei folgen sie einem Konzentrationsgradienten vom sauren Meerwasser zur alkalinen hydrothermalen Flüssigkeit. Hydroxidionen (OH−) strömen in die entgegengesetzte Richtung von der alkalinen hydrothermalen Flüssigkeit zum sauren Meerwasser, jedoch ausschließlich durch die Membran. Die Gesamtdurchflussrate der Protonen hängt ab von der Permeabilität der Membran für H+, der Neutralisierung durch OH− (zu Wasser, H2O), der Zahl der Membranproteine, der Größe der Zelle und der durch die Wanderung der Ionen von einer Phase in die andere erzeugten Ladung über der Membran.
Dieses Konzentrationsgefälle wird durch die chemiosmotische Kopplung zur Gewinnung für ATP genutzt. Der wichtigste chemiosmotische Vorgang in der Zellbiologie ist sicherlich der Protonenfluss während der Atmungskette bei der Zellatmung (Abb. 2) sowie der Protonenfluss während der Lichtreaktion bei der Photosynthese. Die Protonen fließen durch eine ATP-Synthase durch die innere Mitochondrienmembran bzw. durch die Tylakoidmembran. In unseren Mitochondrien haben wir einen elektrischen Schaltkreis, bei dem die Membran wie ein Isolator wirkt: Wir pumpen Protonen durch die Membran, und die meisten von ihnen kehren durch Proteine zurück, die wie Turbinen wirken und die Arbeit antreiben. Im Falle der ATP-Synthase treibt der Protonenstrom durch diesen nanoskopisch kleinen, rotierenden Motor die ATP-Synthese an. Bedenken wir aber, dass das gesamte System auf aktives Protonenpumpen angewiesen ist. Werden die Pumpen blockiert, kommt alles zum Stillstand. Das passiert, wenn wir eine Zyanidkapsel schlucken: Sie stoppt die letzte Protonenpumpe der Atmungskette in unseren Mitochondrien.
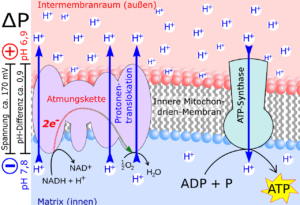
Abb.2: Schema der Atmungskette in den Mitochondrien.
Die Nutzung von Protonengradienten (oder anderen Ionengradienten) über Membranen zur Energieerhaltung, wie bei der chemiosmotischen Kopplung, ist so universell wie der genetische Code selbst, doch seine Ursprünge sind unklar. Soweit die Phylogenetik irgendeinen Hinweis auf die tiefsten Äste des Stammbaums des Lebens geben kann, stehen autotrophe, chemiosmotische Zellen immer an dessen Basis (Say & Fuchs 2010; Stetter 2006; Maden 1995). Obwohl es kaum Zweifel gibt, dass der letzte universelle gemeinsame Vorfahre (LUCA) chemiosmotisch war und über eine membrangebundene ATP-Synthase verfügte (Mulkidjanian et al. 2007), wurde die Frage, wie Ionen durch Membranen gepumpt wurden, bisher kaum untersucht.
Wichtige Hinweise auf die frühe Evolution der Energieerhaltung ergeben sich aus biochemischen Untersuchungen von Methanogenen und Acetogenen, die von der Reduktion von CO2 unter Verwendung von Elektronen aus H2 leben (Fuchs 2011; Kaster et al. 2011; Buckel & Thauer 2012, Berg 2011). Beide Gruppen haben wir in den letzten Episoden kennengelernt und beide gehören unterschiedlichen Domänen an: die Methanogenen gehören zu den Archaeen und die Acetogenen zu den Bakterien. Beide können, wie erwähnt, aus H2 und CO2 organische Moleküle herstellen und nutzen für die Fixierung des Kohlenstoffs den reduktiven Acetyl-Co-A-Weg, der einfachsten Form der CO2-Fixierung. In den Details unterscheiden sich aber beide Gruppen und liefern Hinweise dafür, warum sich Archaeen und Bakterien so fundamental unterscheiden – dazu später mehr. Was aber zu beachten ist: Acetogene und Methanogene – genauso wie alle anderen lebenden Organismen nutzen Proteine, die in der Lage sind, einen Gradienten zu erzeugen und zu nutzen. Zu diesen Proteinen gehört die erwähnte ATP-Synthase. Die ATP-Synthase war ein Produkt langer Selektion in den frühen Phasen der Evolution, und war bereits beim letzten gemeinsamen Vorfahren vorhanden (Mulkidjanian et al. 2007). Dies wirft das erste evolutionäre Henne-Ei-Problem auf: Die Proteinsynthese verbraucht 75 % des ATP-Budgets einer Zelle (Harold 1986), und der ATP-Pool wird letztlich durch Proteine aufgefüllt, die sich chemiosmotische Gradienten zunutze machen. Wenn aber die von den Proteinen konservierte Energie zur Herstellung von Proteinen benötigt wird, woher kam dann die Energie, die die ersten Proteine hervorbrachte?
Die Lösungen haben wir in den letzten Episoden ebenfalls kennengelernt: die physikalische Struktur der weißen Raucher, also alkalinen Hydrothermalquellen, ermöglichte die Entstehung natürlicher Protonengradienten über halbleitende dünne Mineralwände, sodass CO2 mit H2 reagieren kann.
Mehrere Arbeiten (Shock et al. 1998; Shock & Canovas 2010; Amend & McCollom 2009, Russell et al. 1993; Russell & Hall 1997, Sleep et al. 2011, Tamblyn & Hermann 2023, Wimmer 2021a, b) haben gezeigt, dass ein anhaltendes Ungleichgewicht an submarinen hydrothermalen Quellen, die mit dem Ozeanwasser in Berührung kommen, Bedingungen schafft, die die Synthese der Bausteine des Lebens, insbesondere der Aminosäuren, aus H2, CO2 und Ammonium-Ionen (NH4+) thermodynamisch begünstigen.
Sind diese Bedingungen erfüllt, läuft ein Großteil der untersuchten Reaktionen ohne weitere Energiezufuhr ab und setzt teilweise sogar noch Energie frei, die dann die nächstfolgenden Reaktionen anstößt. Das ist deshalb aufregend, weil der sonst so komplizierte Stoffwechsel auf einmal eine natürliche Tendenz offenbart, sich unter den richtigen Bedingungen von alleine zu entfalten. Der Urstoffwechsel selbst könnte demnach als interne Energiequelle für die ersten Anfänge des Lebens gedient haben. Es konnte gezeigt werden, dass die Energie am Ursprung des Lebens rein chemischer Natur ist. Wir brauchen kein Sonnenlicht, keine Meteoriten, kein UV-Licht: nur Wasserstoff und Kohlendioxid plus etwas Ammoniak und Salz. Unter diesen Bedingungen sind auch keine Membranproteine, Cytochrome oder Enzyme notwendig. Und das sind genau die Zutaten, die die ersten Zellen an heißen, alkalischen Schloten der Tiefsee gefunden haben. Dieses Milieu entspricht genau der Umgebung, die man im Hydrothermalfeld ‚Lost City‘ im Atlantis-Massiv, einem unterseeischen Gebirge im Mittelatlantik, vorfindet. Solche Schlote sind über Zeiträume von 30.000 Jahren und mehr stabil (Kelley et al. 2002) und wären auf der frühen Erde weit verbreitet gewesen (Arndt & Nisbet, 2012). Laufen solche Reaktionen spontan ab, können zusätzliche Proteine die Energiebilanz optimieren und die natürliche Selektion kann hierauf zurückgreifen.
Weitere Studien werfen ein Licht darauf, wie es die Urzellen schafften Wasserstoffgas H2 als Energiequelle zu nutzen. Denn um aus Wasserstoff Energie zu gewinnen, müssen Zellen das H2-Molekül zunächst aufspalten und dessen Elektronenpaar auf spezielle Weise aufteilen. Einem der beiden Elektronen wird dabei so viel Energie entzogen, dass das andere Elektron in einen höheren Energiezustand versetzt werden kann. Dieser Vorgang wird als Elektronenbifurkation bezeichnet (Buckel & Thauer 2013, 2018, Müller et al. 2018, Katsyv et al. 2023, Lubner et al. 2017, Schuchmann & Müller 2012, Abb. 3). Heutige Zellen besitzen für diesen Prozess mehrere metallhaltige Enzyme und organische Cofaktoren, die die Elektronen zu einem eisenhaltigen Molekül namens Ferredoxin befördern (Hermann et al. 2008, Li et al. 2008, Kaster et al. 2011, Kayastha et al. 2021). Dieses fungiert als Elektronenträger und ermöglicht die Aufteilung in zwei energetisch unterschiedliche Teilchen. Doch solche Enzyme und Cofaktoren gab es auf der Urerde noch nicht. Wie haben die evolutionär frühen Zellen diese energetische Aufwärtsreaktion dann stattdessen vollbracht? Ein Team von der Heinrich-Heine-Universität Düsseldorf hat das nun genauer untersucht (Brabender et al. 2024). Die Analysen ergaben: Bei dem für viele hydrothermale Quellen typischen pH-Wert von 8,5 sind offenbar tatsächlich keine Enzyme erforderlich, um die Wasserstoff-Moleküle zu spalten. Allerdings ist neben der alkalischen Umgebung auch metallisches Eisen nötig. Die H–H-Bindung von H2 spaltet sich dann an der Eisenoberfläche und erzeugt Protonen, die vom alkalischen Wasser verbraucht werden, und Elektronen, die dann einfach direkt auf Ferredoxin übertragen werden.
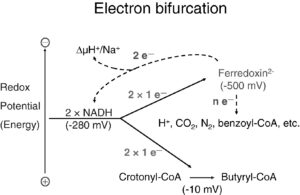
Abb. 3: Elektronen-Bifurkation
In allen Stadien nutzten die Protozellen sowohl organische Membranen als auch anorganische Wände, um das geochemische chemiosmotische Potenzial nutzbar zu machen. Um jedoch als unabhängige, freilebende Zellen aus den Schloten zu entkommen, muss man sich nicht mehr auf die natürlichen Protonengradienten verlassen, sondern echte Zellen bilden, die in der Lage sind, selbst aktiv Ionengradienten zu erzeugen.
Das Problem mit der Membrandurchlässigkeit
Das ist aber nicht so einfach, wenn wir uns die Ursprünge der Zellmembran vorstellen:
Die Protozellen in einer Pore des hydrothermalen Schlots hatten eine für Ionen durchlässige Membran, d. h. anders als bei modernen Zellen (Pohorille & Deamer, 2009; Mulkidjanian et al., 2012, Lane 2017 Abb. 1). Die eine Seite der Zelle ist dem sauren Meerwasser ausgesetzt (ph 5-7), die andere dem konstanten Strom alkaliner hydrothermaler Flüssigkeit (ph 9-11). Das ergibt einen Unterschied in der Protonenkonzentration zwischen der Innen- und der Außenseite, sodass Protonen dem Gradienten folgend nach innen strömen. Protonen (H+) können direkt durch die Lipidmembran treten oder aber durch in die Membran eingebettete Proteine. Dabei folgen sie einem Konzentrationsgradienten vom sauren Meerwasser zur alkalinen hydrothermalen Flüssigkeit. Hydroxidionen (OH−) strömen in die entgegengesetzte Richtung von der alkalinen hydrothermalen Flüssigkeit zum sauren Meerwasser, jedoch ausschließlich durch die für Ionen durchlässige Membran. Protonen strömen also ins Zellinnere hinein und verlassen sie auf der anderen Seite der Zelle.
Eine durchlässige Membran stellt de facto ein geringeres Hindernis für den Protonenstrom dar. Überdies treten Hydroxidionen (OH−) aus den alkalinen Flüssigkeiten mit etwa derselben Rate durch die Membran wie die Protonen. Wenn H+ und OH− aufeinandertreffen, reagieren sie zu Wasser (H2O) und eliminieren die positive Ladung des Protons auf einen Schlag. Einige Arbeitsgruppen (Sojo et al. 2014) berechneten, dass die verfügbare Antriebskraft von der Durchlässigkeit der Membranen für Protonen abhängig. Ist die Membrandurchlässigkeit groß, strömen Protonen in großen Massen ein und verlassen diese auch wieder, eliminiert durch den raschen Eintritt von OH–-Ionen. Hatten durchlässige Membranen zusätzlich Membranproteine, wie die ATP-Synthase, konnten die Protonen noch schneller in das Zellinnere gelangen als durch die Lipide der Zellmembran. Und hier das Paradoxe: Zellen mit einer durchlässigen Membran gewinnen aus einem natürlichen Protonengradienten von 3 pH-Einheiten genauso viel Energie wie moderne Zellen mit einer semipermeablen, also halbdurchlässigen, Membran! Sie könnten sogar sehr viel mehr gewinnen.
Methanogene produzieren durchschnittlich 40-mal mehr Abfallprodukte (Methan und Wasser) als organische Materie. Etwa 98% der ihnen zur Verfügung stehenden Energie verwenden sie darauf, per Methanogenese Protonengradienten zu erzeugen und nur 2 Prozent für die Bildung neuer organischer Materie (Thauer et al. 2008, Philips & Milo 2009).
Würden durchlässige Membranen vorliegen, wäre dieser enorme Energieaufwand gänzlich überflüssig. Es steht exakt dieselbe Antriebskraft zur Verfügung, aber der Mehraufwand wird um mindestens das 40-Fache zurückgefahren. Eine solche Zelle hätte keine Energie darauf verschwenden müssen, Protonen zu pumpen, daher sollte sie gut wachsen, selbst mit ihren ineffizienten frühen Enzymen, die noch nicht über Jahrmilliarden der Evolution optimiert wurden. Aber Zellen mit durchlässigen Membranen sitzen an Ort und Stelle fest und sind abhängig von den hydrothermalen Schloten, unfähig in einer anderen Umgebung zu überleben. Versiegt der Strom, sterben diese Protozellen ab. Anderseits: wenn eine durchlässige Membran genauso viel Energie oder mehr erzeugen kann, erfüllt eine undurchlässige Membran keinen Selektionsvorteil, Evolution wäre nicht möglich. Schlimmer noch: eine undurchlässige Membran würde den Protonengradienten zusammenbrechen lassen, weil keine Möglichkeit mehr besteht, die Protonen aus dem zellinneren loszuwerden. Alle Zellvarianten, die eine „modernere“ undurchlässige Membran hervorbrachten, wurden also durch die Selektion eliminiert. Natürlich nur bis sie die Fähigkeit zum Protonenpumpen entwickelten. Aber es macht keinen Sinn, Protonen durch eine durchlässige Membran zu pumpen, es gäbe keinen Selektionsvorteil, selbst wenn die Durchlässigkeit der Membran um drei Größenordnungen reduziert wird. Die verfügbare Antriebskraft bleibt dieselbe wie ohne Protonenpumpe.
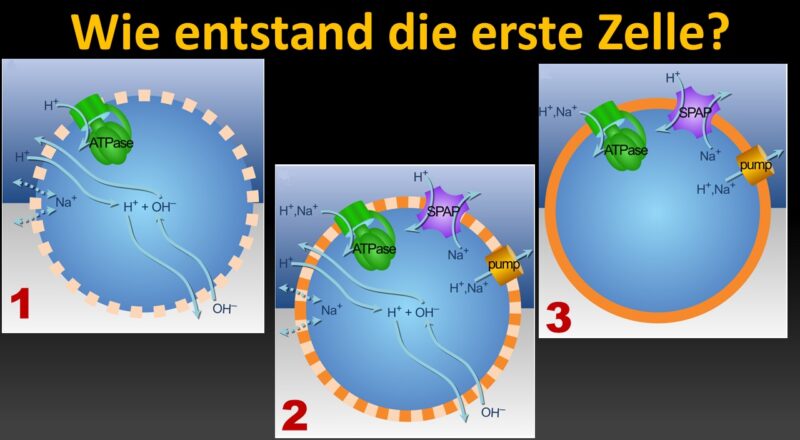
Und dennoch: moderne Zellen haben eine undurchlässige Membran und Pumpen Ionen. Was haben wir übersehen? Die Antwort: einen Antiporter (Abb. 4)!
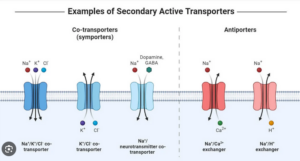
Abb. 4: Darstellung verschiedener Transportproteine der Zellmembran. Für die Ausbildung einer semipermeablen Membran ist vor allem der Na+/H+-Antiporter ganz rechts im Bild entscheidend.
Evolution des Antiporters
Einen solchen Antiporter nutzen auch die Methanogenen. Er tauscht ein Natriumion (Na+) gegen ein Proton (H+) aus. Für jedes Natriumion, das in die Zelle gelangt, wird ein Proton hinausbefördert. Es ist eine Protonenpumpe, die von einem Natriumgradienten angetrieben wird. Doch Antiporter unterscheiden nicht sehr genau, ihnen ist es egal, in welche Richtung sie arbeiten. Pumpt eine Zelle Protonen und nicht Natriumionen, läuft der Antiporter einfach rückwärts. Für jedes Proton, das in die Zelle gelangt, wird dann ein Natriumion hinausbefördert. Wenn also unsere durchlässige Zelle in ihrem alkalinen hydrothermalen Schlot einen solchen Antiporter entwickelte, würde dieser als protonengetriebene Natriumionen-Pumpe arbeiten! Theoretisch konnte der Antiporter einen natürlichen Protonengradienten in einen biochemischen Natriumgradienten umwandeln.
Lipidmembranen sind generell um etwa sechs Größenordnungen für Natriumionen undurchlässiger als für Protonen (van de Vossenberg et al. 1995). Eine Membran, die extrem durchlässig für Protonen ist, wie wir es bei der Urzelle vorliegen haben, ist demnach relativ undurchlässig für Natriumionen. Wird ein Proton hinausgepumpt, kommt es sofort wieder zurück. Wird aber ein Natriumion durch dieselbe Membran gepumpt, kehr es nicht annähernd so schnell wieder zurück.
Ein Antiporter kann demnach durch einen natürlichen Protonengradienten angetrieben werden: Für jedes Proton, das in die Zelle kommt, wird ein Natriumion hinausbefördert. Solange die Membran – wie in den Protozellen – durchlässig für Protonen ist, wird der Protonenstrom durch den Antiporter unvermindert ablaufen und die Natriumionen-Ausschleusung antreiben (Surin et al. 2007). Da die Membran weniger durchlässig für Natriumionen ist, sollte dieses eher draußen bleiben, oder genauer: Es sollte durch Membranproteine wieder in die Zelle gelangen und nicht auf direktem Wege durch die Lipide. Und das fördert die Koppelung des Natrium-Einstroms an zu leistende Arbeit.
Das ist natürlich nur von Nutzen, wenn die Membranproteine, die den Kohlenstoff- und den Energiestoffwechsel antreiben – wie die ATP-Synthase – zwischen Natriumionen und Protonen nicht unterscheiden können. Das klingt merkwürdig, könnte aber erstaunlicherweise durchaus zutreffen. Manche Methanogene haben offenbar ATP-Synthasen, die ungefähr gleich gut durch Protonen wie durch Natriumionen angetrieben werden können (Surin et al. 2007, Schlegel et al. 2012, Mulkidjanian et al. 2007, 2008, Buckel & Thauer 2013, Batista et al. 2012, Lane et al. 2010, Poehlein et al. 2012, Lane & Martin 2012). Der Grund dafür könnten die gleiche Ladung und die sehr ähnlichen Durchmesser der beiden Ionen sein. Ein Proton ist zwar viel kleiner als ein Natriumion, aber Protonen existieren nur selten in isolierter Form. In Lösung binden sie an Wasser und bilden Hydronium-Ionen (H3O+), dessen Durchmesser mit dem von Natriumionen praktisch identisch ist. Andere Membranproteine, darunter auch die Ech-Hydrogenase, sind ebenfalls promisk für Protonen und Natriumionen. Wenn ein natürlicher Protonengradient als Antrieb dient, kostet die Ausschleusung von Natriumionen praktisch keine Energie, und sobald ein Natriumgradient besteht, treten Natriumionen eher durch Membranproteine wie Ech-Hydrogenase und die ATP-Synthase wieder ein als durch die Membranlipide. Die Membran ist nun besser „gekoppelt“, also besser isoliert, und tendiert weniger zum „Kurzschließen“. Somit stehen nun mehr Ionen als Antrieb für den Kohlenstoff- und Energiestoffwechsel zur Verfügung, und der Ertrag pro ausgepumptem Ion ist größer.
Diese einfache Erfindung zieht einige überraschende Effekte nach sich. Einer ist, quasi nebenbei, das Absinken der Natriumkonzentration im Zellinneren durch das Auspumpen von Natriumionen (Mulkidjanian et al. 2012). Wir wissen, dass zahlreiche Enzyme bei Bakterien und Archaeen (etwa diejenigen, die für Transkription und Translation verantwortlich sind) durch die Selektion dahingehend optimiert wurden, dass sie bei niedrigen Natrium-Konzentrationen arbeiten, obwohl sie sich wahrscheinlich im Ozean entwickelten, wo die Natrium-Konzentration wohl auch schon vor vier Milliarden Jahren hoch war. Die Tätigkeit eines Antiporters schon zu einem frühen Zeitpunkt der Evolution könnte erklären, warum alle Zellen für ein Milieu mit niedriger Natrium-Konzentration optimiert sind, obwohl sie sich in einer Umwelt mit hoher Natrium-Konzentration entwickelten.
Für unsere Sache noch signifikanter ist die Tatsache, dass der Antiporter zusätzlich zu einem bestehenden Protonen-Gradienten noch einen Natrium-Gradienten erzeugt. Die Zelle wird nach wie vor durch den natürlichen Protonengradienten angetrieben, braucht also immer noch protonendurchlässige Membranen. Aber nun verfügt sie außerdem über einen Natriumionengradienten, der sie mit mehr Energie versorgt, als ihr zuvor allein durch Protonenkraft zur Verfügung stand. Das verschafft Zellen zwei große Vorteile. Erstens haben Zellen mit einem Antiporter mehr Antriebskraft, können also schneller wachsen und sich schneller replizieren als Zellen ohne Antiporter – der Selektionsvorteil liegt hier auf der Hand. Zweitens konnten die Zellen auch bei einem weniger ausgeprägten Protonengradienten überleben. Zellen mit einem Antiporter konnten, indem sie die Antriebskraft eines natürlichen Protonengradienten erhöhten, bei einem pH-Gradienten von weniger als zwei pH-Einheiten überleben; so konnten sie größere Bereiche des Schlotes oder angrenzende Flächen besiedeln. Zellen mit Antiporter setzten sich wohl somit gegenüber anderen Zellen eher durch und breiteten sich auch in den Schloten aus. Da sie aber immer noch vollkommen abhängig von dem natürlichen Protonengradienten waren, konnten sie die Schlote nicht verlassen. Dazu brauchte es noch einen weiteren Schritt.
Mit einem Antiporter waren die Zellen vielleicht noch nicht in der Lage, den Schlot zu verlassen, aber sie waren nun darauf vorbereitet (Abb. 5). In der Fachsprache ausgedrückt ist ein Antiporter eine „Präadaption“ – ein notwendiger erster Schritt, der eine spätere evolutionäre Entwicklung begünstigt. Ein Antiporter begünstigt erstmals die Evolution des aktiven Pumpens. Wenn Protonen nach außen gepumpt werden, kehren einige von ihnen nicht durch die durchlässige Lipidmembran, sondern durch den Antiporter zurück, der statt ihrer dann Natriumionen ausschleust. Und da die Membran für Natriumionen weniger durchlässig ist, wird ein größerer Anteil der Energie, die für das Auspumpen von Protonen aufgewandt wurde, als Ionengradient über der Membran gespeichert. Für jedes ausgepumpte Ion besteht eine leicht erhöhte Wahrscheinlichkeit, dass es draußen bleibt. Und das bedeutet, dass das Protonenpumpen im Gegensatz zu vorher nun einen kleinen Vorteil mit sich bringt. Protonenpumpen zahlt sich nur mit einem Antiporter aus. Nach der Entwicklung der Protonenpumpe brachte es nun zum ersten Mal einen Vorteil mit sich, die Membran zu verbessern.
In Anwesenheit eines Antiporters bringt es einen Vorteil mit sich, Protonen auszupumpen, die Abhängigkeit vom natürlichen Gradienten wird also verringert. Und jetzt ist es auch besser, über eine weniger durchlässige Membran zu verfügen. Eine etwas weniger durchlässige Membran vergrößert den Vorteil ein wenig, und so schreitet die Entwicklung fort, bis hin zu den heutigen, protonenundurchlässigen Membranen.
Und was noch besser ist: Diese Zwänge könnten die tiefe Divergenz von Bakterien und Archaeen erklären. Beide erzeugen ATP mithilfe von Protonengradienten über Membranen, doch sind diese Membranen in den beiden Bereichen grundlegend verschieden.
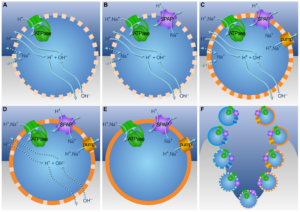
Abb. 5: (A) Ionen durchqueren die Membran als Reaktion auf Konzentrationsgradienten und elektrisches Potenzial. OH– neutralisiert ankommende Protonen. Der H+-Gradient kann den Energiestoffwechsel über die ATPase und den Kohlenstoffstoffwechsel über Ech (nicht gezeigt) antreiben. (B) Ein Na+/H+-Antiporter (SPAP) erzeugt einen Na+-Gradienten aus dem H+-Gradienten. Da Na+ weniger durchlässig ist als H+, verbessert SPAP die Kopplung, da die Membranproteine für H+ und Na+ promiskuitiv sind. (C) Membranpumpen erzeugen Gradienten, indem sie H+– oder Na+-Ionen ausstoßen. (D) Die Ausnutzung natürlicher Gradienten erfordert eine hohe Membrandurchlässigkeit, aber das Pumpen mit SPAP treibt die Entwicklung dichterer Membranen voran und erleichtert die Besiedlung weniger alkalischer Umgebungen. (E) Undurchlässige Membranen leiten den Ionenfluss durch bioenergetische Proteine, unabhängig von natürlichen Gradienten. (F) Von unten nach oben begünstigt SPAP Divergenz, Selektion für aktives Pumpen und dichtere Membranen. Pumpen und Phospholipidmembranen haben sich unabhängig voneinander in Archaeen und Bakterien entwickelt.
Unterschiede zwischen Bakterien und Archaeen
Zu guter Letzt kommen wir nun zu jenen Unterschieden im reduktiven Acetyl-Coenzym-A-Weg bei Methanogenen und Acetogenen. Diese Unterschiede legen die Vermutung nahe, dass aktives Pumpen unabhängig in zwei unterschiedlichen Populationen entstand, die sich mithilfe eines Antiporters aus einer gemeinsamen Vorläuferpopulation entwickelt hatten (Martin & Russell 2003, 2007, Koonin & Martin 2005, Lane & Martin 2012, Kaster et al. 2011, Koga et al. 1998, Lombard et al. 2012, Ragsdale & Pierce 2008 , Martin et al. 2014, Lane 2010, 2017, Lane & Martin 2012, Welte et al. 2010, Nitschke & Russel 2013, Sojo et al. 2016). Wir erinnern uns: Methanogene sind Archaeen, Acetogene Bakterien – Vertreter der beiden großen Domänen der Prokaryoten, der untersten Verzweigungen des „Stammbaums des Lebens“. Wir stellten fest, dass Bakterien und Archaeen Ähnlichkeiten in der DNA-Transkription und -Translation, in ihren Ribosomen und der Proteinsynthese aufweisen, sich aber in anderer Hinsicht grundlegend unterscheiden, darunter auch im Aufbau ihrer Zellmembran (Woese et al. 1990, Doolittle 2000a, b, Edgell & Doolittle 1997, Fox 2010, Koonin 2003, Charlebois & Doolittle 2004, Hansmann & Martin 2000, Lombard et al. 2012, Koga et al. 1998 Abb. 6).
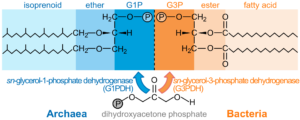
Abb. 6: Membranlipide von Archaeen und Bakterien. Archaeenlipide (links) bestehen in der Regel aus Isoprenoidketten, die durch Etherbindungen mit einem sn-Glycerin-1-Phosphat (G1P)-Grundgerüst verbunden sind. Die Chiralität der beiden Glycerin-Grundgerüste ist innerhalb jeder Gruppe nicht nur in der Struktur, sondern auch in den nicht verwandten synthetischen Enzymen vollständig konserviert. Obwohl in bakteriellen Membranen Etherbindungen beobachtet wurden (Lombard et al. 2012) und Isoprenoide allen drei Domänen gemeinsam sind, bestehen bakterielle Lipide (rechts) typischerweise aus Fettsäuren in Esterbindung an ein sn-Glycerin-3-phosphat (G3P)-Gerüst. Trotz des weit verbreiteten horizontalen Gentransfers wurde bisher kein Bakterium mit dem archaeischen Enantiomer beobachtet und umgekehrt (Koga et al. 1998).
Wie die Methanogene lassen auch die Acetogene H2 mit CO2 über eine Reihe analoger Schritte zu Acetyl-CoA reagieren. Beide Gruppen bedienen sich zum Antrieb der Protonenpumpen eines cleveren Tricks, der Elektronen-Bifurkation (Buckel & Thauer 2013, 2018). Im Grunde ist die Elektronen-Bifurkation nichts anderes als ein kurzzeitiger Energiekredit, gewährt auf das Versprechen hin, ihn umgehend zurückzuzahlen. Wie wir bereits wissen, setzt die Reaktion von H2 mit CO2 insgesamt Energie frei, doch die ersten Schritte bedürfen der Zufuhr von Energie. Durch die Elektronen-Bifurkation ist es möglich, einen Teil der in den späteren Reaktionsschritten freigesetzten Energie für die schwierigen ersten Schritte aufzuwenden. Da in den letzten Schritten mehr Energie freigesetzt wird, als in den ersten Schritten aufgewandt werden muss, kann ein Teil der Energie als Protonengradient über einer Membran gespeichert werden. Insgesamt dient die bei der Reaktion von H2 mit CO2 freigesetzte Energie als Antriebskraft für die Ausschleusung von Protonen durch eine Membran.
Die „Drähte“, über die die Elektronen-Bifurkation abläuft, sind bei Methanogenen und Acetogenen unterschiedlich (Sojo et al. 2016, Abb. 7). Beide greifen auf ähnliche Eisen-Nickel-Schwefel-Proteine zurück, aber der exakte Mechanismus ist doch nicht derselbe, so wie sich auch viele der benötigten Proteine unterscheiden. Wie die Methanogene speichern auch die Acetogene die bei der Reaktion von H2 und CO2 freigesetzte Energie als Protonen- oder Natriumionen-Gradient über einer Membran. In beiden Fällen dient der Gradient als Antriebskraft für den Kohlenstoff- und den Energiestoffwechsel. Wie die Methanogene verfügen auch die Acetogene über eine ATP-Synthase und Ech-Hydrogenase. Doch während Methanogene Ech-Hydrogenase benutzen, um den Kohlenstoff-Stoffwechsel anzutreiben, nutzen Acetogene diesen als Protonen- oder Natriumionen-Pumpe. Und der exakte Reaktionsweg, den sie zum Antreiben des Kohlenstoff-Stoffwechsels benutzen, ist grundlegend anders. Diese Unterschiede scheinen von fundamentaler Natur zu sein, so sehr, dass manche Fachleute die Ähnlichkeit zwischen beiden eher auf eine evolutionäre Konvergenz oder auf lateralen Gentransfer zurückführen als auf eine gemeinsame Abstammung (Abb. 7).
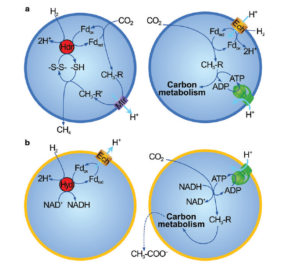
Abb. 7: Schematische Darstellung der Methanogenese und Acetogenese. (a) Methanogenese. Linke Tafel: Die flavinbasierte Elektronenbifurkation in der Methanogenese erzeugt reduziertes Ferredoxin und reduzierte Thiole. Ferredoxin wird verwendet, um CO2 zu einer Methylgruppe zu reduzieren; seine Übertragung von einem Cofaktor auf einen anderen setzt genügend Energie frei, um einen Ionengradienten (hier der Einfachheit halber als H+-Gradient dargestellt) über das Membranprotein Mtr zu erzeugen. Methan wird als Abfallprodukt ausgeschieden. Rechte Tafel: Der Ionengradient wird genutzt, um die Ferredoxin-Reduktion direkt zur Bildung einer Methylgruppe anzutreiben, die mit CO zur Bildung von Acetyl-CoA umgesetzt wird (der Einfachheit halber nicht dargestellt); ATP wird über eine Standard-Rotor-Stator-ATP-Synthase synthetisiert. Acetyl-CoA und ATP treiben zusammen den Intermediärstoffwechsel an. (b) Acetogenese. Linke Tafel: Die Elektronenbifurkation in Acetogenen erzeugt reduziertes Ferredoxin und NADH. Die Oxidation von Ferredoxin treibt die Extrusion von Ionen (typischerweise Na+, hier aber der Einheitlichkeit halber als H+ dargestellt) über das Membranprotein Ech oder Rnf an. Rechte Tafel: CO2 wird mit Hilfe von NADH und ATP reduziert, um eine Methylgruppe zu bilden. Diese wird mit CO (nicht dargestellt) zu Acetyl-CoA umgesetzt, das zusammen mit ATP zum Antrieb des Kohlenstoffstoffwechsels verwendet wird. Acetat wird ausgeschieden, um das Redox-Gleichgewicht aufrechtzuerhalten.
Doch die Ähnlichkeiten und Unterschiede ergeben allmählich einen Sinn, wenn wir davon ausgehen, dass LUCA tatsächlich von natürlichen Protonengradienten abhängig war (Abb. 8).
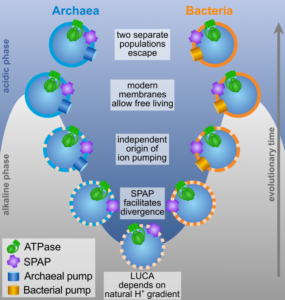
Abb. 8: Entstehung von Bakterien und Archaeen.
In der ursprünglichen Vorläuferpopulation diente der normale Einstrom von Protonen via Ech-Hydrogenase dazu, Ferredoxin zu reduzieren, was seinerseits die CO2-Reduktion antrieb. Zwei getrennte Populationen entwickelten dann unabhängig voneinander das Protonenpumpen. Eine Population, aus der sich die Acetogene entwickeln sollten, kehrte die Arbeitsrichtung der Ech-Hydrogenase um, sodass nun Ferredoxin oxidiert und die freigesetzte Energie dazu benutzt wurde, Protonen aus der Zelle zu pumpen. Das ist eine feine und einfache Sache, schafft aber ein unmittelbares Problem. Das vorher zur Reduktion von Kohlenstoffverbindungen benutzte Ferredoxin dient nun dazu, Protonen zu pumpen. Die Acetogene mussten einen neuen Weg finden, Kohlenstoffverbindungen zu reduzieren, der nicht vom Ferredoxin abhängig war. Und ihre Vorfahren fanden eine Methode – den cleveren Trick der Elektronen-Bifurkation, dank derer sie in der Lage waren, CO2 indirekt zu reduzieren.
Die zweite Population, aus der die Methanogene hervorgingen, fand einen anderen Weg. Wie ihre Vorfahren nutzten sie weiterhin Protonengradienten zur Reduktion von Ferredoxin und anschließend Ferredoxin zur Fixierung von Kohlenstoff. Doch sie mussten aus dem Nichts eine Protonenpumpe „erfinden“. Es scheint so, als hätten sie einen Antiporter zur Pumpe umgemodelt. Das ist an sich nicht schwierig, birgt aber ein Problem: Wie soll die Pumpe angetrieben werden? Die Methanogene entwickelten eine andere Form der Elektronen-Bifurkation; dabei benutzten sie zum Teil dieselben Proteine wie die Acetogene, aber ganz anders geschaltet, denn ihre eigenen Bedürfnisse waren andere und mit einer anderen Pumpe verknüpft.
Sobald die Zellen Antiporter und Ionenpumpen hatten, war es von Vorteil, die Membranlipide mit Glycerinköpfen zu versehen. Und die beiden Domänen scheinen das unabhängig voneinander vollbracht zu haben, wobei die Archaeen ein Stereoisomer des Glycerins benutzten und die Bakterien dessen spiegelbildliches Gegenstück.
Die Zellen hatten nun aktive Ionenpumpen und moderne Membranen, sodass sie schließlich die Schlote verlassen und in die Freiheit des offenen Meeres vordringen konnten (Abb. 9). Aus einem gemeinsamen Vorfahren, der von Protonengradienten in hydrothermalen Schloten lebte, gingen unabhängig voneinander die ersten frei lebenden Zellen, Bakterien und Archaeen, hervor. Es überrascht nicht, dass Bakterien und Archaeen auch unterschiedliche Zellwände entwickelten, die sie gegen die neuartigen Einwirkungen schützten, und dass sie die DNA-Replikation womöglich unabhängig voneinander „erfunden“ haben. Bakterien binden ihre DNA während der Zellteilung an die Zellmembran; diese Bindung sorgt dafür, dass jede Tochterzelle eine Kopie des Genoms erhält. Die molekulare Maschinerie für die Bindung der DNA an die Membran und viele Einzelheiten der DNA-Replikation müssen zumindest teilweise von der Mechanik dieser Bindung abhängen. Die Tatsache, dass sich Zellmembranen unabhängig voneinander entwickelten, liefert eine erste Erklärung dafür, dass die DNA-Replikation bei Bakterien und Archaeen so unterschiedlich abläuft. Dasselbe gilt größtenteils für die Zellwände, deren Bestandteile allesamt aus dem Zellinneren durch spezifische Membranporen ausgeschleust werden müssen – daher hängt die Synthese der Zellwand von den Eigenschaften der Zellmembran ab und muss sich bei Bakterien und Archaeen unterscheiden.
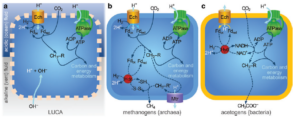
Abb. 9: Vorgeschlagene Evolution des Protonenpumpens in Archaeen und Bakterien. (a) Vorgeschlagener Kohlenstoff- und Energiestoffwechsel, angetrieben durch natürliche Protonengradienten in einer Urprotozelle mit undichter Membran. Ech, energieumwandelnde Hydrogenase; Fdox, Ferredoxin; Fdred, reduziertes Ferredoxin. Der Einfachheit halber wird in dieser Abbildung nur der -CH3-Zweig eines postulierten Acetyl-CoA-Wegs dargestellt. R ist einer von mehreren Cofaktoren, die sich zwischen Methanogenen und Acetogenen unterscheiden. Die Richtung des Protonenflusses durch Ech ist entscheidend und unterscheidet sich in (c). (b) Vereinfachter Kohlenstoff- und Energiestoffwechsel von Methanogenen (Archaeen). Ech reduziert Ferredoxin mit Hilfe eines Protonengradienten wie in (a), aber jetzt müssen Ionen gepumpt werden, um das Membranpotential zu regenerieren. Dies wird durch Elektronenverzweigung erreicht, wobei H2 zur gleichzeitigen Reduktion von Ferredoxin und einem Heterodisulfid (-S-S-) verwendet wird. Die Methanogenese regeneriert das Membranpotenzial über eine neue Ionenpumpe (Mtr), die sich möglicherweise aus einem Na+/H+-Antiporter entwickelt hat. (c) Vereinfachter Kohlenstoff- und Energiestoffwechsel von Acetogenen (Bakterien). Ech kehrt sich um und oxidiert Ferredoxin, um ein Membranpotenzial zu erzeugen. Nun muss das reduzierte Ferredoxin regeneriert werden, was durch eine besondere Form der Elektronenverzweigung erreicht wird, bei der H2 zur gleichzeitigen Reduktion von Ferredoxin und NAD+ verwendet wird. Acetogene können Ferredoxin jedoch nicht mehr zur CO2-Reduktion verwenden und mussten daher einen neuen Weg des Kohlenstoffstoffwechsels entwickeln, bei dem ATP und NADH anstelle von Ferredoxin verwendet werden. Neue Wege des Energiestoffwechsels Stoffwechselwege sind mit gestrichelten Linien dargestellt.
Literatur
Amend, J.P., McCollom, T.M. (2009): Energetics of biomolecule synthesis on early earth. In Chemical Evolution II: From the Origins of Life to Modern Society, L. Zaikowski, J.M. Friedrich, and S.R. Seidel, eds. (Washington, DC: American Chemical Society), pp. 63–94.
Arndt, N., Nisbet, E. (2012): Processes on the young earth and the habitats of early life. Annu. Rev. Earth Planet. Sci. 40, 521–549
Batista, A.P., Marreiros, B.C., Pereira, M.M. (2012): The role of proton and sodium ions in energy transduction by respiratory complex I. IUBMB Life 64, 492–498.
Berg, A. I. (2011): Ecological aspects of the distribution of different autotrophic CO2 fixation pathways. Appl. Environ. Microbiol. 77, 1925–1936.
Brabender M, Henriques Pereira DP, Mrnjavac N, Schlikker ML, Kimura ZI, Sucharitakul J, Kleinermanns K, Tüysüz H, Buckel W, Preiner M, Martin WF. (2024): Ferredoxin reduction by hydrogen with iron functions as an evolutionary precursor of flavin-based electron bifurcation. Proc Natl Acad Sci U S A. 2024 121(13):e2318969121.
Buckel, W., Thauer, R.K. (2012): Energy conservation via electron bifurcating ferredoxin reduction and proton/Na+ translocating ferredoxin oxidation (Biochim. Biophys. Acta).
Buckel, W., Thauer, R.K. (2018): Flavin-based electron bifurcation, a new mechanism of biological energy coupling. Chem. Rev. 118, 3862–3886.
Charlebois, R. L., Doolittle W. F. (2004): Computing prokaryotic gene ubiquity: rescuing the core from extinction. Genome Res. 14, 2469–2477.doi:10.1101/gr.3024704
Doolittle, W. F. (2000a): Searching for the common ancestor. Research in Microbiology 151: 85–89.
Doolittle W. F. (2000b): Uprooting the tree of life. Sci Am. 282(2):90-5.
Edgell, D. R., Doolittle, W. F. (1997): Archaea and the origin(s) of DNA replication proteins. Cell 89: 995–98.
Fox, G. E. (2010): Origin and evolution of the ribosome. Cold Spring Harb. Perspect. Biol. 2(9), A003483
Fuchs, G. (2011): Alternative pathways of carbon dioxide fixation: insights into the early evolution of life? Annu. Rev. Microbiol. 65, 631–658.
Hansmann S., Martin, W. (2000): Phylogeny of 33 ribosomal and six other proteins encoded in an ancient gene cluster that is conserved across prokaryotic genomes. Int. J. Syst. Evol. Microbiol. 50, 1655–1663
Harold, F.M. (1986): The Vital Force: A Study of Bioenergetics (New York: W.H. Freeman).
Herrmann, G., et al. (2008): Energy conservation via electron-transferring flavoprotein in anaerobic bacteria. J. Bacteriol. 190, 784–791.
Kaster, A.-K., Moll, J., Parey, K., Thauer, R.K. (2011): Coupling of ferredoxin and heterodisulfide reduction via electron bifurcation in hydrogenotrophic methanogenic archaea. Proc. Natl. Acad. Sci. USA 108, 2981–2986.
Katsyv, A. et al. (2023): Molecular basis of the electron bifurcation mechanism in the [FeFe]-hydrogenase complex HydABC. J. Am. Chem. Soc. 145, 5696–5709.
Kayastha, K. (2021): Flavins in the electron bifurcation process. Arch. Biochem. Biophys. 701, 108796.
Kelley, D.S., Baross, J.A., Delaney, J.R. (2002): Volcanoes, fluids, and life at mid-ocean ridge spreading centers. Annu. Rev. Earth Planet. Sci. 30, 385–491.
Koga Y, Kyuragi T, Nishihara M, Sone N. (1998): Did archaeal and bacterial cells arise independently from noncellular precursors? A hypothesis stating that the advent of membrane phospholipid with enantiomeric glycerophosphate backbones caused the separation of the two lines of descent. Journal of Molecular Evolution 46: 54–63.
Koonin, E. V. (2003): Comparative genomics, minimal gene-sets, and the last universal common ancestor. Nature Reviews Microbiology 1: 127–136.
Koonin, E.V., Martin, W. (2005): On the origin of genomes and cells within inorganic compartments. Trends Genet. 21, 647–654.
Lane, N. (2010): Why Are Cells Powered by Proton Gradients? Nature Education 3(9):18
Lane, N. (2017): Der Funke des Lebens Energie und Evolution. Konrad Theis Verlag
Lane, N., Allen, J.F., Martin, W. (2010): How did LUCA make a living? Chemiosmosis in the origin of life. Bioessays 32, 271–280.
Lane, N, Martin, W. (2012): The origin of membrane bioenergetics. Cell 151: 1406–16.
Li, F. et al. (2008): Coupled ferredoxin and crotonyl coenzyme A (CoA) reduction with NADH catalyzed by the butyryl-CoA dehydrogenase/Etf complex from Clostridium kluyveri. J. Bacteriol. 190, 843–850.
Lombard J, López-García P, Moreira D. (2012): The early evolution of lipid membranes and the three domains of life. Nature Reviews in Microbiology 10: 507–15.
Lubner, E. et al. (2017): Mechanistic insights into energy conservation by flavin-based electron bifurcation. Nat. Chem. Biol. 13, 655–659.
Maden, B.E.H. (1995): No soup for starters? Autotrophy and the origins of metabolism. Trends Biochem. Sci. 20, 337–341.
Martin, W., Russell, M.J. (2003): On the origins of cells: a hypothesis for the evolutionary transitions from abiotic geochemistry to chemoautotrophic prokaryotes, and from prokaryotes to nucleated cells. Philos. Trans. R. Soc. Lond. B Biol. Sci. 358, 59–83, discussion 83–85.
Martin, W., Russell, M.J. (2007): On the origin of biochemistry at an alkaline hydrothermal vent. Philos. Trans. R. Soc. Lond. B Biol. Sci. 362, 1887–1925.
Martin, W., Sousa, F. L., Lane, N. (2014): Energy at life’s origin. Science 344: 1092–93.
Mitchell P. (1961): Coupling of phosphorylation to electron and hydrogen transfer by a chemiosmotic type of mechanism. Nature 191:144-148.
Mulkidjanian, A.Y., Bychkov, A.Y., Dibrova, D.V., Galperin, M.Y., Koonin, E.V. (2012): Origin of first cells at terrestrial, anoxic geothermal fields. Proc. Natl. Acad. Sci. USA 109, E821–E830.
Mulkidjanian, A.Y., Galperin, M.Y., Makarova, K.S., Wolf, Y.I., Koonin, E.V. (2008): Evolutionary primacy of sodium bioenergetics. Biol. Direct 3, 13
Mulkidjanian, A.Y., Makarova, K.S., Galperin, M.Y., Koonin, E.V. (2007): Inventing the dynamo machine: the evolution of the F-type and V-type ATPases. Nat. Rev. Microbiol. 5, 892–899.
Müller, V., Chowdhury, N. P., Basen, M. (2018): Electron bifurcation: A long-hidden energy-coupling mechanism. Annu. Rev. Microbiol. 72, 331–353.
Nitschke, W., Russell, M. J. (2013): Beating the acetyl coenzyme A-pathway to the origin of life. Philos Trans R Soc Lond B Biol Sci.;368(1622):20120258.
Phillips, R., Milo, R. (2009): A feeling for the numbers in biology. Proc. Natl. Acad. Sci. USA 106, 21465–21471
Poehlein, A., Schmidt, S., Kaster, A.-K., Goenrich, M., Vollmers, J., Thürmer, A., Bertsch, J., Schuchmann, K., Voigt, B., Hecker, M., et al. (2012): An ancient pathway combining carbon dioxide fixation with the generation and utilization of a sodium ion gradient for ATP synthesis. PLoS ONE 7, e33439.
Pohorille, A., Deamer, D.W. (2009): Self-assembly and function of primitive cell membranes. Res. Microbiol. 160, 449–456.
Ragsdale SW, Pierce E. (2008): Acetogenesis and the Wood-Ljungdahl pathway of CO2 fixation. Biochimica Biophysica Acta 1784: 1873–98.
Russell, M.J., Daniel, R.M., Hall, A. (1993): On the emergence of life via catalytic iron-sulphide membranes. Terra Nova 5, 343–347.
Russell, M.J., Hall, A.J. (1997): The emergence of life from iron monosul- phide bubbles at a submarine hydrothermal redox and pH front. J. Geol. Soc. London 154, 377–402.
Say, R.F., Fuchs, G. (2010): Fructose 1,6-bisphosphate aldolase/phosphatase may be an ancestral gluconeogenic enzyme. Nature 464, 1077–1081.
Schlegel, K., Leone, V., Faraldo-Gomez, J.D., and Müller, V. (2012): Promiscuous archaeal ATP synthase concurrently coupled to Na + and H + translocation. Proc. Natl. Acad. Sci. USA 109, 947–952.
Schuchmann, K., Müller, V. (2012): A bacterial electron-bifurcating hydrogenase. J. Biol. Chem. 287, 31165–31171.
Shock, E., Canovas, P. (2010): The potential for abiotic organic synthesis and biosynthesis at seafloor hydrothermal systems. Geofluids 10, 161–192.
Shock, E.L., McCollom, T., Schulte, M.D. (1998): The emergence of metabolism from within hydrothermal systems. In Thermophiles: the Keys to Molecular Evolution and the Origin of Life, J. Wiegel and M.W.W. Adams, eds. (Washington, DC: Taylor and Francis), pp. 59–76
Sleep, N. H., Bird, D. K., Pope, E. C. (2011): Serpentinite and the dawn of life. Philos. Trans. R. Soc. Lond. B Biol. Sci. 366, 2857–2869.
Sojo, V., Herschy, B., Whicher, A., Camprubí, E., Lane, N. (2016): The Origin of Life in Alkaline Hydrothermal Vents. Astrobiology, 16(2), 181–197.
Sojo V, Pomiankowski A, Lane N (2014): A Bioenergetic Basis for Membrane Divergence in Archaea and Bacteria. PLoS Biol 12(8): e1001926.
Stetter, K.O. (2006): Hyperthermophiles in the history of life. Philos. Trans. R. Soc. Lond. B Biol. Sci. 361, 1837–1842, discussion 1842–1843.
Surin, S., Cubonova, L., Majernik, A.I., McDermott, P., Chong, J.P.J., and Smigan, P. (2007): Isolation and characterization of an amiloride-resistant mutant of Methanothermobacter thermautotrophicus possessing a defective Na +/H + antiport. FEMS Microbiol. Lett. 269, 301–308.
Tamblyn, R., Hermann, J. (2023): Geological evidence for high H2 production from komatiites in the Archaean. Nat. Geosci. 16, 1194–1199.
Thauer, R.K., Kaster, A.-K., Seedorf, H., Buckel, W., Hedderich, R. (2008): Methanogenic archaea: ecologically relevant differences in energy conservation. Nat. Rev. Microbiol. 6, 579–591
van de Vossenberg, J.L.C.M., Ubbink-Kok, T., Elferink, M.G.L., Driessen, A.J.M., and Konings, W.N. (1995): Ion permeability of the cytoplasmic membrane limits the maximum growth temperature of bacteria and archaea. Mol. Microbiol. 18, 925–932.
Wimmer, J. L. E., Vieira, A. D. N., Xavier, J. C., Kleinermanns, K., Martin, W. F., and Preiner, M. (2021a): The autotrophic core: an ancient network of 404 reactions converts H2, CO2, and NH3 into amino acids, bases, and cofactors. Microorganisms 9:458.
Wimmer JLE, Xavier JC, Vieira ADN, Pereira DPH, Leidner J, Sousa FL, Kleinermanns K, Preiner M, Martin WF (2021): Energy at Origins: Favorable Thermodynamics of Biosynthetic Reactions in the Last Universal Common Ancestor (LUCA). Front Microbiol. 13;12:793664