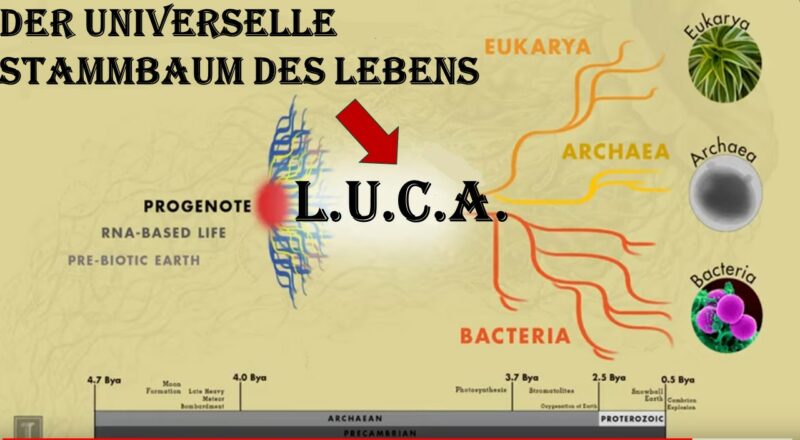
Einführung
Text als pfd
Eine der Grundeigenschaften des Lebens ist der Stoffwechsel. Im letzten Beitrag legten wir dar, dass Stoffwechsel eine Form der chemischen Reaktion ist und dass die weißen Raucher Bedingungen schaffen, wie sich die ersten Stoffwechselprozesse entwickelt haben können.[1] Dort lernten wir die Reaktion von CO2 mit Wasserstoffgas kennen. Obwohl beide Stoffe nicht einfach so miteinander reagieren, erlaubte die physikalische Struktur der weißen Raucher – natürliche Protonengradienten über dünne halbleitende Wände – ihre Reaktion und die Bildung von organischen Stoffen. Solche Protonengradienten sind die treibende Kraft, um den Stoffwechsel anzutreiben. Auch heute noch sind einige urtümliche Mikroorganismen in der Lage CO2 mit Wasserstoffgas reagieren zu lassen, um daraus organische Moleküle entstehen zu lassen.
Aber wie können wir sicher sein, dass gerade dieser Stoffwechselweg der ursprünglichere ist?
Um das zu ergründen, müssen wir einen anderen Ansatz wählen als in den bisherigen. Unser bisheriges Vorgehen war ein bottom-up-Verfahren, also von unten nach oben. Wir fingen mit der Entstehung der Biomoleküle aus anorganischen Molekülen an, gingen über zur Polymerisation und zu den ersten chemischen Reaktionen und Ausbildung von Urzellen in den Mineralwänden der weißen Raucher. Eine andere Methode ist die des „Top-down“ (Camprubi et al. 2019). Top-down geht vom Allgemeinen schrittweise hin zum Konkreten. In unserem Fall würden wir von den Stoffwechselprozessen heute lebender Organsimen ausgehen und daraus ergründen, welche der ursprünglichere ist. Dies ist möglich durch die phylogenetische Rekonstruktion der Lebewesen (Abb. 1). Eine universelle Phylogenie sollte es uns ermöglichen, über die zelluläre Natur hinaus weitere Merkmale der frühesten Lebensformen abzuleiten. Hier werden wir natürlich einige Bereiche anreißen, die wir im Detail noch gar nicht besprochen haben, z. B. die Speicherung der genetischen Information. Aber diese Top-Down-Methode ermöglicht es uns in den weiteren Episoden sich auf das Wesentliche zu konzentrieren.
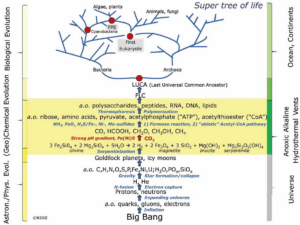
Abb. 1: Superstammbaum des Lebens auf der Erde auf der Grundlage der Theorie der alkalischen Hydrothermalquellen. Man beachte, dass die astronomische und physikalische Evolution und ein Teil der (geo)chemischen Evolution universell ist und zu einer ersten lebenden Zelle (FLC) führen kann, die in gewissem Maße der auf der Erde entstandenen Zelle ähnelt. FPE = erster photosynthetischer Eukaryote
Universeller Stammbaum des Lebens
Als Biologen Methoden entwickelten, um die Sequenzen der Aminosäuren in den Proteinen und die Sequenzen der Nukleotide in der DNA und RNA zu lesen, wurde schnell eine neue Technik zur Schätzung der Stammesgeschichte entwickelt (Zuckerkandl & Pauling 1965). Einige der Details dieser Technik sind tückisch, aber die Grundidee ist einfach. Stellt euch vor, wir haben eine Gruppe von Arten, die alle ein bestimmtes Gen in ihrem Genom tragen. Wir können die Nukleotidsequenz dieses Gens bei jeder Art ablesen und dann die Sequenzen vergleichen. Wenn die Arten eng miteinander verwandt sind, sollten ihre Sequenzen ziemlich ähnlich sein. Befinden sich die Arten auf weit entfernten Ästen des Stammbaums, sollten ihre Sequenzen weniger ähnlich sein. Folglich können wir die relative Ähnlichkeit der Sequenzen der Arten nutzen, um Rückschlüsse auf ihre evolutionären Beziehungen ableiten. Wir platzieren Arten mit ähnlicheren Sequenzen auf benachbarte Äste des Stammbaums und Arten mit weniger ähnlichen Sequenzen auf weiter entfernte Äste.
Die Herausforderung bei der Verwendung von Sequenzdaten zur Schätzung des Stammbaums aller Lebewesen besteht darin, ein Gen zu finden, das erkennbare Sequenzähnlichkeiten selbst zwischen so weit voneinander entfernten Arten wie Escherichia coli und Homo sapiens aufweist (Woese 1991). Wir brauchen ein Gen, das in allen Organismen vorkommt und für ein Produkt kodiert, dessen Funktion essentiell ist und daher einer starken Selektion unterliegt. Ohne diese Selektion würden innerhalb von Milliarden von Jahren der Evolution alle erkennbaren Ähnlichkeiten in den Sequenzen von entfernt verwandten Organismen ausgelöscht werden. Auch die Funktion des Gens muss in allen Organismen gleichgeblieben sein. Denn wenn sich die Funktion eines Genprodukts bei einigen Arten ändert, bei anderen aber nicht, kann die Selektion auf die neue Funktion eine rasche Divergenz in der Nukleotidsequenz bewirken, die die Arten entfernter erscheinen lässt, als sie tatsächlich sind.
Ein Gen, das alle Kriterien für die Rekonstruktion der universellen Phylogenie erfüllt, ist das Gen, das für die kleine Untereinheit der ribosomalen RNA, kurz rRNA, kodiert (Woese & Fox 1977a, b, Woese 1968, 1991, 1998, 2000, 2002 Woese et al. 1990, Sapp & Fox 2013, Abb. 2).
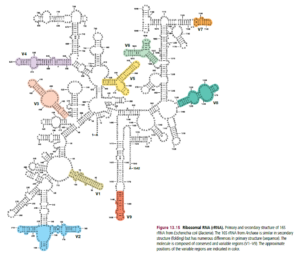
Abb. 2: Struktur der kleinen Untereinheit der rRNA (SSU rRNA) bei E. coli.
Alle Organismen haben Ribosomen, und in allen Organismen haben die Ribosomen mit ähnlicher Struktur und Zusammensetzung, die sowohl rRNA als auch Protein umfasst. Die Funktion der Ribosomen ist in allen Organismen gleich: Sie sind die Maschinen, die für die Proteinsynthese verantwortlich sind (Woese 2002, Smith et al. 2008, Fox 2010, Monro 1967, Petrov et al. 2014a, b, 2015, Lanier et al. 2016, Bernier et al. 2018, Abb. 3).
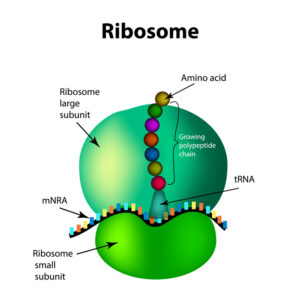
Abb. 3: Aufbau und Funktion des Ribosoms
Diese ist so lebenswichtig, und die Organismen unterliegen einer so starken natürlichen Selektion, dass die rRNAs von Menschen und ihren Darmbakterien große Ähnlichkeiten in der Nukleotidsequenz aufweisen, obwohl Menschen und Bakterien zuletzt vor Milliarden von Jahren einen gemeinsamen Vorfahren hatten. Die kleine Untereinheit der rRNA wurde von Carl R. Woese, dem Hauptpionier bei der Verwendung molekularer Sequenzen zur Schätzung der universellen Phylogenie, ausgewählt (Fox et al. 1977, Woese & Fox 1977b; siehe auch Doolittle & Brown 1994).
Die Ergebnisse dieser phylogenetischen Analyse brachten erstaunliches zu Tage, die das bis dahin gültige traditionelle Bild der Einteilung der Lebewesen revolutionierten. Die belebte Welt teilte man ursprünglich in fünf Königreiche ein (Whittaker 1969, Margulis 1974): Monera, Pflanzen, Tiere, Pilze und Protista. Monera sind die Prokaryoten, also die Zellen ohne Zellkern, gewöhnlich als Bakterien bezeichnet und die Protista sind die einzelligen Lebewesen mit Zellkern (Abb. 4).
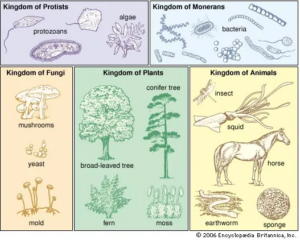
Abb. 4: Die fünf Königreiche der Lebenswelt
Die rRNA-Phylogenie des gesamten Lebens führte zu einer dramatischen Revision unserer traditionellen Sichtweise der Organisation des Lebens, da sie zeigt, dass das System der fünf Königreiche nur eine begrenzte Ähnlichkeit mit den tatsächlichen evolutionären Beziehungen hat (Woese et al. 1990; für eine gegenteilige Ansicht siehe Margulis 1996).
Denn statt von den 5 Königreichen, haben wir drei Domänen: Bacteria, Archaea und Eukarya (Abb. 5).
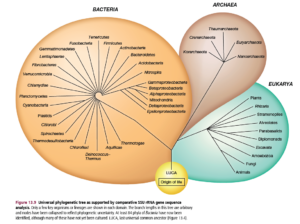
Abb. 5: Domänen des Lebens
Die Prokaryoten beispielsweise, die in der traditionellen Klassifizierung alle im Königreich Monera zusammengefasst sind, nehmen zwei der drei Hauptzweige des rRNA-Baums ein. Einer dieser Zweige, die Bacteria, umfasst praktisch alle bekannten Prokaryonten. Der andere Zweig der Prokaryoten, die Archaea, ist nicht so bekannt. Viele der Archaea leben in extremen Umgebungen, lassen sich nur schwer in Kulturen züchten und wurden erst kürzlich entdeckt (siehe Madigan & Marrs 1997). Viele von ihnen sind zum Beispiel hyperthermophil und leben in heißen Quellen bei Temperaturen von bis zu 110°C, andere sind anaerobe Methanproduzenten und wieder andere, die Haloarchaea, sind stark salztolerant und werden daher als extrem halophil bezeichnet (Abb. 6).
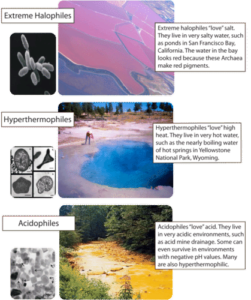
Abb. 6: extreme Lebensräume der Archaea
Aufgrund ihrer prokaryotischen Zellstruktur wurden die Archaea ursprünglich als Bakterien angesehen. Als Woese und Kollegen entdeckten, dass diese Organismen nur entfernt mit den übrigen Bakterien verwandt sind, benannten sie sie in Archaebakterien um (Fox et al. 1977, Woese & Fox 1977b). Schließlich erkannten die Biologen, dass die Archaebakterien tatsächlich enger mit den Eukaryoten verwandt sind als mit den echten Bakterien (siehe Bult et al. 1996, Olsen & Woese 1996). In Anerkennung dieser Tatsache schlugen Woese et al. (1990) eine neue Klassifizierung vor. Diese geht von den erwähnten drei Domänen des Lebens aus: den Bakteria, Archaea und Eukarya.
Die Protista, die in der traditionellen Klassifizierung ein einziges Reich bilden, sind über Zweige innerhalb der Eukarya verstreut. Die Diplomonaden beispielsweise, zu denen der Darmparasit Giardia lamblia gehört, bilden einen der tiefsten Zweige der Eukarya. Sie sind deutlich von anderen Protisten wie den Flagellaten, zu denen Euglena gehört, und den Ciliaten, zu denen Paramecium gehört, getrennt (Abb. 7).
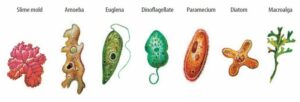
Abb. 7: Protista
Die verbleibenden drei Reiche der traditionellen Klassifikation, die Tiere, Pflanzen und Pilze, bedürfen zwar nur einer geringfügigen Überarbeitung, aber es zeigt sich, dass in dem universellen rRNA-Stammbaum diese nur einzelne Zweige innerhalb der Domäne der Eukarya ausmachen. Die vielzelligen, makroskopischen Organismen dieser drei Reiche sind Neulinge auf der evolutionären Bühne; sie haben einen relativ jungen letzten gemeinsamen Vorfahren. Bei Genen, die von allen Organismen geteilt werden, wie z. B. das Gen für die kleine Untereinheit der rRNA, scheinen Tiere, Pflanzen und Pilze weniger als 10 % der auf der Erde beobachteten Vielfalt zu besitzen (Olsen & Woese 1996).
Die Wurzel des Stammbaums der drei Reiche markiert den gemeinsamen Vorfahren, den last universal common ancestor – oder kurz LUCA. Diesem Baum zufolge haben sich die Nachkommen von LUCA zu den Bakterien auf der einen Seite und den Archaea-Eucarya auf der anderen Seite entwickelt. Basierend auf den Arbeiten aller Forschergruppen kommen viele zu der Erkenntnis, dass die Archaea mit den Eukarya näher verwandt sind als mit den Bakterien (Gogarten et al. 1989, Iwabe et al. 1989, Brown & Doolittle 1995, Baldauf et al. 1996). Doch nicht immer erweist sich das verwandtschaftliche Verhältnis zwischen den drei Gruppen als eindeutig und die Wurzel des Stammbaums des Lebens war und ist ein aktives Forschungsfeld (siehe Zhaxybayeva et al. 2005, Dagan et al. 2010, Fournier & Gogarten 2010).
Horizontaler Gentransfer
Wir würden eigentlich erwarten, dass die Schätzungen der universellen Phylogenie, die auf verschiedenen Genen basieren, weitgehend deckungsgleich sind. Tatsächlich sind sie das aber nicht. James R. Brown und W. Ford Doolittle (Brown & Doolittle 1997) schätzten die Gesamtstammesgeschichte auf der Grundlage von etwa vier Dutzend Genen (Abb. 8). Gene für Proteine, die an der Speicherung und Verarbeitung genetischer Informationen beteiligt sind, ergeben häufig einen Baum, der mit dem Baum für die rRNA übereinstimmt (Abb. 8a). Gene für Proteine, die am Stoffwechsel beteiligt sind, ergeben jedoch oft einen Baum, in dem die Bakterien und die Archaeen am nächsten verwandt sind (Abb. 8b). Wieder andere Gene ergeben einen Baum, in dem die Bakterien und die Eukaryonten die engsten Verwandten sind (Abb. 8c), oder in dem es eine ungelöste Trichotomie der drei Domänen gibt (Abb. 8d). Wie lässt sich die Unstimmigkeit zwischen den aus verschiedenen Genen geschätzten Phylogenien erklären? Viele Forscher, darunter auch Carl Woese (1998, 2000, 2002), argumentieren, dass die Konflikte zwischen den Datensätzen zu zahlreich und anhaltend sind, um diese zu ignorieren.
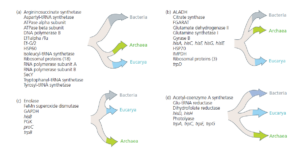
Abb. 8: Verschiedene Gene ergeben unterschiedliche Schätzungen der universellen Phylogenie. Als James Brown & Ford Doolittle (1997) den Stammbaum des Lebens unter Verwendung einer Vielzahl von Genen rekonstruierten, stellten sie fest, dass unterschiedliche Gene zu unterschiedlichen Phylogenien führen. Einige Gene ergeben Bäume, in denen Archaea und Eucarya die engsten Verwandten sind (a); andere ergeben Bäume, in denen Bakterien und Archaea die engsten Verwandten sind (b). Wieder andere Gene ergeben Bäume, in denen Bacteria und Eucarya die engsten Verwandten sind (c), oder in denen die Beziehungen zwischen den drei Domänen ungelöst sind (d). Aus Brown & Doolittle (1997).
Die mögliche Lösung ist eine Form des Genaustausches, der bei Tieren und Pflanzen extrem selten vorkommt: der horizontale Gentransfer (Koonin 2016, Gogarten et al. 2002, Abb. 9). Horizontaler Gentransfer (HGT) bezeichnet eine Übertragung von genetischem Material nicht entlang der Abstammungslinie, also nicht von einer Generation zur darauffolgenden, sondern „horizontal“ von einem Organismus in einen bereits existierenden anderen hinein. Im Unterschied dazu erfolgt der vertikale Gentransfer von Vorfahren zu Nachkommen.
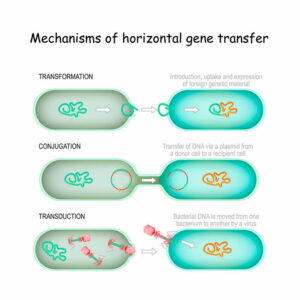
Abb. 9: Mechanismen des horizontalen Gentransfers
Die Vorstellung von einem Baum des Lebens geht von einer „vertikalen“ Vererbung aus, bei der die Eltern Kopien der Gene durch sexuelle Fortpflanzung an ihre Nachkommen weitergeben. Über Generationen hinweg werden die Gene hauptsächlich innerhalb einer Art weitergegeben. Populationen, die reproduktiv isoliert sind, divergieren im Laufe der Zeit langsam, da die Interaktionen zwischen ihnen abnehmen, und bilden schließlich neue Arten. Daraus ergibt sich der verzweigte Baum des Lebens. Das trifft praktisch auf alle Eukaroyten zu, bei der die vertikale Vererbung dominiert und horizontaler Gentransfer praktisch nicht vorkommt. Bei Bakterien und Archeen ist die Sache nicht so eindeutig. Sie haben keinen Geschlechtsverkehr wie die Eukaryoten, bilden also auch nicht auf dieselbe Weise eine schöne, saubere Art. Die eigentliche Schwierigkeit bei Bakterien besteht jedoch darin, dass sie ihre Gene durch horizontalen Gentransfer verbreiten können, indem sie eine Handvoll Gene wie Kleingeld von einem zum anderen weitergeben. Nichts davon untergräbt die Evolution in irgendeiner Weise – es handelt sich immer noch um Abstammung mit Modifikation; es ist nur so, dass die “Modifikation” auf mehr Arten erreicht wird, als wir früher dachten (House 2009, Puigbò et al. 2009, 2010, Abby et al. 2012, Fitz Gibbon & House 1999, Snel et al. 1999).
Das Gen, auf welches die Einteilung der drei Domänen geschlossen wurde, die kleine Untereinheit der rRNA, wird selten, wenn überhaupt, durch horizontalen Gentransfer vererbt.
Wir können die rRNA eines Adlers sequenzieren, und sie wird uns sagen, dass es sich um einen Vogel handelt. Wir können daraus schließen, dass er einen Schnabel, Federn, Krallen und Flügel hat. Das ist so, weil die vertikale Vererbung gewährleistet, dass es immer eine gute Korrelation zwischen dem ribosomalen “Genotyp” und dem allgemeinen “Phänotyp” gibt: Die Gene, die all diese vogelähnlichen Merkmale kodieren, sind Mitreisende; sie segeln gemeinsam durch die Generationen. Sie werden im Laufe der Zeit zwar verändert, aber nur selten auf sehr dramatische Weise.
Stellen wir uns nun aber vor, dass der horizontale Gentransfer überwiegt. Wir sequenzieren also die rRNA und erfahren, dass wir es mit einem Vogel zu tun haben. Erst jetzt sehen wir uns diesen “Vogel” genauer an und es stellt sich heraus, dass er sechs Beine, Augen auf den Knien, und ein Fell hat; er dazu einen Froschlaich, hat keine Flügel und heult wie eine Hyäne. Ja, das ist natürlich absurd; aber genau das ist das Problem beim horizontalen Gentransfer bei Bakterien. Monströse Chimären starren uns regelmäßig ins Gesicht; weil Bakterien typischerweise klein und morphologisch einfach sind, schreien wir nicht auf.
Das Problem ist, dass man Anhand der rRNA nicht alleine auf ursprüngliche Stoffwechselprozesse von LUCA ableiten kann. Wenn die Rate des horizontalen Gentransfers gering ist (wie bei Pflanzen und Tieren, vielen Protisten und einigen Bakterien), dann gibt es eine gute Korrelation zwischen ribosomalem Genotyp und Phänotyp. Wenn die horizontale Gentransferrate jedoch schnell ist, kann diese Korrelation sehr schnell zunichte gemacht werden. Der Unterschied zwischen pathogenen Varianten von E. coli und harmlosen Stämmen spiegelt sich nicht in der der ribosomalen RNA, sondern im Erwerb anderer Gene, die aggressives Wachstum bewirken. Bis zu 30 % des Genoms können bei verschiedenen E. coli-Stämmen variieren. – Das ist das 10-fache der Unterschiede zwischen uns und den Schimpansen! Das bedeutet, dass es fast unmöglich ist, herauszufinden, wie ein Bakterium vor 3 Milliarden Jahren seinen Lebensunterhalt verdient hat, da durch horizontalen Gentransfer in diesem Zeitraum alle seine Gene mehrfach ersetzt sein könnten.
Eine wichtige Implikation ist, dass bei dem Versuch, auf die Natur des letzten gemeinsamen Vorfahren zu schließen, die beste Metapher für die Geschichte des Lebens weder ein einfacher Baum noch ein Netz ist, sondern eine Reihe miteinander verbundener Wurzeln (Abb. 10). Es ist daher möglicherweise falsch, den letzten gemeinsamen Vorfahren aller heute lebenden Organismen als eine einzige Art zu betrachten. Eine genauere Beschreibung des gemeinsamen Vorfahren könnte lauten, dass es sich um eine Gemeinschaft interagierender Arten handelte, die bereitwillig ihre Gene austauschten (Woese 1998, Doolittle 2000a,b, Doolittle & Bapteste 2007, Whitfield 2004, Dagan & Martin 2006, 2007, Martin 2011, Charlebois & Doolittle 2004). Wie die Bakterien, Eukaryonten und Archaeen aus dieser verworrenen Basis hervorgegangen sein könnten, ist ein Thema, das wir nach und nach in den folgenden Kapiteln behandeln werden. Konzentrieren wir uns zuerst darauf, welche Eigenschaften LUCA definitiv hatte. Aufgrund des horizontalen Gentransfers ist das zwar nicht so einfach, dennoch konnten einige Untersuchungen einige Klarheiten gewinnen.
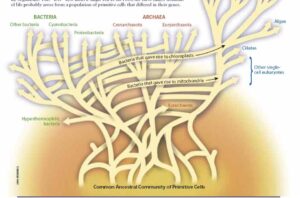
Abb. 10: Web of Life
Ein eindrucksvoller Test wurde von Bill Martin durchgeführt (Sousa et al. 2013, vgl, auch Charlebois & Doolittle 2004, Dagan & Martin 2006). Er betrachtete 48 Gene, die in allen Lebewesen konserviert sind, und erstellte für jedes dieser Gene einen Genbaum, um die Beziehung zwischen 50 Bakterien und 50 Archaeen aufzuzeigen. An den Spitzen dieses Baums ergaben alle 48 Gene genau die gleiche Beziehung zwischen allen 100 Bakterien- und Archaeenarten.

Abb. 11: Der “erstaunliche verschwindende Baum” von 48 Universalgenen für einen Satz von 100 Arten. Ein Baum, der aus einem konkatenierten Alignment von 48 Universalgenen generiert wurde, verglichen mit den zugrunde liegenden Einzelgenbäumen. Die Artenauswahl umfasst 50 Archaebakterien und 50 Eubakterien. Um die Inkonsistenz zwischen den Einzelgenbäumen und dem konkatenierten Baum abzuschätzen, wurde die Häufigkeit jedes Knotens im konkatenierten Baum mit seiner Häufigkeit in den Einzelgenbäumen verglichen. Die Transparenz der Zweige spiegelt wider, wie oft der zugehörige Knoten in den einzelnen Genbäumen vorkam. Die 48 Universalgene bestehen aus den 31 Genen, die zuvor als universell identifiziert [Charlebois & Doolittle 2004] und später in der phylogenetischen Analyse [Ciccarelli et al. 2006] verwendet wurden, nämlich (ArgRS, RNApol(a), LeuRS, metallabhängige Protease, PheRS, GTPase, SecY, Rpl1, Rpl11, Rpl13, Rpl14, Rpl15, Rpl16/L10E, Rpl18, Rpl22, Rpl3, Rpl5, Rpl6, Rps11, Rps12, Rps13, Rps15/13E, Rps17, Rps2, Rps3, Rps4, Rps5, Rps7, Rps8, Rps9, Rps, SerRS) sowie 17 weitere Gene (PRPP, AlaRS, PCNA homologue, RNApol(b), HisRS, Met-aminopeptidase, MetRS, PheRS beta subunit, ProRS, RecA, Rpl4, ThrRS, EfG, translation release factor, eIF5A, TyrRS, ValRS), die in dieser Prokaryotenprobe vorhanden sind, die keine Mitglieder mit stark reduzierten Genomen enthält. Die Taxa wurden für eine breite Probenahme ausgewählt. Zu diesem Zweck wurden Proteome von 1606 Prokaryoten aus der RefSeq-Datenbank (v03.2012) [Pruitt et al. 2012] abgerufen. Paarweise Sequenzvergleiche wurden für das ribosomale Protein L3 durchgeführt. Auf der Grundlage dieser Ergebnisse wurden alle Prokaryonten mit Hilfe eines hierarchischen Clustering-Algorithmus geclustert. Aus jedem Cluster wurden 100 Probentaxa (50 Archaeabakterien und 50 Eubakterien) ausgewählt. Bildmaterial: Sousa et al. (2013)
Auch an der Basis waren sich fast alle 48 Gene “einig”, dass der tiefste Ast im Baum des Lebens zwischen den Bakterien und den Archaeen liegt. Mit anderen Worten: LUCA war der gemeinsame Vorfahre von Bakterien und Archaeen. Aber wenn es darum geht, die Verwandtschaften innerhalb der Bakterien oder Archaeen zu ergründen, konnte kein einziger Genbaum übereinstimmen. Alle 48 Gene ergaben einen anderen Baum! Was bedeutet das? Im Wesentlichen bedeutet es, dass wir nicht feststellen können, welche Arten von Bakterien oder Archaeen die ältesten sind. Wenn ein Genbaum besagt, dass die Methanogenen die ältesten Archaeen sind, sagt der nächste Baum, dass sie es nicht sind, so dass es praktisch unmöglich ist, zu rekonstruieren, welche Eigenschaften die ältesten Zellen gehabt haben könnten.
Die Tatsache, dass alle 48 Universalgene von Bill Martin darin übereinstimmen, dass die tiefste Divergenz im Stammbaum des Lebens zwischen Bakterien und Archaeen liegt, gibt jedoch Anlass zur Hoffnung. Wenn wir herausfinden können, welche Eigenschaften alle Bakterien und Archaeen gemeinsam haben und welche sich unterscheiden, weil sie vermutlich erst später in bestimmten Gruppen entstanden sind, dann können wir ein “passendes Foto” von LUCA zusammenstellen. Je nützlicher ein Gen ist, desto wahrscheinlicher ist es, dass es schon früh in der Evolution weit verbreitet war. Um einen weit verbreiteten horizontalen Gentransfer auszuschließen, sind wir gezwungen, auf echte Universalgene zurückgreifen, die Vertreter praktisch aller Gruppe von Bakterien und Archaeen gemeinsam haben. Das minimiert zumindest die Möglichkeit, dass diese Gene durch frühen lateralen Gentransfer weitergegeben wurden. Das Problem ist nun, dass es weniger als 100 solcher Universalgene gibt, eine bemerkenswert geringe Zahl, und sie zeichnen ein sehr merkwürdiges Bild von LUCA.
Welche Eigenschaften hatte LUCA?
Für bare Münze genommen, verfügte LUCA über Proteine und DNA: Der universelle genetische Code war bereits in Betrieb, die DNA wurde in RNA-Transkripte abgelesen und dann von Ribosomen in Proteine übersetzt, jenen mächtigen molekularen Fabriken, die in allen bekannten Zellen Proteine aufbauen.
Von den Genen, die in den Genomen aller zellulären Organismen in allen Bereichen vorkommen, sind 30 ribosomale Proteine und 15 Aminoacyl-tRNA-Synthetasen – Enzyme, die Aminosäuren an ihre tRNAs anhängen (Koonin 2003, Charlebois & Doolittle 2004, Hansmann & Martin 2000). Daraus lässt sich schließen, dass LUCA über Enzyme aus Protein und eine gut entwickelte Fähigkeit zu deren Herstellung verfügte.
Die Tatsache, dass alle heute lebenden Organismen DNA verwenden, deutet darauf hin, dass der LUCA dasselbe getan hat. Eine andere Möglichkeit ist, dass der gemeinsame Vorfahre seine genetische Information in einem anderen Molekül, z. B. RNA, gespeichert hat, dass aber die Speicherung in DNA durch natürliche Selektion so stark begünstigt wurde, dass eine Umstellung von auf DNA unabhängig in mehr als einer Domäne erfolgte. Die Verwendung von DNA durch den gemeinsamen Vorfahren erscheint wahrscheinlicher als dieses Szenario einer konvergenten Evolution. Ein Anhaltspunkt ist, dass die bei der Transkription verwendeten DNA-abhängigen RNA-Polymerasen in allen drei Domänen starke Ähnlichkeiten aufweisen. Dies deutet darauf hin, dass eine DNA-abhängige RNA-Polymerase bereits beim letzten gemeinsamen Vorfahren vorhanden war. Der Besitz einer DNA-abhängigen RNA-Polymerase impliziert das Vorhandensein von DNA (Benner et al. 1989).
Andererseits sind einige Komponenten der DNA-Replikationsmaschinerie, also die Art und Weise, wie sich Genome verdoppeln, in Bakterien, Archaeen und Eukaryonten so unterschiedlich, dass wir daraus schließen können, dass sie sich unabhängig voneinander entwickelt haben (Leipe et al. 1999, Edgell & Doolittle 1997). Vielleicht speicherte der letzte gemeinsame Vorfahre seine genetische Information in der DNA, kopierte sie aber anders als moderne Organismen.
Auch Bakterien und Archaeen sind alle chemiosmotisch und treiben die ATP-Synthese mit Hilfe von Protonengradienten über die Membranen an (vgl. auch: Weiss et al. 2016, Adam et al. 2018, Camprubi et al. 2017, Ferry & House 2005, Martin & Sousa 2016, Martin & Russel 2003, 2007, Martin 2011, Martin et al. 2014, Sousa et al. 2013 Fuchs 2011, Lane et al. 2010, Ooka et al. 2018, Ducluzeau et al. 2014, Herschy et al. 2014, Huber & Wächtershäuser 1997). Das Enzym ATP-Synthase ist eine weitere außergewöhnliche molekulare Maschine, die dem Ribosom in nichts nachsteht und anscheinend ebenso alt ist wie dieses. Wie das Ribosom ist auch die ATP-Synthase über alle Lebewesen hinweg konserviert, unterscheidet sich aber in einigen Details ihrer Struktur in Bakterien und Archaeen, was darauf hindeutet, dass sie sich von einem gemeinsamen Vorfahren in LUCA entwickelt hat, ohne dass es später zu einem störenden horizontalen Gentransfer gekommen ist (Weiss et al. 2016). Die ATP-Synthase scheint also, wie Ribosomen, DNA und RNA, bereits in LUCA vorhanden gewesen zu sein. Und dann gibt es noch ein paar Teile der Kernbiochemie, wie die Aminosäurebiosynthese und Teile des Krebszyklus, die in Bakterien und Archaeen auf gemeinsamen Wegen ablaufen, was wiederum bedeutet, dass sie in LUCA vorhanden waren; aber sonst gibt es bemerkenswert wenig.
Neben Unterschieden in der DNA-Replikation, sind auch die Zellmembran ist bei beiden verschieden aufgebaut. Das Gleiche gilt für die Zellwand.
Für diese Unterschiede gibt mehrere mögliche Erklärungen (Koga et al. 1998, Lombard et al. 2012).
So könnte LUCA von allem zwei Kopien besessen haben und eine der Kopien ging jeweils bei Bakterien und Archeen verloren. Ganz auszuschließen ist das nicht, denn so ist z. B. bekannt, dass eine Mischung aus bakteriellen und archaeischen Lipiden stabile Membranen bilden können. Vielleicht besaß LUCA beide Arten von Lipiden, und ihre Nachkommen spezialisierten sich später, indem sie das eine oder das andere verloren. Das könnte für einige Merkmale zutreffen, ist aber nicht auf alle verallgemeinerbar.
Die nächste Möglichkeit ist, dass LUCA ein ganz normales Bakterium war, mit der für sie typischen Zellmembran und DNA-Replikation. Zu einem späteren Zeitpunkt spezialisierten sich dann einige Populationen an extreme Lebensbedingungen und entwickelten sich zu den Archeen mit den benannten Eigenschaften. Es bliebe aber die Frage offen, warum dann die Prozesse der Proteinsynthese sich ähneln, aber die Replikation so unterschiedlich verläuft.
Damit bleibt uns nur noch die letzte, unverschämte Möglichkeit. Das scheinbare Paradoxon ist gar kein Paradoxon: LUCA war tatsächlich chemiosmotisch, mit einer ATP-Synthase, hatte aber weder eine moderne Membran noch einen der großen Atmungskomplexe, die moderne Zellen zum Pumpen von Protonen verwenden. LUCA verfügte tatsächlich über DNA und den universellen genetischen Code, Transkription, Translation und Ribosomen, aber hatte keine moderne Methode der DNA-Replikation entwickelt. Diese seltsame Phantomzelle macht im offenen Ozean keinen Sinn, aber in der Umgebung der alkalischen hydrothermalen Schlote.
Der Schlüssel liegt darin, wie Bakterien und Archaeen in diesen Schloten leben – zumindest einige von ihnen. Und es gibt tatsächlich einen Stoffwechselprozess, welcher als Acetyl-CoA-Weg bezeichnet wird, der eine unheimliche Ähnlichkeit mit der Geochemie der Schlote hat (Russel & Martin 2004, Nitschke & Russel 2013).
Diesen werden wir im nächsten Beitrag näher kennenlernen.
Literatur
Adam, P. S., Borrel, G., Gribaldo, S. (2018): Evolutionary history of carbon monoxide dehydrogenase/acetyl-CoA synthase, one of the oldest enzymatic complexes. Proc. Natl. Acad. Sci. USA 115, E1166–E1173
Abby, S. S. et al. (2012): Lateral gene transfer as a support for the tree of life. Proc. Natl. Acad. Sci. USA 109, 4962–4967
Baldauf, S. L., Palmer, J. D., Doolittle, W. F. (1996): The root of the universal tree and the origin of eukaryotes based on elongation factor phylogeny. Proceedings of the National Academy of Sciences, USA 93:7749–7754.
Benner, S. A. et al. (1989): Modern metabolism as a palimpsest of the RNA world. Proceedings of the National Academy of Sciences, USA 86: 7054–7058.
Bernier, C. R. et al: (2018): Translation: the universal structural core of life. Mol. Biol. Evol. 35(8), 2065–2076
Brown, J. R., Doolittle, W. F. (1995): Root of the universal tree of life based on ancient aminoacyl-tRNA synthetase gene duplications. Proceedings of the National Academy of Sciences, USA 92: 2441–2445.
Brown, J. R., Doolittle, W. F. (1997): Archaea and the prokaryote-to-eukaryote transition. Microbiology and Molecular Biology Reviews 61: 456–502.
Bult, C. J. et al. (1996): Complete genome sequence of the methanogenic archaeon, Methanococcus jannaschii. Science 273: 1058–1073.
Camprubí, E., de Leeuw, J.W., House, C.H. et al. (2019): The Emergence of Life. Space Sci Rev 215, 56 https://doi.org/10.1007/s11214-019-0624-8
Camprubi, E. et al. (2017): Iron catalysis at the origin of life. IUBMB Life 69(6), 373–381
Charlebois, R. L., Doolittle W. F. (2004): Computing prokaryotic gene ubiquity: rescuing the core from extinction. Genome Res. 14, 2469–2477.doi:10.1101/gr.3024704
Ciccarelli, F. D., et al. (2006): Toward automatic reconstruction of a highly resolved tree of life. Science 311, 1283–1287.doi:10.1126/science.1123061
Dagan T., Martin W. (2006): The tree of one percent. Genome Biology 7: 118.
Dagan T., Martin W. (2007): Ancestral genome sizes specify the minimum rate of lateral gene transfer during prokaryote evolution. Proceedings National Academy Sciences USA 104: 870–75.
Dagan, T. et al. (2010): Genome networks root the tree of life between prokaryotic domains. Genome Biology and Evolution 2: 379–392.
Doolittle, W. F. (2000a): Searching for the common ancestor. Research in Microbiology 151: 85–89.
Doolittle W. F. (2000b): Uprooting the tree of life. Sci Am. 282(2):90-5.
Doolittle, W. F., Bapteste, E. (2007): Pattern pluralism and the Tree of Life hypothesis. Proc. Natl. Acad. Sci. (USA) 104:2043-2049.
Doolittle, W. F., Brown; J. R. (1994): Tempo, mode, the progenote, and the universal root. Proceedings of the National Academy of Sciences, USA 91: 6721–6728.
Ducluzeau, A. L. et al. (2014): Free energy conversion in the LUCA: quo vadis? Biochim. Biophys. Acta (BBA)-Bioenerg. 1837(7), 982–988
Edgell, D. R., Doolittle, W. F. (1997): Archaea and the origin(s) of DNA replication proteins. Cell 89: 995–98.
Ferry, J. G., House, C. H. (2005): The stepwise evolution of early life driven by energy conservation. Mol. Biol. Evol. 23(6), 1286–1292 (2005)
Fitz Gibbon, S. T., House, C. H. (1999): Whole genome-based phylogenetic analysis of free-living microorganisms. Nucleic Acids Res. 27(21), 4218–4222
Fournier, G. P., Gogarten, J. O. (2010): Rooting the ribosomal tree of life. Molecular Biology and Evolution 27: 1792–1801.
Fox, G. E. (2010): Origin and evolution of the ribosome. Cold Spring Harb. Perspect. Biol. 2(9), A003483
Fox, G. E., Magrum, L. J. et al. 1977. Classification of methanogenic bacteria by 16S ribosomal RNA characterization. Proceedings of the National Academy of Sciences, USA 74: 4537–4541.
Fuchs, G. (2011): Alternative pathways of carbon dioxide fixation: insights into the early evolution of life? Annu. Rev. Microbiol. 65, 631–658
Gogarten, J. P. et al. (1989): Evolution of the vacuolar H+@ATPase: Implications for the origin of eukaryotes. Proceedings of the National Academy of Sciences, USA 86: 6661–6665.
Gogarten, J. P. et al. (2002): Prokaryotic evolution in light of gene transfer. Mol. Biol. Evol. 19(12), 2226–22238
Hansmann S., Martin, W. (2000): Phylogeny of 33 ribosomal and six other proteins encoded in an ancient gene cluster that is conserved across prokaryotic genomes. Int. J. Syst. Evol. Microbiol. 50, 1655–1663.doi:10.1099/00207713-50-4-1655
Herschy B., Whicher A., Camprubi E., Watson C., Dartnell L., Ward J., Evans J.R.G., Lane N.(2014): An origin-of-life reactor to simulate alkaline hydrothermal vents. Journal of Molecular Evolution 79: 213–27.
House, C. H. (2009): The tree of life viewed through the contents of genomes. Methods Mol. Biol. 532, 141–161
Huber C., Wächtershäuser G. (1997): Activated acetic acid by carbon fixation on (Fe,Ni)S under primordial conditions. Science 276: 245–47.
Iwabe, N. et al. (1989): Evolutionary relationship of archaebacteria, eubacteria, and eukaryotes inferred from phylogenetic trees of duplicated genes. Proceedings of the National Academy of Sciences, USA 86: 9355–9359.
Koga, Y., Kyuragi, T., Nishihara, M., Sone, N. (1998): Did archaeal and bacterial cells arise independently from noncellular precursors? A hypothesis stating that the advent of membrane phospholipid with enantiomeric glycerophosphate backbones caused the separation of the two lines of descent. Journal of Molecular Evolution 46: 54–63
Koonin, E. V. (2003): Comparative genomics, minimal gene-sets, and the last universal common ancestor. Nature Reviews Microbiology 1: 127–136.
Koonin, E. V. (2016): Horizontal gene transfer: essentiality and evolvability in prokaryotes, and roles in evolutionary transitions. F1000 Res 5, 1805
Lane, N., Allen, J. F., Martin, W. (2010): How did LUCA make a living? Chemiosmosis in the origin of life. BioEssays 32, 271–280
Lanier, K. A. et al. (2016): Imprint of ancient evolution on rRNA folding. Biochemistry 55(33), 4603–4613
Leipe, D. D. et al. (1999): Did DNA replication evolve twice independently? Nucleic Acids Research 27: 3389–3401.
Lombard, J., López-García, P., Moreira, D. (2012): The early evolution of lipid membranes and the three domains of life. Nature Reviews in Microbiology 10: 507–15.
Madigan, M. T., Marrs, B. L. (1997): Extremophiles. Scientific American 276(4): 82–87.
Margulis, L. (1974): Five-kingdom classification and the origin and evolution of cells, in Evolutionary Biology, vol. 7, ed. by T. Dobzhansky, M.K. Hecht, W.C. Steere (Springer, Boston), pp. 45–78
Margulis, L. (1996): Archaeal-eubacterial mergers in the origin of Eukarya: Phylogenetic classification of life. Proceedings of the National Academy of Sciences, USA 93: 1071–1076.
Martin W. (2011): Early evolution without a tree of life. Biology Direct 6: 36.
Martin W., Russell, M. (2003): On the origins of cells: a hypothesis for the evolutionary transitions from abiotic geochemistry to chemoautotrophic prokaryotes, and from prokaryotes to nucleated cells. Philosophical Transactions Royal Society B 358 59–83.
Martin W., Russell, M. (2007): On the origin of biochemistry at an alkaline hydrothermal vent. Philosophical Transactions of the Royal Society B 367: 1887–1925.
Martin, W., Sousa, F. L. (2016): Early microbial evolution: the age of anaerobes. Cold Spring Harb Perspect Biol. 8, A018127
Martin, W., Sousa, F.L., Lane, N. (2014): Energy at life’s origin. Science 344: 1092–93.
Monro, R. E. (1967): Catalysis of peptide bond formation by 50S ribosomal subunits from Escherichia coli. J. Mol. Biol. 26(1), 147–151
Nitschke, W., Russell, M. (2013): Beating the acetyl coenzyme A-pathway to the origin of lifePhil. Trans. R. Soc. B3682012025820120258
Olsen, G. J., Woese, C. R. (1996): Lessons from an Archaeal genome: What are we learning from Methanococcus jannaschii? Trends in Genetics 12: 377–379
Ooka, H., McGlynn, S. E., Nakamura, R. (2018): Electrochemistry at deep-sea hydrothermal vents: utilization of the thermodynamic driving force towards the autotrophic origin of life. ChemElectroChem 6(5), 1316–1323
Petrov, A. S. et al. (2014a): Secondary structures of rRNAs from all three domains of life. PLoS ONE 9(20), e88222
Petrov, A. S. et al. (2014b): Evolution ft he ribosome at atomic resolution. Proc. Natl. Acad. Sci. USA 111(28), 10251–10256
Petrov, A. S. et al. (2015): History ft he ribosome and the origin of translation. Proc. Natl. Acad. Sci. USA 112(50), 15396–15401
Pruitt, K. D. et al. (2012): NCBI reference sequences (RefSeq): current status, new features and genome annotation policy. Nucleic Acids Res. 40, D130–D135.doi:10.1093/nar/gkr1079
Puigbò, P. et al. (2009): Search for a ‘Tree of Life’ in the thicket of the phylogenetic forest. J. Biol. 8, 59
Puigbò, P. et al. (2010): The tree and net components of prokaryote evolution. Genome Biol. Evol. 2, 745–756
Russell, M., Martin, W. (2004): The rocky roots of the acetyl-CoA pathway. Trends in Biochemical Sciences 29: 358–63 (2004).
Sapp, J., Fox, G. E. (2013): The singular quest for a universal tree of life. Microbiol. Mol. Biol. Rev. 77(4), 541–550
Smith, T. F. et al. (2008): The origin and evolution of the ribosome. Biol. Direct 3, 16
Snel, B. et al. (1999): Genome phylogeny based on gene content. Nat. Genet. 21(1), 108
Sousa F. L. et al. (2013): Early bioenergetic evolution. Philosophical Transactions of the Royal Society B 368: 20130088.
Weiss, M. C. (2016): The physiology and habitat of the last universal common ancestor. Nat. Microbiol. 25/1(9), 16116
Whitfield, J. (2004): Born in a watery commune. Nature 427: 674–676.
Whittaker, R. H. (1969): New concepts of kingdoms of organisms. Science 163: 150–160.
Woese, C. R. (1968): The fundamental nature of the genetic code: prebiotic interactions between polynucleotides and polyamino acids and their derivatives. Proc. Natl. Acad. Sci. USA 59(1), 110–117
Woese, C. R. (1991): The use of ribosomal RNA in reconstructing evolutionary relationships among bacteria. In Evolution at the Molecular Level, ed. R. K. Selander, A. G. Clark, and T. S. Whittam. Sunderland, MA: Sinauer, 1–24.
Woese, C. R. (1998): The universal ancestor. Proceedings of the National Academy of Sciences, USA 95: 6854–6859.
Woese, C. R. (2000): Interpreting the universal phylogenetic tree. Proceedings of the National Academy of Sciences, USA 97: 8392–8396.
Woese, C. R. (2002): On the evolution of cells. Proceedings of the National Academy of Sciences, USA 99: 8742–8747.
Woese, C. R., Fox, G. E. (1977a): The concept of cellular evolution. J. Mol. Evol. 10(1), 1–6
Woese, C. R., Fox, G. E. (1977b): Phylogenetic structure of the prokaryotic domain: The primary kingdoms. Proceedings of the National Academy of Sciences, USA 74: 5088–5090.
Woese, C. R., Kandler, O., Wheelis, M. L. (1990): Towards a natural system of organisms: Proposal for the domains Archaea, Bacteria, and Eucarya. Proceedings of the National Academy of Sciences, USA 87: 4576–4579.
Zhaxybayeva, O., P. et al. (2005): Ancient gene duplications and the root(s) of the tree of life. Protoplasma 227: 53–64.
Zuckerkandl, E., Pauling, L. (1965): Molecules as documents of evolutionary history. Journal of Theoretical Biology 8: 357–366.
[1] https://internet-evoluzzer.de/die-bedeutung-und-urspruenge-des-stoffwechsels/ und die dortigen Quellen