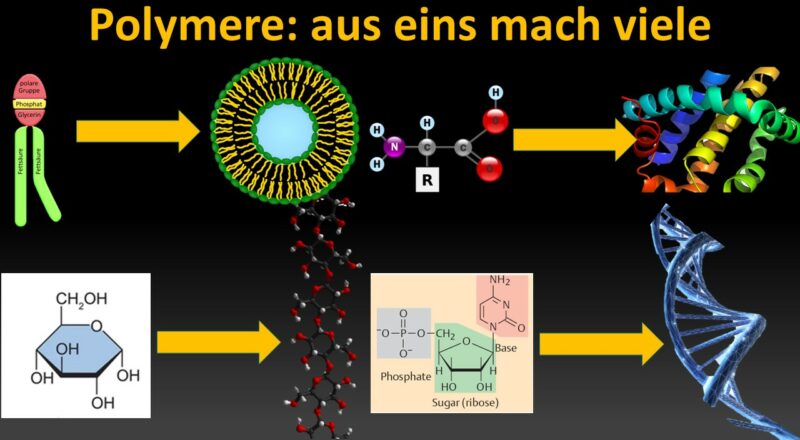
Einleitung: Grundlagen der Polymerisation
In den letzten beiden Episoden haben wir das Experiment von Miller und weitere Ursuppenexperimente gezeigt, die bestätigen, dass unter abiotischen Bedingungen die Grundbausteine des Lebens: Aminosäuren, Nukleinsäuren, Zucker und Lipide auf natürliche Weise entstehen können. Diese Grundbausteine bezeichnen wir als Monomere. Man konnte belegen, dass sich praktisch alle relevanten Biomoleküle abiotisch, auf der Grundlage physiko-chemischer Gesetze bilden können.
Der nächste Schritt ist es, dass sich die einzelnen Monomere zu komplexeren Makro-Molekülen vereinigen. Man spricht hier von Polymerisation.
Aminosäuren verbinden sich zu Proteinen, Nukleotide zu RNA und DNA. Monosacharide verbinden sich zu komplexeren Kohlenhydraten. Lipide vereinen sich zu komplexeren Lipidschichten, die wir z. B. in Zellmembranen haben (Abb. 1).
Text als pdf
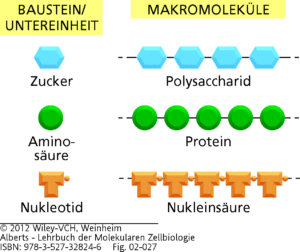
Abb. 1: Monomerke und die daraus entstehenden Makromoleküle
Formal sollte die Synthese von Polymeren einfach sein: Zwei Monomere können miteinander verknüpft werden, indem ein Wasserstoffatom (H) aus einem Monomer und eine Hydroxylgruppe (-OH) aus einem anderen Monomer eliminiert werden und dann das entstehende Wassermolekül freigesetzt wird (Abb. 2).
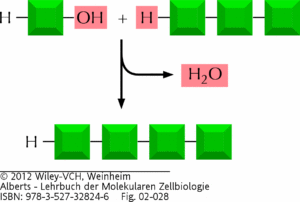
Abb. 2: Hydrolyse
Die Ursuppentheorie hat hier aber ein ernst zu nehmendes Problem, welches zu berücksichtigen ist. Denn die Bildung langkettiger Biomoleküle ist im Urozean nicht möglich bzw. sehr aufwendig.
Durch große Mengen Wasser wird die Entstehung langer Polymere verhindert; bereits kleine Ketten spalten sich wieder auf. Außerdem ist Energie nötig, um die Monomere zu Polymeren zu verbinden, ist energetisch also ungünstig. Woher kam diese Energie?
Ein weiteres Problem besteht in der geringen Stabilität wässriger Zucker- und Aminosäurelösungen; die Produkte zerfallen in der Regel nach kurzer Zeit.
Wie konnten also Polymere auf natürlichem Wege entstehen? Hier können Mineraloberflächen eine wichtige Rolle spielen (Bernal 1949).
Bedeutung von Mineraloberflächen für die Polymerisation
Für ein längeres, komplexeres Polymer braucht man ein “Gerüst” oder eine “Schablone”, ein anderes Material, das alle organischen Moleküle anzieht und in die gleiche Richtung ausrichtet. Sobald alle Moleküle in die gleiche Richtung ausgerichtet und dicht gepackt sind, ist es ein Leichtes, sie miteinander zu verbinden und komplexe organische Polymere herzustellen.
Während die Gesetze der Thermodynamik in freier Lösung die Spaltungsreaktion begünstigen, fördern sie auf Oberflächen die Kettenbildung. Auch die Stabilität oberflächengebundener Substanzen (Zucker, Nukleinbasen etc.) ist weitaus größer als in freier Lösung, und eine Reihe von Mineralen haben eine katalytische Wirkung, das heißt sie können selektiv ganz bestimmte Reaktionen beschleunigen oder überhaupt erst ermöglichen.
Leslie Orgel, der diese Vorstellung als “Leben auf den Steinen” bezeichnete, entwickelte die Theorie für ihre Entstehung (Gibbs et al. 1980; Ferris et al. 1996; Orgel 1998). Orgel stellte fest, dass bei der Bindung eines Polymers an eine Mineraloberfläche die freie Bindungsenergie linear ansteigt, wenn der Polymerkette ein Monomer nach dem anderen hinzugefügt wird.
Als Energiequelle für die Polymerisation eignen sich folgende Reaktionen, die auf der frühen Erde abgelaufen sein könnten:
a: (FeS2) + H2→ (FeS) + (H2S) + E,
b: 3H2 + CO → CH4 + H2O + E,
c: 4H2 + CO2→ CH4 + 2H2O + E,
b und c sind sogenannte Sabatier-Reaktionen, bei der Kohlenstoffdioxid und Wasserstoff in Methan und Wasser umgewandelt werden. Die Reaktionen sind exotherm, geben also Wärme ab und brauchen Mineraloberflächen als Katalysator.
Welche Minerale eignen sich besonders für die Bildung von Polymeren?
Zeolithe
Die bekanntesten Kandidaten sind die als Zeolithe bekannten Minerale (Abb. 3), komplexe Silikatminerale, die typischerweise durch den Zerfall von vulkanischem Glas in heißen Gasblasen in der Lava entstehen. Zeolithe haben eine komplexe, sich wiederholende Mineralstruktur, die ihnen hilft, organische Reaktionen zu katalysieren und diese Reaktionen zu beschleunigen. In der Tat werden sie in der Industrie genau zu diesem Zweck eingesetzt, insbesondere bei der Erdölraffination, bei der Filtration und bei der Absorption von Chemikalien, oder z. B. bei Katzenstreu. Ihre poröse Struktur erlaubt, dass sie als Molekularsiebe fungieren können und große Moleküle vom Wasser trennen, Man bräuchte nur ein paar Zeolithe in einer Ursuppe, und schon könnten die Aminosäuren zu viel komplexeren Proteinen aufgereiht werden (Wright et al. 1985, Newsam 1986).

Abb. 3: Zeolithe
Ton
Eine andere Idee stammt von Alexander Graham Cairns-Smith, der eine noch bescheidenere Vorlage als Zeolithe vorschlug: gewöhnlichen Ton (Cairns-Smith 1982, 1985). Die komplexen, offen geschichteten Strukturen von Tonmineralien (Abb. 4) sind ideal, um organische Moleküle aufzunehmen und sie entlang der Tonmineralstruktur anzuordnen. Die grundlegende Schichtstruktur von Tonmineralen wiederholt sich immer wieder, mit kleinen Unvollkommenheiten in den Kristallen, vergleichbar mit Mutationen im genetischen Code. Cairns-Smith behauptet sogar, dass das Leben als Tonmineralien begann, die sich während des Kristallisationsprozesses immer wieder selbst kopierten. Tonminerale können wachsen, ihre Umgebung verändern und sich vermehren. Diese hochspekulativen Ideen sind natürlich auch umstritten, aber angesichts unserer Kenntnisse über die Chemie von Tonen und organischen Molekülen nicht unmöglich.
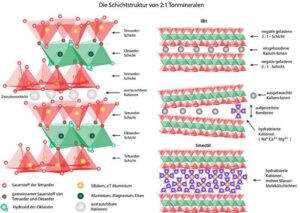
Abb. 4: Tonminerale
Pyrit
Aber es gibt noch eine weitere mögliche Vorlage, die man in Betracht ziehen kann: das Mineral Pyrit, FeS2, besser bekannt als “Katzengold” (Abb. 5). Gunter Wächtershäuser hat gezeigt, dass Pyritkristalle eine positive Ladung auf der Oberfläche haben, die die negativ geladenen Enden vieler organischer Moleküle anziehen könnte. Sobald sie angezogen, aufgereiht und dicht aneinander gepackt sind, können sich die organischen Moleküle leicht zu komplexen Polymeren verbinden. Sobald sie miteinander verbunden sind, können sie sich vom Pyrit-Muster lösen und als freie Biomoleküle schwimmen. Seine Theorie ist auch als Oberflächenmetabolismus bekannt (Wächtershäuser 1988a; 1988b; 1990; 1992; 1994; 2000). Als Grundlage der Erzeugung wichtiger chemischer Verbindungen dient Schwefelwasserstoff (H2S), der neben Kohlenmonoxid, Methan, Stickstoff und CO2 beständig aus heißen Schloten der Tiefsee ausgast. Dieser reagiert mit Eisenmonosulfid (Mackinawit, FeS) zu Pyrit. Bei dieser Reaktion wird Wasserstoff (H2) gebildet und Energie frei, die zur Erzeugung von so wichtigen Grundstoffen wie Blausäure oder Ammoniak genutzt werden kann (Dörr et al. 2003). Auf der Pyritoberfläche gebunden könnten daraus wiederum Aminosäuren und diverse Zucker entstanden sein, die in der Lage sind, lange Polymere zu erzeugen, aus denen sich stufenweise Isoprenoide, Hüllmembranen und einfache Stoffwechselprozesse gebildet haben könnten.
Die Stärke dieser Vermutung liegt darin, dass sie zu einer anderen erstaunlichen wissenschaftlichen Entdeckung passt: den “schwarzen Rauchern” auf dem Meeresgrund. Diese werden wir in einem späteren Beitrag näher kennenlernen.
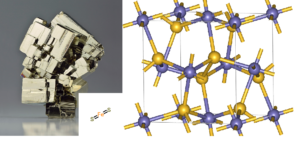
Abb. 5: Pyrit
Metallionen
Wie Minerale könnten auch Metallionen eine wichtige katalytische Rolle bei der Entstehung des Lebens auf der Erde gespielt haben. Es ist bekannt, dass verschiedene Metallionen die Synthese von kurzen RNA-Oligomeren aus aktivierten Monomeren katalysieren, aber das Uranyl-Ion (UO22+) ist eines der wenigen Metallionen (Abb. 6), von denen bekannt ist, dass sie die Bildung von Oligomeren mit zehn oder mehr Monomereinheiten katalysieren. Es bildeten sich Oligomere aus Adenin, Uracil und Cytosin, wobei die maximale Kettenlänge 16, 12 bzw. 10 betrug (Sawai et al. 1992).
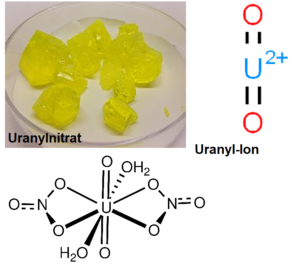
Abb. 6 Uranyl
Metallionen wie Calcium (C2+) können negativ geladene organische Stoffe an der Oberfläche binden. Ein gutes Beispiel ist das Mineral Hydroxylapatit, Ca5(PO4)3(OH), in dem Ca2+ sowohl an Asparaginsäure als auch an Glutaminsäure bindet (Hill et al. 1998). Ein anderes Mineral, das gewöhnliche Tonmineral Illit, bindet ebenfalls negativ geladene Aminosäuren (Liu und Orgel 1998).
Bildung von Polypeptiden
Wenn Aminosäuren eine Verbindung eingehen, spricht man von Peptidbindung. Proteine sind Polypeptide, weil sie Polymere mehrerer Aminosäuren sind. Bei einer Peptidbindung geht die Carboxylgruppe von Aminosäure 1 eine Verbindung mit der Aminogruppe von Aminosäure 2 ein, dabei spaltet sich ein Wassermolekül ab und die Amino- und Carboxylgruppe verbinden sich miteinander (Abb. 7).
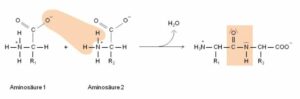
Abb. 7: Peptidbindung
Frühe Polymerisationsexperimente führte Sidney Fox 1958 durch (Fox & Harada, 1958). Er erhitze Glutaminsäure in einem Ölbad und später mit Asparaginsäure und ein Aminosäuregemisch zugesetzt und unter einer CO2-Schicht erhitzt. Die Lösung wurde dann abgekühlt und später mit Wasser und Ethanol versetzt und filtriert. Der beim Filtrieren zurückbleibende Feststoff wurde in Zellophan-Dialyseschläuche gefüllt und in ein Wasserbad gelegt. Nach einigen Tagen wurde das Innere der Röhrchen untersucht und die Forscher fanden Polypeptidketten, die sie Proteinoide nannten (Abb. 8).
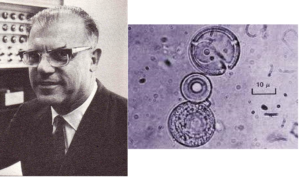
Abb. 8: Sidney Fox und seine Proteinoide
Man erhielt aus verdünnten Aminosäurelösungen, die mit Ton gekocht wurden, Polypeptide, die bereits aus bis zu einem Dutzend Aminosäuren aufgebaut waren (Kämpfe 1992). Dabei konnte eine Arbeitsgruppe Polypeptide mit einer Länge von bis zu 56 Monomeren erzeugen (Paecht-Horowitz & Eirich 1988).
Für die Proteinsynthese in Lebewesen werden Enzyme benötigt, Bildung von Peptiden energetisch ungünstig ist. Wie können dann abiotische Bedingungen zur Bildung von Peptiden führen, wenn Enzyme, die ja selbst Proteine sind, nicht vorhanden sind? Eine Forschergruppe um Dylon Holden (Holden et al. 2022) nahm sich dieser Frage an. Hier spielen Grenzflächen an mikrometergroßen Flüssigkeitströpfchen eine Rolle, deren Struktur die Bildung von Peptiden, bestehend aus bis zu 6 Monomereinheiten erleichtert (Abb. 9).
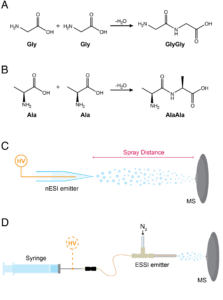
Abb. 9: Reaktionen, Versuchsaufbauten und Auswirkungen von Sprühparametern im Zusammenhang mit der spontanen Erzeugung von Dipeptiden aus freien Aminosäuren in wässrigen Mikrotropfen. A und B zeigen Kondensationsreaktionen, bei denen GlyGly und AlaAla aus freiem Gly bzw. Ala entstehen. C und D zeigen die Versuchsanordnungen einschließlich Hochspannung (HV), die zur Erzeugung von Mikrotröpfchen in nESI (C) und ESSI (D) verwendet wurden.
Eine Arbeitsgruppe aus dem Jahr 2007 (Chen & Yang 2007) berichtet, dass die Oxidation von Schwefel (IV) eine Peptidbindung einleiten kann. Dies unterstützt die Hypothese, dass frühes Leben in einer vulkanischen Umgebung entstanden sein könnte (Furnes et al. 2004, Rasmussen 2000). Auch heute noch beziehen viele Mikroorganismen die Energie für ihr Wachstum aus der Oxidation von Schwefelverbindungen zu Schwefelsäure (Suzuki 1999).
Eine weitere Forschergruppe (Rodriguez-Garcia et al. 2015) führten Experimente zur Bildung von Oligopeptidketten mit einer Ausbeute von etwa 50% durch. Zur Untersuchung dieses Prozesses wurde ein digitales, rekursives Reaktorsystem entwickelt, das diese Reaktionen unter Kontrolle von Parametern wie Temperatur, Anzahl der Zyklen, Zyklusdauer, anfänglicher Monomerkonzentration und anfänglichem pH-Wert durchführt. Es wurden Glycin-Oligopeptide mit einer Länge von bis zu 20 Aminosäuren gebildet. Nachdem die Bildung von Peptiden, die nur aus Glycin bestanden, nachgewiesen wurde, konnten auch Bindungen von Glycin mit acht anderen Aminosäuren (Alanin, Asparaginsäure, Glutaminsäure, Histidin, Lysin, Prolin, Threonin und Valin) erreicht werden.
Die Abfolge der Aminosäuren bezeichnet man als Primärstruktur der Proteine. Doch können Proteine komplexere Strukturen durch Wasserstoffbrückenbindungen eingehen. Eine Wasserstoffbrückenbindung ist eine anziehende Wechselwirkung. Sie entsteht zwischen einem gebundenen Wasserstoffatom eines Moleküls und einem stark elektronegativen Atom eines zweiten Moleküls. Das elektronegative Atom ist häufig ein Stickstoff (N) oder Sauerstoff (O) und trägt mindestens ein freies Elektronenpaar. Weil in den funktionellen Gruppen der Aminosäuren gerade diese elektronegativen Atome vorkommen, gehen benachbarte Bereiche eines Proteins solche Wasserstoffbrückenbindungen ein und bilden eine Sekundärstruktur aus. Die bekanntesten Sekundärstrukturen sind die a-Helix und das ß-Faltblatt. Neben der Sekundärstruktur gibt es auch die Tertiärstruktur, welche durch Wechselwirkungen in weiter entfernt liegenden Bereichen eines Proteins entstehen. Außerdem gibt es die Quartärstruktur, welches Proteine sind, die aus mehreren Untereinheiten bestehen (Abb. 10).
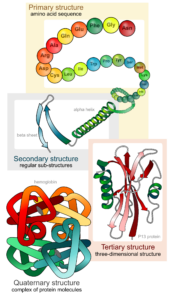
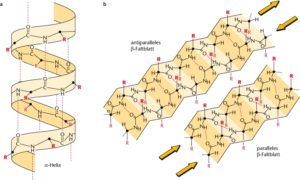
Abb. 10: Proteine
Das Beta-Faltblatt als Sekundärstruktur bildet sich spontan aus Polypeptidketten, die aus Aminosäuren mit abwechselnd hydrophoben und hydrophilen Seitenketten bestehen, wie beim Leucin-Lysin-Polymer (Brack & Orgel 1975, Brack 1993, 1994).
Experimentelle Studien stützen den Vorschlag, dass Beta-Faltblätter bei der Entstehung des Lebens eine wichtige Rolle gespielt haben, weil sie zehnmal widerstandsfähiger gegen einen Abbau sind als Peptidketten, die aus zufälligen Windungen bestehen oder eine alpha-Helix aufweisen. Damit hätten sie eine längere Lebensdauer und wären leichter für den Einbau in die ersten Lebensformen verfügbar gewesen. In diesem Fall hätte diese Vorliebe zu einer präbiotischen Selektion von Aminosäuren geführt, die in der Lage sind, Beta-Faltblätter zu bilden. Eine solche Selektivität deutet außerdem darauf hin, dass die frühesten biologischen Systeme eine begrenzte Anzahl verschiedener Aminosäuren enthielten (Brack 1987).
Es ist bekannt, dass das vulkanische Gas Carbonylsulfid (COS) die Kondensation von Aminosäuren unter wässrigen Bedingungen katalysiert (Leman et al. 2004, Abb. 11). Auch andere vulkanische Gase wie Kohlenstoffdisulfid unterstützen die Polymerisation von Aminosäuren (Leman et al. 2015), aber die berichteten Peptide sind zu kurz, um eine regelmäßige Tertiärstruktur zu bilden.
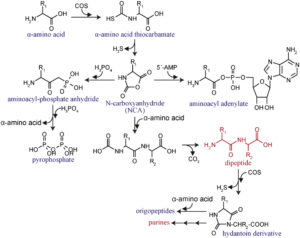
Abb. 11: COS-vermittelte chemische Evolution von Peptiden und Nukleotiden.
Eine Arbeit von Jason Greenwald (Greenwald et al. 2016) konnte demonstrieren, dass vier Aminosäuren, Alanin, Valin, Glycin und Asparat, von denen man annimmt, dass sie zu den häufigsten auf der präbiotischen Erde gehörten, zu Peptiden polymerisieren und sich anschließend zu geordneten Proteinaggregaten zusammensetzen können. Diese Proteinaggregate sind sogenannte Amyloide, die durch eine fibrilläre Morphologie und eine Beta-Faltblatt-Sekundärstruktur gekennzeichnet sind. Als Katalysator wurde das Vulkangas Carbonylsulfid, welches wahrscheinlich auch schon vor Milliarden von Jahren in der Atmosphäre existierte, verwendet (Abb. 12).
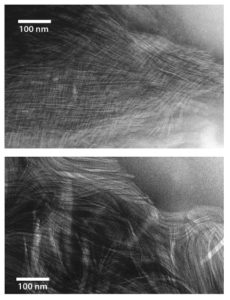
Abb. 12: Amyloide aus Alanin (oben) und Valin (unten) unterm Elektronenmikroskop nach Greenwald et al. (2016).
Im Laborversuch bauten sich die Aminosäuremoleküle mit Hilfe des Carbonylsulfids spontan zu kurzen Ketten (Peptiden) aus 5 bis 14 Monomeren auf. Diese Ketten wiederum ordneten sich parallel zu Beta-Faltblättern an. Im Experiment nahmen diese Blattstrukturen die Form von Fasern an und bestanden typischerweise aus Tausenden von aneinandergereihten Peptidketten, die die Wissenschaftler mit Hilfe eines Elektronenmikroskops identifizieren konnten.
Um sicherzustellen, dass sich die Aminosäuremoleküle zu ausreichend langen Peptidketten formten, tropften die Wissenschaftler in einem mehrstündigen Verfahren mit Carbonylsulfid aktivierte Aminosäuremoleküle langsam in ein Reagenzglas. Es ist denkbar, dass in der Urgeschichte der Erde ein ebenso langsamer, möglicherweise mehrere Jahre dauernder Prozess mit einem stetigen Fluss neuer chemischer Verbindungen stattgefunden hat.
Selbst einfache Amyloide sind in der Lage bestimmte chemische Funktionen auszuführen (Friedman et al. 2015) und sind stabiler als RNA, weswegen die Forscher davon ausgehen, dass diese eher die Vorläufer von Lebewesen seien als die komplexere und schwer zu bildende RNA. Diese Behauptung steht zu der sog. RNA-Welt, die postuliert, dass die Vorläufer der Lebewesen aus RNA-Molekülen bestanden entgegen. Es ist aber noch nicht klar, ob Amyloide in der Lage sind, sich selbst zu replizieren (also zu vermehren), wie es bei der RNA der Fall ist. Die Forscher arbeiten noch dran.
Auch komplexere Strukturen von Aminosäuren lassen sich unter präbiotischen Bedingungen bilden.
Ein Forscherteam der Universität Triest (Italien) hat ein selbstorganisierendes, selbstfaltendes Molekül entworfen und damit begonnen, die Konstruktionsprinzipien hinter der Proteinfaltung aufzudecken. Ihre Peptid-Nukleobase braucht nur eine Salzwasserlösung, um in 95 %iger Ausbeute einen sog. Makrozyklus aus 15 Monomeren zu bilden (Abb. 13). Der 75-atomige Ring faltet sich zu einer komplizierten, verdrehten Form, mit einem hydrophoben Kern und einer hydrophilen Oberfläche – genau wie ein Protein. Disulfidbrücken, Wasserstoffbrücken und aromatische Ring-Stapelwechselwirkungen halten das Peptid zusammen und verleihen dem Ring seine gewundene Sekundärstruktur. Der Makrozyklus hat auch eine Tertiärstruktur, die aus fünf Stapeln von drei aromatischen Ringen besteht, die sich kachelartig anordnen. Die Forscher untersuchen nun, wie die Veränderung der Monomerstruktur das Selbstorganisations- und Faltungsverhalten des Moleküls verändert, um beispielsweise größere oder kleinere Ringe zu bilden (Liu et al, 2019).
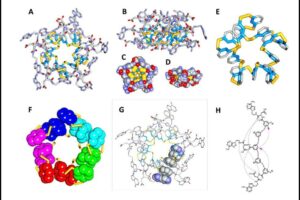
Abb. 13: Röntgenkristallstruktur des 15mers. (a) Draufsicht auf den zentralen Hohlraum des Makrozyklus. (b) Seitenansicht. (c) Draufsicht und (d) Seitenansicht des 15mers in raumfüllender Darstellung. (e) Der Ring aus Disulfidbindungen, der die Phenylringe verbindet. (f) Kernteil des Foldamers, der fünf Stapel von drei durch Disulfidbindungen verbundenen Phenylringen zeigt. (g) Draufsicht auf das 15mer mit einem Stapel von drei Kernphenylringen und zwei Adeninen am oberen und unteren Ende des Stapels. (h) Intermolekulare Wasserstoffbrückenbindungen zwischen den drei Bausteinen, die den Stapel aus drei Phenylringen bilden.
Polymerisation von Kohlenhydraten und Nukleinsäuren
Der Zucker Ribose gehört als Bestandteil der RNA zu den wichtigsten Zuckermolekülen bei der Entstehung der Bausteine des Lebens.
Chemische Analysen des Kohlenhydrat-Prämetabolismus zeigen, dass Formaldehyd und Glycolaldehyd mit Borat-, Molybdat- und Kalziummineralen die Bildung von Tetrosen (C4H8O4), Heptosen (C7H14O7) und Pentosen (C5H10O5), einschließlich der Ribose, steuern (Kim et al. 2011). Diese Ergebnisse zeigen, dass sich Pentosen mit ziemlicher Sicherheit als stabile Boratkomplexe auf der Oberfläche einer frühen Erde unter einer feuchten CO2-Atmosphäre gebildet haben könnten (Abb. 14).
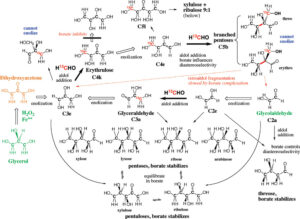
Abb. 14: Bildung verschiedener Kohlenhydrate mithilfe von Boratminerale nach Kim et al. (2011)
Dass Mineraloberflächen eine wichtige Rolle bei der Stabilisierung und Polymerisierung von RNA zeigen eine Reihe an Experimenten:
Eine der erfolgreichsten Anwendungen zur Polymerisation von RNA war die Bildung von RNA-Oligomeren mit 6 bis 14 Monomereinheiten. Die Polymere entstanden durch Bindung auf Montmorillonit-Ton. In diesen Polymeren war die aktivierende Gruppe ein Imidazol, das an eine Phosphatgruppe gebunden war (Ferris & Ertem 1992, 1993, Ferris 1993, 2006, Ferris et al. 2004, Ertem & Ferris 1996, Ding et al. 1996, Kawamura & Ferris 1999, siehe auch Kaddour et al. 2018). Es bilden sich Oligomere, unabhängig davon, ob es sich bei der beteiligten Nukleotidbase um Adenin, Cytosin, Guanin oder Uracil handelt (Abb. 15).
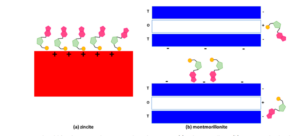
Abb. 15: Vorgeschlagenes Modell für die Adsorptions-Polymerisations-Beziehung auf Zinkit (a) und Montmorillonit (b). Auf Zinkit ist der Phosphatanteil fest an die Mineraloberfläche gebunden und steht daher für die Polymerisation nicht zur Verfügung. Auf Montmorillonit erfolgt die Adsorption an Montmorillonit über die Nukleobasen oder die funktionelle Amingruppe des Monomers, so dass die Phosphateinheit für ein ankommendes (oder benachbart adsorbiertes) Monomer zur Polymerisation zugänglich ist. Die schematische Darstellung soll nur die Assoziation des aktivierten Nukleotids mit der Oberfläche andeuten. Gelber Kreis steht für 2-Methylimidazolphosphat. Nach Kaddour et al. (2018).
In einer Reihe von Experimenten wurden täglich reaktive Monomere an das an Montmorillonit Adenosin hinzugefügt, und der Fortschritt wurde 14 Tage lang verfolgt (Ferris et al. 1996). Die Länge der Polymere nahm schrittweise zu Polymeren mit 40 bis 50 Monomereinheiten zu. Eine Kettenlänge mit mindestens 40 Monomereinheiten ist das Minimum, bei der ein RNA-Molekül in der Lage ist, Reaktionen anderer RNA-Moleküle zu katalysieren (Joyce & Orgel 1999; Szostak & Ellington 1993). Sie können damit quasi als fehlende Bindeglieder interpretiert werden, die notwendig sind, wie kurze RNA-Einheiten zu einer RNA-Welt, bestehend aus langkettigen RNA-Molekülen, führen.
Die Sequenzinformation in der RNA bleibt erhalten, wenn eine RNA-Kette als Vorlage für die Synthese ihrer komplementären Kette dient. Die neu entstandene komplementäre Kette kann dann als Vorlage für eine weitere Synthese verwendet werden, was zur Bildung einer RNA führt, die ein Abbild der ursprünglichen Sequenz enthält (Ertem & Ferris 1996).
Weitere Experimente zeigen, dass RNA-Monomere, wenn sie als Triphosphate vorliegen (also die Nukleotidbasen drei Phosphatgruppen gebunden haben, wie wir das auch beim Adenosintriphosphat, ATP, haben) polymerisieren können, wenn sie auf Vulkanglas inkubiert werden, die denen ähneln, die wahrscheinlich vor 4,3 bis 4,4 Milliarden Jahren auf der Erdoberfläche vorhanden waren (Jerome et al. 2022). Diese Polyribonukleinsäure hat eine durchschnittliche Länge von 100-300 Nukleotiden, reicherte sich an und war monatelang stabil. Diese Ergebnisse deuten darauf hin, dass Polyribonukleotide im Hadaikum verfügbar waren, wenn Triphosphate vorhanden waren.
Kim & Benner (2021) berichten, dass Nickel(II) in Gegenwart von Borat-Mineralen beim Verdampfen von Harnstoff, Salzen und zyklischem Trimetaphosphat (CTMP) beachtliche Mengen an Nukleosid-Triphosphaten bilden kann (Abb. 16).
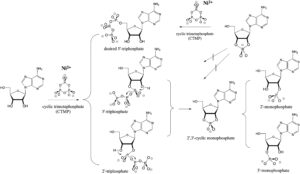
Abb. 16: Vielfältige Reaktionen, die auftreten, wenn CTMP mit einem Nukleosid, hier Adenosin, reagiert. Die Stereochemie ist willkürlich. Die Reaktion von CTMP mit dem freien Nukleosid ergibt das gewünschte 5′-Triphosphat sowie (vermutlich) das unerwünschte 2′-Triphosphat und 3′-Triphosphat. Die 2′- und 3′-Triphosphate reagieren leicht mit der benachbarten Hydroxylgruppe zu einem gemeinsamen 2′,3′-cyclischen Phosphat, das sich dann zu einem Gemisch der 2′- und 3′-Monophosphate in etwa gleichen Mengen öffnet. Alle Reaktionen werden durch Ni2+ katalysiert. In Gegenwart von Borat (oben rechts) wird jedoch vermutlich ein 2′,3′-Borat-Komplex gebildet, der die 2′- und 3′-Hydroxylgruppen schützt und zur bevorzugten Bildung des gewünschten 5′-Triphosphats führt. Nach Kim & Benner (2021).
Einige Forscher haben auch die Schutzwirkung von Mineralien untersucht, die RNA vor dem Abbau schützen (Biondi et al. 2017). An Siliziumdioxid gebundene RNA ist bei einem pH-Wert von 9,5 wesentlich stabiler als das gleiche RNA-Molekül, wenn es frei in wässriger Lösung vorliegt. Dies könnte einen Mechanismus darstellen, durch den eine in einer präbiotischen Umgebung gebildete RNA konzentriert worden sein könnte. Die Wirkung der Mineralien kann auch über den bloßen Schutz der RNA-Moleküle vor dem Abbau durch Hydrolyse hinausgehen. So können Mineraloberflächen eine verstärkte Polymerisation begünstigen (Kaddour et al. 2018).
RNA kann selbst auch Flüssigkristalle bilden (Fraccia et al. 2015, Li et al. 2016).
In all diesen Studien wurden jedoch aktivierte Nukleotide, wie z. B. Imidazol (Ferris et al. 1996), als Ausgangsmaterial verwendet (Abb. 17). Obwohl einige Forscher mögliche präbiotische Synthesewege für die Bildung von imidazolaktivierten Nukleotiden (Yi et al. 2018, Burcar et al. 2015) und ATP (Rodrigues et al. 2019) gefunden haben, ist es fraglich, ob diese in ausreichender Konzentration auf der frühen Erde vorhanden sind.
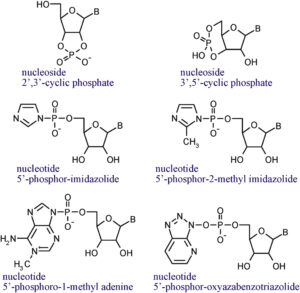
Abb. 17: Struktur aktivierter Nukleotide
Die Synthese von nicht aktivierten Nukleotiden unter präbiotischen Bedingungen ist aber noch nicht sehr gut verstanden. Eine der Schwierigkeiten bei der abiotischen Synthese von Nukleotiden besteht darin, dass ihre Bausteine unter den richtigen Bedingungen kombiniert werden müssen. Verschiedene Bedingungen können dabei jedoch zu isomeren Formen führen. Zudem ist die Phosphorylierung in Wasser bei einer Vielzahl von Temperaturen und pH-Werten thermodynamisch ungünstig, daher wurde die Reaktion meist unter trockenen Bedingungen oder unter Verwendung von Kondensationsmitteln untersucht (Kitadai & Maruyama 2018).
Eine Alternative Möglichkeit RNA unter präbiotischen Bedingungen zu synthetisieren ist, von wesentlich einfacheren Vorstufen wie Glykolaldehyd, Glyceraldehyd, Cyanamid, Cyanoacetylen und Phosphat auszugehen (Powner et al. 2009). Bei diesem Ansatz spielt Phosphat in jedem Schritt der Reaktionsfolge eine wichtige Rolle, da es als pH-Puffer und chemischer Puffer zur Beseitigung unerwünschter Nebenprodukte und als Bestandteil der Nukleotide fungiert.
Der vorgeschlagene Mechanismus verringert die Probleme bei der abiotischen Nukleotidsynthese erheblich. Allerdings ist bei diesem Ansatz nicht klar, ob die Herstellung von Nukleotiden auch in Gegenwart anderer präbiotisch relevanter Substanzen möglich ist.
Einfache Amine oder Ammoniak sind in irgendeiner Form an allen bekannten nicht-enzymatischen Nukleotid-Synthesen beteiligt, was darauf schließen lässt, dass Amine bei der Entstehung der RNA-Welt eine zentrale Rolle gespielt haben könnten (Sponer et al. 2016, Da Silva et al. 2015). Diese konnten auch die ersten Cofaktoren, Puffer und ladungsausgleichenden Kationen gewesen sein. Ammoniumkationen haben einen günstigen Einfluss auf die Bildung von Oligonukleotiden aus nicht aktivierten Monomeren.
2008 (Rajamani et al. 2008) erschien jedoch eine Publikation, die eine andere Hypothese zur nicht-enzymatischen Polymerisierung von Nukleotiden untersuchte und dabei die Rolle von Lipiden hervorhob.
Im Grunde genommen wurden nicht aktivierte Mononukleotide in ein lipidhaltiges Gemisch eingebracht, und dieses Gemisch wurde unter sauren geothermischen Bedingungen wiederholten Zyklen der Dehydratisierung und Rehydratisierung unterzogen. Nach mehreren solcher Zyklen konnte man Polymere nachweisen, die einer Länge von etwa 20 bis 100 Nukleotiden entsprachen.
Darüber hinaus ist es wichtig zu bedenken, dass die Lipidumgebung die Mobilität der Ausgangsreaktanten einschränkt (Toppozini et al. 2013), was eine erhöhte lokale Konzentration ermöglicht und energetisch ansteigende Reaktionen wie die Oligomerisierung von Nukleotiden begünstigt.
Darüber hinaus kann die RNA nach ihrer Bildung von Lipiden in Vesikeln eingekapselt werden, ein Prozess, der durch die Anwesenheit anderer Moleküle wie hydrophober Peptide begünstigt wird (Isaad et al. 2014), ähnlich wie es auf der frühen Erde der Fall gewesen sein soll. Ein solches Reaktionssetting ermöglicht auch einen Informationstranfer von einem Polynukleotid auf ein neu polymerisiertes hin (Olasagasti et al. 2011, Abb. 18).
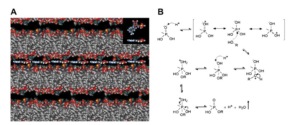
Abb. 18: A. Mononukleotide können organisiert und konzentriert werden, wenn sie bei der Dehydratisierung von Lipidvesikeln eingeschlossen werden. Wenn die Vesikel getrocknet werden, verschmelzen sie zu multilamellaren Strukturen, und alle vorhandenen gelösten Stoffe können zwischen abwechselnden Lipiddoppelschichten eingeschlossen werden. Da das Lipid ein Flüssigkristall ist, können die gelösten Stoffe innerhalb der zweidimensionalen Ebene der Multischicht frei diffundieren. Wenn einzelsträngige DNA-Vorlagen vorhanden sind, können die konzentrierten Mononukleotide eine komplementäre Basenpaarung am Vorlagenmolekül eingehen und dann durch Kondensation unter Wasserverlust Phosphodiesterbindungen bilden, um doppelsträngige DNA zu erzeugen. Zur Veranschaulichung wird hier ein Ribonukleotid gezeigt (siehe Kasten). B. Möglicher Mechanismus für die Bildung von Phosphodiesterbindungen in einer Kondensationsreaktion, die durch simulierte hydrothermale Bedingungen unter sauren Bedingungen, pH 3, angetrieben wird. Unter den wasserfreien Bedingungen der Reaktion würde das Eindringen und Verschwinden von Wasser durch die Lipiddoppelschichten in die CO2-Atmosphäre die Verschiebung der Reaktion hin zur Bildung des Polymers fördern. Nach Olasagasti et al. (2011).
Lipid-Welt
Wenn Lipide bei der Polymerisation von Nukleotiden eine wichtige Rolle spielten, so ist es sinnvoll sich die Polymerisierung von Lipiden anzuschauen. Membranbildende Lipide sind Lipide, die einen hydrophilen und einen hydrophoben Teil besitzen – also amphiphil sind. Dies erlaubt es ihnen, in polaren Lösungsmitteln wie Wasser je nach Beschaffenheit entweder kugelförmige Aggregate oder Doppellipidschichten bilden – wobei immer der hydrophile Teil mit dem polaren Lösungsmittel interagiert. Aus diesen Doppellipidschichten sind alle Biomembranen aufgebaut, welche den Inhalt einer Zelle gegen die Umgebung abgrenzt und somit membranbildende Lipide zu einer der Grundvoraussetzungen allen Lebens macht.
Schon Oparin (1938, 1963) postulierte die Existenz von Zellen und Membranen (Coazervate, Vesikel) als notwendige Voraussetzung für den Ablauf weiterführender Schritte bei der Entstehung des Lebens. Heute geht man allgemein davon aus, dass Vesikel durch die spontane Bildung von Kleinkompartimenten aus unterschiedlichsten Arten von Lipidmolekülen unter wässrigen Bedingungen und somit matrixunabhängig entstanden sind (Deamer & Oro 1980, Deamer & Barchfeld 1982, Baeza et al. 1987, Bachmann et al. 1992; Segre et al. 2001).
Wenn diese Lipidtröpfchen getrocknet und dann rehydriert werden, bilden sie kugelförmige Strukturen, die auch die vorhandene DNA bis zu 100-fach konzentrieren. Solche Strukturen haben somit schon Eigenschaften einer Vorstufe des Lebens (Protolife). Tatsächlich hat Sidney Fox genau solche Strukturen hergestellt, die er Proteinoide nannte, und Oparin stellte Tröpfchen her, die er Koazervate nannte. Diese Strukturen verhalten sich ähnlich wie lebende Zellen, wachsen und teilen sich spontan in Tochtertröpfchen auf.
Das Problem bei der Synthese stabiler Doppelmembranen ist, dass sie stark konzentrations-, temperatur- und pH-Wert-abhängig sind und schon durch geringe Milieuschwankungen rasch wieder zerstört werden können (Thomas & Rana 2007). Doppelmembranen können aber z. B. mithilfe von Pyrit stabilisiert werden. Forscher haben nun herausgefunden, dass sich Vesikel 100-mal so schnell wie üblich bilden können, wenn man den Versuchsmischungen Ton hinzufügt. Einige Vesikel können durch die Lipidwände hindurch Substanzen von außen aufnehmen und sie zum Aufbau neuer Wände und neuer Inhalte verwenden: Sie können also wachsen. Man fand heraus, dass ein aktives Vesikel einen Teil der Membran von einem weniger aktiven Nachbarn “stehlen” und zum Wachstum verwenden kann! Vesikel können eine Art “Reproduktion” in dem Sinne zeigen, dass sich ein großes Vesikel teilen kann, wobei jedes einen Teil des ursprünglichen Vesikelinhalts behält (Chen et al. 2004, Chen & Walde 2010, Chen 2009, Kee & Monnard 2017, Lopez & Fiore 2019, Abb. 19a & b).
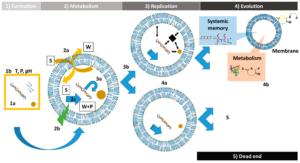
Abb. 19a: Der Lebenszyklus und die Entwicklung der Protozellen. 1: Bildung. Protozellen können aus einem Gemisch von Verbindungen (1a) entstehen, die in einem bestimmten Medium mit charakteristischen Bedingungen wie T: Temperatur, P: Druck, pH (1b) vorhanden sind. 2: Metabolismus. Funktionelle Protozellen sind in der Lage, mit dem Medium in Austausch zu treten (2a), und sie verfügen über eine Energiequelle (2b), die ein katalytisches Netzwerk (2c) antreibt, das aus Substraten (S) Produkte (P) und Abfälle (W) erzeugt. 3: Replikation. Die Teilung einer Protozelle beinhaltet die Replikation eines Teils ihres Inhalts (3a), aber auch die Replikation des Behälters (3b). 4: Entwicklung. Durch Teilung können die Protozellen neue Verbindungen erwerben, die eine höhere Fitness für das System mit sich bringen (4a). Diese Protozellen werden bei der Selektion und Evolution bevorzugt konserviert (4b). Wenn eine Protozelle jedoch die verschiedenen erworbenen Netzwerke nicht beibehält oder über weniger effiziente Netzwerke verfügt, verliert sie den Wettbewerb, und in diesem Fall ist sie eine Sackgasse für das System (5). Diese Abbildung wurde aus Kee et al., 2017, übernommen.

Abb. 19b: Schematische Darstellung der Selbstreproduktion von Vesikeln. Dieser Prozess, der bereits als Wachstum und Teilung bezeichnet wird, besteht darin, dass ein wachsendes Vesikel (a→b) zunächst seine Kugelform in eine Hantelform (b→c) umwandelt und sich dann in zwei kugelförmige Tochtervesikel (c→d) teilt. Knospung, Wachstum und Perlenbildung sind weitere Prozesse, die Teil des Wachstums- und Teilungsprozesses sind, jedoch nicht immer deutlich zu beobachten sind.
Auch bei der Frage der Polymerisation zeigt sich: man braucht keine höhere Macht, keinen intelligenten Designer, damit sich z. B. Aminosäuren zu Proteinen verbinden. Es ist simple Chemie. Es gibt jedoch einige weitere chemische und physikalische Aspekte, die wir uns näher ansehen sollten. Zu diesem gehört u. a. auch die Chiralität, also die Spiegelbildsymmetrie chemischer Moleküle. Was das ist und welche Bedeutung dies für die Entstehung des Lebens hat, werden wir im nächsten Beitrag erfahren.
Literatur
Bachmann, P., Luisi, P., Lang, J. (1992): Autocatalytic self-replicating micelles as models for prebiotic structures. Nature 357:57–59.
Baeza, I., Ibanes, M., Lazcano, A. et al. (1987): Liposomes with polyribonucleotides as model of precellular systems. Origins Life 17:321–331.
Bernal, J. D. (1949): The physical basis of life. Proceedings of the Royal Society of London 357A:537–58.
Biondi, E., Howell, L., Benner, S.A. (2017): Opal Absorbs and Stabilizes RNA—A Hierarchy of Prebiotic Silica Minerals. Synlett 28:84–88.
Brack, A. (1987): Selective emergence and survival of early polypeptides in water. Origins of Life and Evolution of the Biosphere 17:367–79.
Brack, A. (1993): From amino acids to prebiotic active peptides: A chemical restitution. Pure and Applied Chemistry 65:1143–51.
Brack, A. (1994): Are peptides possible support for self-amplification of sequence information. In Selfproduction of supramolecular structures. From synthetic structures to models of minimal living systems, ed. G. R. Fleischaker, S. Colonna, and P. L. Luisi, 115–24. Dordrecht, Netherlands: Kluwer.
Brack, A., Orgel, L. E. (1975): Structures of alternating polypeptides and their possible prebiotic significance. Nature 256:383–87.
Burcar, B. T., Jawed, M., Shah, H., McGown, L. B. (2015): In Situ Imidazole Activation of Ribonucleotides for Abiotic RNA Oligomerization Reactions. Orig. Life Evol. Biosph. 45:31–40.
Cairns-Smith, A. G. (1982): Genetic takeover and the mineral origins of life. Cambridge University Press, Cambridge, 477S.
Cairns-Smith, A. G. (1985): Seven clues to the origin of life: a scientific detective story. Cambridge University Press, Cambridge, 127S.
Chen, F., Yang, D. (2007): Condensation of amino acids to form peptides in aqueous solution induced by the oxidation of sulfur(iv): An oxidative model for prebiotic peptide formation. Orig Life Evol Biosph 37, 47–54. https://doi.org/10.1007/s11084-006-9012-y
Chen, I. A. (2009): Cell division: breaking up is easy to do. Current Biology 19: R327–328.
Chen, I. A., Roberts, R. W., Szostak, J. W. (2004): The emergence of competition between model protocells. Science 305(5689):1474-6. doi: 10.1126/science.1100757. PMID: 15353806; PMCID: PMC4484590.
Chen, I. A., Walde, P. (2010): From Self-Assembled Vesicles to Protocells. Cold Spring Harb. Perspect. Biol. 2: a002170.
Da Silva, L., Maurel, M.C., Deamer, D. (2015): Salt-Promoted Synthesis of RNA-like Molecules in Simulated Hydrothermal Conditions. J. Mol. Evol. 80:86–97.
Deamer, D. W., Barchfeld, G. L. (1982): Encapsulation of macromolecules by lipid vesicles under simulated prebiotic conditions. Journal of Molecular Biology 18:203–206.
Deamer, D. W., Oro, J. (1980): Role of lipids in prebiotic structures. Biosystems, 12(3-4): 167–175. doi:10.1016/0303-2647(80)90014-3
Ding, P. Z., Kawamura, K., Ferris, J. P. (1996): Oligomerization of uridine phosphorimidazolides on montmorillonite: a model for the prebiotic synthesis of RNA on minerals Origins of Life and Evolution of the Biosphere, 26:151-171
Dörr, M., Käßbohrer, J., Grunert, R. et al. (2003): Eine mögliche präbiotische Bildung von Ammoniak aus molekularem Stickstoff auf Eisensulfidoberflächen. Angewandte Chemie 115, 1579–1581.
Ertem, G., Ferris, J. P. (1996): Synthesis of RNA oligomers on heterogeneous templates Nature, 379:238-240
Ferris, J. P. (1993): Catalysis and prebiotic RNA synthesis. Origins of Life and Evolution of the Biosphere 23:307–15.
Ferris, J. P. (2006): Montmorillonite-catalyzed formation of RNA oligomers: the possible role of catalysis in the origins of life Philosophical Transactions of the Royal Society B, 361:1777-1786
Ferris, J. P., Ertem, G. (1992): Oligomerization reactions of ribonucleotides on montmorillonite: Reaction of the 5-phosphorimidazolide of adenosine. Science 257:1387–89.
Ferris, J. P., Ertem, G. (1993): Montmorillonite catalysis of RNA oligomer formation in aqueous solution. A model for the prebiotic formation of RNA Journal of the American Chemical Society, 115: 12270-12275
Ferris, J. P., Hill Jr., A. R., Liu, R., Orgel, L. E. (1996): Synthesis of long prebiotic oligomers on mineral surfaces. Nature 381:59–61.
Ferris, J. P., Joshi, P. C., Wang, K. J., Miyakawa, S., Huang, W. (2004): Catalysis in prebiotic chemistry: application to the synthesis of RNA oligomers Advances in Space Research, 33 (2004), pp. 100-105
Fox, S. W., Harada, K. (1958): Thermal Copolymerization of Amino Acids to a Product Resembling Protein. Science. New Series. 128 (3333): 1214
Fraccia, T. P., Zanchetta, G., Rimoldi, V., Clark, N. A., Bellini, T. (2015): Evidence of Liquid Crystal-Assisted Abiotic Ligation of Nucleic Acids. Orig. Life Evol. Biosph. 45:51–68.
Friedmann, M. P., Torbeev, V., Zelenay, V., Sobol, A., Greenwald, J., Riek, R. (2015): Towards Prebiotic Catalytic Amyloids Using High Throughput Screening. PLOS ONE 10 (12): e0143948 DOI: 10.1371/journal.pone.0143948
Furnes, H., Banerjee, N. R., Muehlenbachs, K., Staudigel, H., de Wit, M. (2004): Early life recorded in Archean Pillow Lavas. Science 304:578–581
Gibbs, D., R. Lohrmann, and L. E. Orgel. 1980. Template-directed synthesis and selective adsorption of oligoadenylates on hydroxyapatite. Journal of Molecular Evolution 15:347–54.
Greenwald, J., Friedmann, M. P., Riek, R. (2016): Amyloid Aggregates Arise from Amino Acid Condensations under Prebiotic Conditions. Angewandte Chemie International Edition, 55(38), 11609–11613. doi:10.1002/anie.201605321
Hill, A. R. Jr., Böhler, C., Orgel, L. E. (1998): Polymerization on the rocks: Negatively charged amino acids. Origins of Life and Evolution of the Biosphere 28:235–43.
Holden, D. T., Morato, N. M., Cooks, R. G. (2022): Aqueous microdroplets enable abiotic synthesis and chain extension of unique peptide isomers from free amino acids. PNAS 119 (42) e2212642119
Isaad, A. L., Carrara, P., Stano, P., Krishnakumar, K. S., Lafont, D., Zamboulis, A., Buchet, R., Bouchu, D., Albrieux, F., Strazewski, P. (2014): A hydrophobic disordered peptide spontaneously anchors a covalently bound RNA hairpin to giant lipidic vesicles. Org. Biomol. Chem. 12:6363–6373.
Jerome, C. A., Kim, H.-J., Mojzsis, S. J., Benner, S. A., Biondi, E. (2022): Catalytic Synthesis of Polyribonucleic Acid on Prebiotic Rock Glasses. Astrobiology 22(6):629-768
Joyce, G. F., Orgel, L. E. (1999): Prospects for understanding the origin of the RNA world. In The RNA world. The nature of modern RNA suggests a prebiotic RNA, ed. R. G. Gesteland, T. R. Cech, and J. F. Atkins. 49–77. Cold Spring Harbor, N. Y.: Cold Spring Harbor Laboratory Press.
Kämpfe, L. (1992): Evolution und Stammesgeschichte der Organismen. Stuttgart.
Kaddour, H., Gerislioglu, S., Dalai, P., Miyoshi, T., Wesdemiotis, C., Sahai, N. (2018): Nonenzymatic RNA Oligomerization at the Mineral-Water Interface: An Insight into the Adsorption-Polymerization Relationship. J. Phys. Chem. C 122:29386–29397
Kawamura, K., Ferris, J. P. (1999): Clay catalysis of oligonucleotide formation: kinetics of the reaction of the 5′-phosphorimidazolides of nucleotides with the non-basic heterocycles uracil and hypoxanthine. Origins of Life and Evolution of the Biosphere 29: 563-591
Kee, T. P., Monnard, P.-A. (2017): Chemical systems, chemical contiguity and the emergence of life. Beilstein J. Org. Chem. 13: 1551–1563.
Kim, H.-J., Benner, S. A. (2021): Abiotic Synthesis of Nucleoside 5′-Triphosphates with Nickel Borate and Cyclic Trimetaphosphate (CTMP) Astrobiology 21(3): 298-306
Kim, H.-J., Ricardo, A., Illangkoon, H. I., Kim, M. J., Carrigan, M. A., Frye, F., Benner, S. A. (2011): Synthesis of Carbohydrates in Mineral-Guided Prebiotic Cycles. J. Am. Chem. Soc. 133(24): 9457–9468 https://doi.org/10.1021/ja201769f
Kitadai, N., Maruyama, S. (2018): Origins of building blocks of life: A review. Geosci. Front. 9:1117–1153.
Leman, L. J., Huang, Z. Z., Ghadiri, M. R. (2015): Peptide Bond Formation in Water Mediated by Carbon Disulfide. Astrobiology 15(9):709-16. doi: 10.1089/ast.2015.1314. Epub 2015 Aug 26. PMID: 26308392.
Leman, L., Orgel, L., Ghadiri, M. R. (2004): Carbonyl sulfide-mediated prebiotic formation of peptides. Science 306(5694):283-6. doi: 10.1126/science.1102722. PMID: 15472077.
Li, C., Cafferty, B.J., Karunakaran, S.C., Schuster, G.B., Hud, N.V. (2016): Formation of supramolecular assemblies and liquid crystals by purine nucleobases and cyanuric acid in water: Implications for the possible origins of RNA. Phys. Chem. Chem. Phys. 18:20091–20096.
Liu, R., Orgel, L. E. (1998): Polymerization of amino acids in aqueous solution. Origins of Life and Evolution of the Biosphere 28:47–60.
Liu, B., Pappas, C. G., Zangrando, E., Demitri, N., Chmielewski, P. J., Otto, S. (2018): Complex Molecules That Fold Like Proteins Can Emerge Spontaneously J. Am. Chem. Soc., DOI: 10.1021/jacs.8b11698
Lopez, A., Fiore, M. (2019): Investigating Prebiotic Protocells for a Comprehensive Understanding of the Origins of Life: A Prebiotic Systems Chemistry Perspective. Life 9(2):49. https://doi.org/10.3390/life9020049
Newsam, J. M. (1986): The zeolite cage structure. Science 231 (4742): 1093-1099
Olasagasti, F., Kim, H.J., Pourmand, N., Deamer, D.W. (2011): Non-enzymatic transfer of sequence information under plausible prebiotic conditions. Biochimie 93:556–561.
Oparin, A. I. (1938): The origin of life. New York.
Oparin, A. I. (1963): Das Leben. Seine Natur, Herkunft und Entwicklung. Jena.
Orgel, L. E. 1998. Polymerization on the rocks: Theoretical introduction. Origins of Life and Evolution of the Biosphere 28:227–34.
Paecht-Horowitz, M., Eirich, F. R. (1988): The polymerization of amino acid adenylates on sodium-montmorillonite with preadsorbed peptides. Origins of Life and Evolution of the Biosphere 18:359–87
Powner, M.W., Gerland, B., Sutherland, J.D. (2009): Synthesis of activated pyrimidine ribonucleotides in prebiotically plausible conditions. Nature 459:239–242.
Rajamani, S., Vlassov, A., Benner, S., Coombs, A., Olasagasti, F., Deamer, D. (2008): Lipid-assisted synthesis of RNA-like polymers from mononucleotides. Orig. Life Evol. Biosph. 38:57–74.
Rasmussen, B. (2000): Filamentous microfossils in a 3,235-million-year-old volcanogenic massive sulphide deposit. Nature 405:676–679
Rodrigues, F., Georgelin, T., Gabant, G., Rigaud, B., Gaslain, F. O. M., Guanzheng, Z., da Fonseca, M. G., Valtchev, V., Touboul, D., Jaber, M. (2019): Confinement and Time Immemorial: Prebiotic Synthesis of Nucleotides on a Porous Mineral Nanoreactor. J. Phys. Chem. Lett. 10:4192–4196
Rodriguez-Garcia, M., Surman, A., Cooper, G. et al. (2015): Formation of oligopeptides in high yield under simple programmable conditions. Nat Commun 6, 8385. https://doi.org/10.1038/ncomms9385
Sawai, H., Higa, K., Kuroda, K. (1992): Synthesis of cyclic and acyclic oligocytidylates by uranyl ion catalyst in aqueous solution. Journal of the Chemical Society, Perkin I:505–8.
Segré, D., Ben-Eli, D., Deamer, D.W. et al. (2001): The Lipid World. Orig Life Evol Biosph 31:119–145. https://doi.org/10.1023/A:1006746807104
Sponer, J.E., Sponer, J., Di Mauro, E. (2016): Four Ways to Oligonucleotides Without Phosphoimidazolides. J. Mol. Evol. 82:5–10.
Suzuki, I. (1999): Oxidation of inorganic sulfur compounds: Chemical and enzymatic reactions. Can J Microbiol 45:97–105
Szostak, J. W., Ellington, A. D., eds. (1993): In Vitro selection of functional RNA sequences. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.
Thomas, J. A., Rana, F. R. (2007): The influence of environmental conditions, lipid composition, and phase behavior on the origin of cell membranes. Origins of Life and Evolution of the Biosphere 37:267–285.
Toppozini, L., Dies, H., Deamer, D.W., Rheinstadter, M.C. (2013): Adenosine Monophosphate Forms Ordered Arrays in Multilamellar Lipid Matrices: Insights into Assembly of Nucleic Acid for Primitive Life. PLoS ONE 8:e62810.
Wächtershäuser, G. (1988a): Before enzymes and templates: theory of surface metabolism. Microbiological Reviews 52, 452–484.
Wächtershäuser, G. (1988b): Pyrite formation, the first energy source for life: a hypothesis. Systematic and Applied Microbiology 10, 207–210.
Wächtershäuser, G. (1990): Evolution of the first metabolic cycles. PNAS 87, 200–204.
Wächtershäuser, G. (1992): Ground works for an evolutionary biochemistry: the iron-sulfur world. Progress in Biophysics and Molecular Biology 58, 85–201.
Wächtershäuser, G. (1994): Life in a ligand sphere, PNAS 91, 4283–4287.
Wächtershäuser, G. (2000): Origin of life. Life as we don’t know it. Science 289, 1307–1308.
Wright, P. A., Thomas, J. M., Cheetharn, A. K., Nowak, A. K. (1985): Localizing active sites in zeolite catalysis: neutron powder profile analysis and computer simulation of deuteropyridine bound to gallozeolite-L. Nature 318: 611-614.
Yi, R. Q., Hongo, Y., Fahrenbach, A. C. (2018): Synthesis of imidazole-activated ribonucleotides using cyanogen chloride. Chem. Commun. 54:511–514.