Wir werden alle älter, das ist nicht zu verhindern. Dass wir älter werden, hat z. T. auch mit unserer DNA zu tun, bzw. ihrer Vermehrung, mit der wir uns hier befassen werden.
Wenn sich eine Zelle teilt, so muss ihr Genom auch an die Tochterzellen weitergegeben werden. Damit dies geschehen kann, muss die DNA verdoppelt werden. Als Replikation bezeichnet man einen zellulären Vorgang, bei dem eine DNA-Doppelhelix mithilfe spezifischer Enzyme identisch verdoppelt wird. Nur unter dieser Voraussetzung entstehen bei einer nachfolgenden Zellteilung Tochterzellen mit gleicher genetischer Ausstattung.
Obwohl das Grundschema der Replikation einleuchtet, sind die Details jedoch sehr komplex. Folgende Besonderheiten sind dabei zu beachten:
- Die komplementäre Basenpaarung soll fehlerfrei erfolgen: Adenin paart stets mit Thymin und Guanin stets mit Cytosin.
- Alle bekannten Enzyme, die DNA entlang einer Matrize synthetisieren, arbeiten nur in einer bestimmten Richtung. Außerdem können sie einen vorhandenen Strang zwar verlängern, aber nicht neu beginnen.
- Die beiden DNA-Stränge in der DNA-Doppelhelix verlaufen antiparallel, d. h. sie haben eine entgegengesetzte Leserichtung. Die Synthese der beiden Tochterstränge wird sich also unterscheiden.
- Das lange DNA-Molekül liegt im Zellkern verpackt vor.
- Die Replikation muss zeitlich mit anderen Prozessen an der DNA und in der Zelle abgestimmt sein.
Meselson-Stahl-Experiment
Es gibt theoretisch gesehen drei Möglichkeiten wie die DNA verdoppelt werden könnte (Abb. 1):
Zunächst ist ein Replikationstyp möglich, der als dispersiv bezeichnet wird. Die Tochtermoleküle bestehen aus einer abwechselnden Mischung von alten und neu synthetisieren Strängen.
Von einer konservativen Verdopplung spricht man, wenn eine DNA-Doppelhelix komplett neu synthetisiert wird und die elterliche Doppelhelix unverändert bleibt.
Bei einer semikonservativen Verdopplung trennen sich die Elternstränge und zu jedem der beiden Elternstränge wird der komplementäre Strang neu synthetisiert. So ergeben sich auch hier zwei identische DNA-Doppelhelices; in diesem Fall besteht aber jeder Doppelstrang jeweils aus einem Elternstrang und einem neu synthetisierten Tochterstrang.
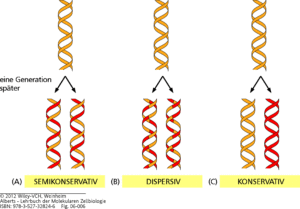
Abb. 1: Möglichkeiten der Replikation
Tatsächlich folgt die Replikation der DNA dem semikonservativen Weg. Dies wurde durch das Experiment von Matthew Meselson und Franklin Stahl nachgewiesen (Abb. 2).
Dafür gaben sie die Coli-Bakterien auf ein Nährmedium, dass das Stickstoffisotop 15N enthielt. So kam es zu einem Einbau des Isotops in die DNA der Bakterien. Nach einer Weile überführten sie die Bakterien auf ein Nährmedium mit dem Stickstoffisotop 14N. Die beiden Isotope unterscheiden sich nur im Hinblick auf ihre Masse und das machten sich Meselson und Stahl zu Nutze: Sie extrahierten die DNA der Nachfolgeneration und zentrifugierten das Erbgut. Das Ergebnis (eine Bande) lag genau zwischen den Dichtegradienten von 14N und 15N, was die konservative Replikation aussschloss. Denn sonst hätte das zentrifugierte Erbgut sich auf die Dichtegradienten 14N und 15N aufteilen müssen, weil die Elterngeneration (P1) der Bakterien 15N eingebaut hatte und die erste Tochtergeneration (F1) nur 14N hätte einbauen können. Damit kamen nur noch die semikonservative oder die disperse Replikation in Betracht. Der Versuch wurde um noch eine weitere Tochtergeneration weitergeführt (F2). Doch dieses Mal bekam man zwei verschieden schwere Banden bestehend aus einer leichten 14N Bande und einer schwereren 14N+15N Bande. Disperse Replikation war damit ebenso ausgeschlossen, denn in diesem Fall hätte sich nur eine Bande bilden dürfen (durch den immer wieder gleichmäßigen Einbau von 14N und 15N).
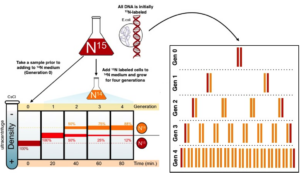
Abb. 2: Meselson-Stahl-Experiment
Ablauf der Replikation
Wie läuft die Replikation der DNA ab? Hier sind mehrere Enzyme beteiligt (Abb. 3)
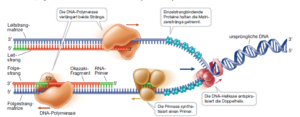
Abb. 3: Replikation
- Zuerst muss die Doppelhelix der DNA entwunden werden. Dies macht das Enzym Topoisomerase
- Bei der Replikation werden die beiden Einzelstränge reißverschlussartig voneinander getrennt, es entsteht somit eine Y-förmige Replikationsgabel. Jeder der frei gewordenen Einzelstränge dient als Matrize für die Synthese des Tochterstrangs. Die Trennung der Doppelstränge wird durch ein Enzym Helikase ermöglicht. Einzelstrangbindende Proteine binden an die getrennten Stränge und verhindern, dass sich dort die Doppelhelix wieder bildet.
- Die Primase synthetisiert an den 3′ Enden der DNA sogenannte Primer, das sind RNA-Moleküle, die für den Beginn der eigentlichen Replikation nötig sind und als Startpunkt dienen.
- Die Synthese des neuen Tochterstranges erfolgt durch das Enzym DNA-Polymerase III. Und hier stehen wir vor einem kleinen Problem: Die DNA-Polymerase III kann nur in eine Richtung kontinuierlich synthetisieren. Wir haben auf dem Beitrag des Aufbaus der DNA gelernt, dass das Zuckermolekül Desoxyribose mit den 5. und 3. C-Atom eine Verbindung mit dem Phosphat eingeht. Dabei bildet sich eine Phosphatbrücke zwischen dem 5´-C-Atom des einen und dem 3´-C-Atom des anderen Zuckers im benachbarten Nucleotid. Jede Polynukleotidkette hat somit ein 5´-Ende mit einer freien Phosphatgruppe und ein 3´-Ende mit einer freien OH-Gruppe.
- Die DNA-Doppelhelix ist also aus zwei antiparallelen Strängen aufgebaut, aus einem Vorwärtsstrang in 3´->5´-Richtung und einem Rückwärtsstrang in 5´->3´-Richtung. Am Vorwärtsstrang kann die DNA-Polymerase kontinuierlich den Leitstrang in 5´->3´ -Richtung Nukleotid für Nukleotid synthetisieren. Am Rückwärtsstrang wird der Folgestrang synthetisiert. Da er jedoch antiparallel ist, unterliegt er einem besonderen Synthese-Mechanismus.
- Reiji Okazaki löste dieses Problem im Jahre 1965. Er zeigte experimentell, dass auch der Rückwärtsstrang in 3´->5´-Richtung von der DNA-Polymerase III abgelesen wird – allerdings stückweise. Denn das funktioniert nur, wenn immer wieder neue Primer gesetzt werden. D. h. die Replikationsgabel öffnet sich und an dem Rückwärtsstrang lagern sich versetzt immer neue Primer an. In die Lücken zwischen den Primern kann die DNA-Polymerase III ansetzen und fragmentweise den Folgestrang am Rückwärtsstrang synthetisieren. Diese einzeln synthetisierten Stücke der DNA bezeichnet man als Okazaki-Fragmente. Man spricht auch von einer diskontinuierlichen Bildung des DNA-Stranges.
- Die DNA-Polymerase I entfernt nun die Primer und ersetzt ihn durch DNA. Zurück bleibt eine kleine Bindungslücke zwischen den aneinandergrenzenden Okazaki-Fragmenten, die von der DNA-Polymerase I nicht geknüpft werden kann. Das Herstellen dieser Bindung wird von der DNA-Ligase katalysiert, wodurch die Fragmente miteinander verbunden werden und der Folgestrang ein Ganzes bildet.
- Die Replikation der DNA erfordert Energie. Die DNA-Polymerase nutzt als Einzel-Nukleotide zur DNA-Synthese Desoxyribonucleosidtriphosphate. Durch die Abspaltung von zwei Photsphatgruppen wird Energie frei und die nun mit nur einem Phosphat bestückten Einzel-Nukleotide bilden den neuen DNA-Strang.
Telomere und Alterungsprozess
Wir haben im vorherigen Beitrag in Erfahrung gebracht, dass die Enden der Chromosomen der Eukaryoten als Telomere bezeichnet werden. Sie bestehen aus repetitiver DNA. Beim Menschen ist TTAGGG-30 die Telomersequenz, die sich an jedem Ende eines Chromosoms etwa 2500-mal wiederholt. An diese Sequenzwiederholungen binden spezielle Proteine, die verhindern, dass das DNA-Reparatursystem die Enden als Brüche erkennt. Darüber hinaus können die Wiederholungen Schleifen bilden, die eine ähnliche Schutzfunktion besitzen.
Es gibt jedoch noch ein weiteres Problem mit den Enden der Chromosomen. Wie wir erfahren haben, erfolgt die Synthese des Folgestrangs durch das Anfügen von Okazaki-Fragmenten an RNA-Primer. Wenn nun der endständige, letzte RNA-Primer entfernt wird, kann keine DNA mehr synthetisiert werden, um ihn zu ersetzen, da es kein 3‘-Ende mehr gibt, das verlängert werden könnte. Es bleibt also an beiden Enden der fertigen DNA am jeweils komplementären Strang ein kurzes, ungepaartes DNA-Stück übrig; meistens wird es enzymatisch entfernt. Auf diese Weise wird das Chromosom bei jeder Zellteilung etwas kürzer. Bei jeder Replikation können Telomere 50 – 200 Basenpaare verlieren.
Nach vielen Teilungen können so die Gene in der Nähe der Chromosomenenden verloren gehen, und die Zelle stirbt. Dieser Effekt erklärt teilweise, warum viele Zelllinien nicht die gesamte Lebenszeit eines Organismus bestehen bleiben: Ihre Telomere gehen verloren. Andererseits verfügen sich ständig teilende Zellen wie die Stammzellen des Knochenmarks und die Urkeimzellen, die die Sperma- und Eizellen produzieren, über einen speziellen Mechanismus, um ihre Telomer-DNA zu erhalten. Das Enzym Telomerase fügt bei diesen Zellen die verloren gegangenen Telomersequenzen an. Die Telomerase enthält eine RNA-Sequenz, die als Matrize für die Sequenzwiederholung der Telomere fungiert.
Bei über 90% aller Tumoren des Menschen wird die Telomerase exprimiert. Wahrscheinlich ist sie ein wichtiger Faktor für die Fähigkeit von Krebszellen, sich ständig zu teilen. Da die meisten normalen Zellen diese Fähigkeit nicht besitzen, ist die Telomerase ein interessantes Ziel für Medikamente, die Tumoren spezifisch angreifen sollen. Auch im Zusammenhang mit dem Alterungsprozess ist die Telomerase im Gespräch. Wenn menschliche Zellkulturen mit einem Gen transformiert werden, das die Telomerase in großen Mengen exprimiert, verkürzen sich deren Telomere nicht. Solche Zellen leben nicht nur 20–30 Generationen und sterben dann, sondern sie werden immortalisiert (unsterblich). Zu klären ist hier noch, inwieweit sich dieser Befund auf das Altern eines ganzen Organismus anwenden lässt.
DNA-Reparatur
Die DNA wird äußerst exakt repliziert und bleibt daher zuverlässig erhalten. Das ist für die Funktionstüchtigkeit aller Zellen essenziell – gleichgültig, ob es sich um einen Prokaryoten oder eine Zelle in einem komplexen, vielzelligen Organismus handelt.
Die Replikation der DNA erfolgt aber nicht mit absoluter Genauigkeit, und DNA unterliegt Veränderungen durch chemische Substanzen und andere Umweltfaktoren.
DNA-Polymerasen machen Fehler bei der Synthese der Polynucleotidstränge – im Allgemeinen wird pro 100.000 replizierten Nucleotiden eine falsche Base eingebaut. Das erscheint zwar angesichts der etwa 3Mrd. Basenpaare beim Menschen vielleicht als wenig bedeutsam, aber die Fehler addieren sich. Wenn die Fehler der DNA-Polymerase nicht repariert würden, gäbe es bei jeder Teilung einer menschlichen Zelle in den neu synthetisierten DNA-Strängen 60.000 falsche Basen. Es ist sogar noch schlimmer: Da die Basen selbst chemisch instabil sind, können sie durch äußere Faktoren wie Strahlung geschädigt werden. So entstehen Mutationen, an denen die korrekte Paarung verhindert wird.
Vorteilhafterweise werden Fehler bei der DNA-Replikation in den Zellen korrigiert und geschädigte Nucleotide repariert. Zellen verfügen mindestens über drei verschiedene DNA-Reparaturmechanismen (Abb. 4):
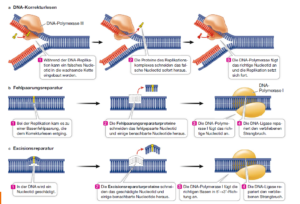
Abb. 4: Reparatur
Ein Mechanismus zum Korrekturlesen korrigiert Replikationsfehler, sobald die Polymerase sie erzeugt. Die meisten DNA-Polymerasen führen die Korrekturlesefunktion jedes Mal durch, wenn sie ein neues Nucleotid in den wachsenden DNA-Strang einfügen. Wenn eine DNA-Polymerase eine Fehlpaarung der Basen erkennt, entfernt sie das unpassend eingebaute Nucleotid und startet einen neuen „Versuch“.
Die Fehlerrate dieses Vorgangs beträgt nur eine von 10.000 reparierten Basenpaaren.
Dadurch verringert sich die Fehlerrate bei der Replikation insgesamt auf einen Fehler pro 10Mrd. replizierten Basen.
Ein zweiter Mechanismus zur Fehlpaarungsreparatur prüft die DNA unmittelbar nach der Replikation und korrigiert alle Basenfehlpaarungen (mismatches).
Nach der Replikation der DNA durchsucht eine zweite Gruppe von Proteinen das neu replizierte Molekül auf Basenfehlpaarungen, die dem Korrekturlesen entgangen sind. So kann ein solcher Mechanismus zur Fehlpaarungsreparatur beispielsweise ein A-C-Paar erkennen, wo eigentlich ein A-T-Paar sein sollte. Das Reparatursystem erkennt, welche der beiden Basen im A-C-Paar die falsche ist und führt die Reparatur durch. Wenn das korrekte Paar A-T ist, ersetzt das Reparatursystem C durch T und stellt so das A-T-Paar wieder her.
DNA-Moleküle können auch während der Interphase einer Zelle beschädigt werden, wenn sich also die DNA nicht verdoppelt. Hochenergetische Strahlung, chemische Substanzen aus der Umgebung und spontan auftretende chemische Reaktionen können die DNA schädigen. Wenn beispielsweise benachbarte Thymine auf demselben DNA-Strang ultraviolettes Licht absorbieren, bilden sich kovalente Bindungen zwischen den Basen, sodass ein Thymindimer entsteht. Diese Dimere stören die Basenpaarung bei der Replikation, sodass zufällige Basen eingebaut werden. Dies ist die primäre Ursache für Hautkrebs bei Menschen. Für diese Art von Schäden sind Mechanismen der Excisionsreparatur als dritte Möglichkeit, zuständig.