Allgemeiner Aufbau der Nukleinsäuren
Text als pdf
Eine wichtige Frage, die sich mit dem Ursprung des Lebens beschäftigt, ist der Ursprung des genetischen Materials. In den letzten Episoden hatten wir uns mit den Ursprüngen des Stoffwechsels befasst in den hydrothermalen Schloten der Tiefsee. Doch erst mit der „Erfindung“ der Vererbung konnte sich das Leben aus diesen porösen Gesteinen befreien, komplexere Stoffwechselvorgänge etablieren und neue Lebensräume erobern. Heute sind die Informationen für die Fortpflanzung und die Herstellung weiterer Kopien lebender Organismen in der DNA verschlüsselt (Abb.1).
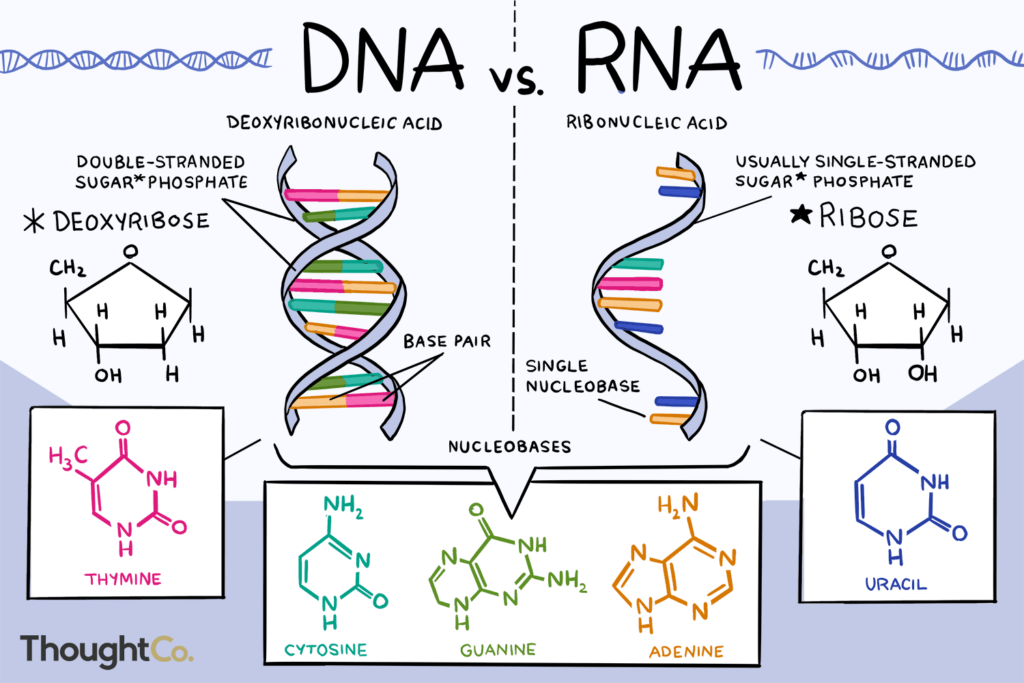
Abb. 1: genereller Aufbau der Nukleinsäuren
Die DNA gehört zur Stoffgruppe der Nukleinsäuren (Savada et al. 2019, Kapitel 4; Campbell et al. 2014; Kapitel 5). Es handelt sich hierbei um Polymere, deren Untereinheiten (Monomere) die Nukleotide sind. Nukleotide bestehen aus drei prinzipiellen Bausteinen: Phosphaten, Zucker, Pyrimidin- oder Purin-Basen. Es gibt vier verschiedene Basen in der DNA: Cytosin und Thymin, die zu den Pyrimidin-Basen gehören sowie Guanin und Adenin, die zu den Purin-Basen gehören. Bei dem Zuckermolekül handelt es sich um die 2-Desoxyribose.
Die DNA ist aber nicht die einzige Nukleinsäure, dass eine große Rolle in der Biologie spielt. Es gibt auch noch den engen molekularen Verwandten, die Ribonukleinsäure, kurz RNA (Abb. 1). Molekular betrachtet ähneln sich beide Nukleinsäuren, es gibt aber zwei wichtige Unterschiede. Der Zucker der DNA besteht wie erwähnt aus 2-Desoyxribose, da die Hydroxylgruppe (OH-Gruppe) am C2-Atom des Zuckers entfernt wurde. Die RNA enthält den Zucker Ribose, bei dem diese OH-Gruppe noch vorhanden ist. Dieser Unterschied hat einen erheblichen Einfluss auf die chemischen Eigenschaften beider Moleküle. Kommt die DNA stets als Doppelstrang vor, ist die RNA bis auf wenige Ausnahmen als Einzelstrang aktiv. Dies hat für die RNA enorme Vorteile, denn es können sich komplementäre Sequenzen desselben Strangs miteinander feste Bindungen eingehen und komplexe dreidimensionale Strukturen bilden, die vielfältige, auch enzymatische, Funktionen erfüllen können. Es gibt eine große Vielfalt an RNA-Molekülen innerhalb einer jeden Zelle (Abb. 2). Bekannt ist z. B. die messengerRNA, kurz mRNA, sowie die transfer-RNA, kurz tRNA, die bei der Synthese von Proteinen eine wichtige Rolle spielen. Andere RNAs, wie die micro-RNAs, spielen bei der Genregulation eine wichtige Rolle. RNAs können also nicht nur genetische Informationen speichern, sondern übernehmen eine wichtige Rolle im Zellstoffwechsel. Da die RNA aber weniger stabil ist als die DNA, dient letztere als reines „Speichermedium“ in der Zelle. Auch heute noch wird die Desoxyribose der DNA aus der Ribose der RNA im Zellstoffwechsel synthetisiert, indem die OH-Gruppe entfernt wird, was die Stabilität der DNA erhöht.
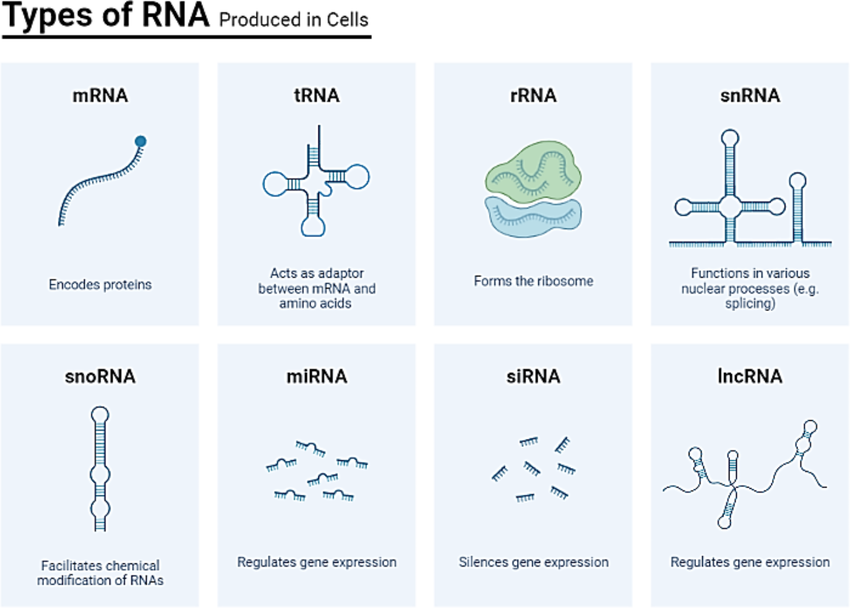
Abb. 2: Typen von RNA
Der zweite Unterschied zwischen DNA und RNA besteht darin, dass bei der RNA die Base Thymin durch Uracil ersetzt wird. Auch hier ist der chemische Unterschied gering. Er besteht aus einer einzigen Methyl-Gruppe, also CH3-Gruppe, die in Thymin vorhanden ist und in Uracil nicht. Thymin ist also nichts weiter als ein chemische modifizierte, mit einer Methylgruppe versehene Form des Uracil. Auch hier bewirkt das Thymin anstelle des Uracils eine chemisch stabilere Form, weswegen Thymin in der DNA vorkommt.
Die präbiotische Synthese der Nukleinsäuren ist schwieriger als die der Proteine, aber nicht unmöglich. Wir haben uns schon in bisherigen Beiträgen mit der Entstehung der RNA und deren Polymeren befasst, weswegen dies hier nicht wiederholt werden soll.[1],[2]
Die wirklich schwierige Frage ist aber nicht die präbiotische Synthese von Nukleinsäuren, sondern eine ganz andere. Lebewesen bestehen aus Proteinen und Nukleinsäuren. Für die Synthese von Proteinen werden Nukleinsäuren gebraucht. Andersrum kann ein solches Speichermedium wie die DNA sich ohne Proteine weder kopieren, noch diese herstellen. Für die Verdoppelung, also Replikation, der DNA sind verschiedene Enzyme, also Proteine, notwendig. Und auch für die Proteinsynthese, bei der sich mRNA-Moleküle an die DNA anlagern und den genetischen Code ablesen, Enzyme notwendig. Und auch bei der Regulation der Genaktivität sind Transkriptionsfaktoren beteiligt, die meistens ebenfalls Proteine sind. Wir haben hier also ein typisches Henne-Ei-Problem. Was war zuerst da: das Proteinreplikationssystem oder das Nukleinsäure-Replikationssystem?
RNA – Informationsspeicher und Enzym zugleich?
Gerade die RNA mit ihren katalytischen wie auch informationsspeichernden Eigenschaften bietet glücklicherweise einen Weg, dieses Rätsel zu lösen (Visser 1984). Im Jahr 1968 schlug der Mitentdecker der DNA, Francis Crick, erstmals vor, dass die früheste Urzelle ein RNA-Strang war (Woese 1967).
Dann, Anfang der 1980er Jahre entdeckten zwei Wissenschaftlerteams unabhängig voneinander kleine Enzyme, die die chemischen Bindungen, die Nukleinsäuren zu Ketten verbinden, aufbrechen und neu bilden konnten (Kruger et al. 1982, Altman et al. 1989). Die Enzyme erfüllten ihre Aufgabe nur unzureichend. Im Vergleich zu den Hunderten von bereits bekannten Enzymen dieser Art waren sie bei ihrer katalytischen Aufgabe langsam und zeigten wenig Vielseitigkeit. Dennoch wurde die Entdeckung als einer der bedeutendsten biologischen Durchbrüche dieser Zeit anerkannt. Im Jahr 1989 erhielten die Leiter des Teams, Sidney Altman und Thomas Cech, gemeinsam den Nobelpreis.
Aber was war an diesen so langsam arbeitenden Enzymen so besonders? Die Antwort liegt darin, dass die Enzyme nicht aus Proteinen, sondern aus RNA bestehen. Bis 1982 waren alle bekannten Enzyme Proteine. RNA wurde oft als die arme Cousine der DNA betrachtet, die die Aufgabe hat, die genetische Information von der DNA, in der sie gespeichert ist, zu den Proteinen zu transportieren, die die eigentliche Arbeit in der Zelle verrichten. Doch die Entdeckung der RNA-Enzyme, der Ribozyme, durch Altman und Cech veränderte die Sichtweise der Biologen auf die Vorgänge in der Zelle.
Da die RNA sowohl die Fähigkeit zur Informationsspeicherung und -übertragung als auch die Fähigkeit zur biologischen Arbeit besitzt, gehen Forscher heute davon aus, dass sie bei der Entstehung des Lebens sowohl den Proteinen als auch der DNA vorausgegangen ist. Man spricht dann von der sogenannten RNA-Welt (Gilbert 1986). Eine RNA-Welt ist verlockend, weil sie viele Merkmale des modernen Lebens aufweisen würde, ohne dass mehr als ein paar organische Moleküle in Lösung nötig wären. Die Hypothese der RNA-Welt beruht auf der seit der Entdeckung der Ribozyme, dass RNA sowohl einen Genotyp als auch einen Phänotyp besitzen kann (Joyce 1989).
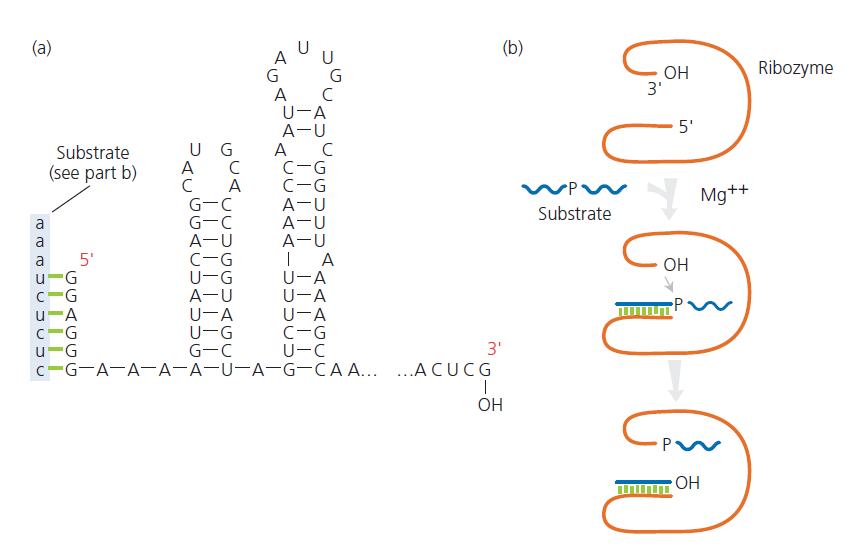
Abb. 3: Das Ribozym aus Tetrahymena thermophila. (a) Die primäre Nukleotidsequenz ist der Genotyp. Eine Sekundärstruktur entsteht, wenn sich die Nukleotide bei der Rückfaltung des Moleküls paaren. (b) Die von dem Ribozym in vitro durchgeführte Katalyse ist der Phänotyp. Ein kurzes Oligonukleotidsubstrat (blau) bindet an das 5‘-Ende des Ribozyms (orange) durch komplementäre Basenpaarung (grüne Häkchen). Das Ribozym katalysiert das Aufbrechen einer Phosphoesterbindung im Substrat und die Ligation des 3‘-Fragments an sein eigenes 3‘-Ende.
Der Genotyp ist die primäre Sequenz von Nukleotiden entlang der RNA (Abb. 3a), ähnlich wie der Genotyp eines modernen Organismus die Sequenz von Nukleotiden entlang der DNA im Chromosom ist. Die katalytische RNA enthält beispielsweise zwischen 30 und 1.000 Ribonukleotide, die ihre primäre Sequenz und damit ihren Genotyp bilden. Das von Cech und Kollegen entdeckte Tetrahymena-Ribozym (Kruger et al. 1982; Zaug & Cech 1986) erstreckt sich über 400 Nukleotide. Bei dieser RNA handelt es sich um ein Intron (eine Zwischensequenz zwischen zwei Genen), das zwei Regionen des Tetrahymena-Genoms trennt, die für ribosomale RNA-Gene (rRNA) kodieren.
Dadurch, dass die RNA als Einzelstrang vorliegt, kann es sich selbst an den komplementären Basen falten und dreidimensionale Strukturen bilden. In Ribozymen kann dieser gefaltete Zustand eine aktive Stelle aufweisen, die es der RNA ermöglicht, eine chemische Reaktion mit einem Substrat zu katalysieren, ähnlich wie ein Proteinenzym. Diese Reaktivität verleiht der RNA ihren Phänotyp. Das Ribozym, das Cech und Kollegen gefunden haben, besitzt die katalytische Fähigkeit, sich nach der Transkription zwischen zwei benachbarten rRNAs herauszuspleißen (Kruger et al. 1982). Im Reagenzglas kann eine verkürzte Version des Ribozyms eine Phosphoester-Transferreaktion an einem kurzen RNA-Substrat, einem so genannten Oligonukleotid (einem Stück einzelsträngiger Nukleinsäure mit einer Länge von 5 bis 30 Nukleotiden), katalysieren (Abb. 3b).
Dutzende von natürlich vorkommenden Ribozymen sind entdeckt worden (Gesteland et al. 1999). Bei den meisten von ihnen geht es um die Bildung und das Aufbrechen von Phosphoesterbindungen in RNA oder DNA (Abb. 3b). Die Chemie dieser Reaktionen ist genau das, was für die Replikation von Nukleinsäuren benötigt wird. Diese Beobachtung unterstützt die Idee einer ursprünglichen RNA-Welt, in der die RNA für ihre eigene Replikation verantwortlich ist. Wenn ein RNA-Molekül eine Kopie von sich selbst erstellen könnte und dabei die Möglichkeit von Fehlern – Mutationen – in Kauf nehmen würde, dann würde es viele der Merkmale des modernen Lebens aufweisen und könnte daher als lebendig betrachtet werden.
Die RNA-Welt-Hypothese besagt, dass die Urform ein auf RNA basierendes lebendes System war, das sich später zu Lebensformen wie den heutigen entwickelte, in denen die DNA biologische Informationen speichert und Proteine diese Informationen manifestieren. Die DNA ist als Informationsspeicher besser geeignet, weil sie chemisch stabiler ist als die RNA. Vor allem wenn sie doppelsträngig ist, kann die DNA hohen Temperaturen und dem spontanen Abbau durch Säuren oder Basen besser widerstehen. Was ist der Beweis dafür, dass RNA uralt ist? Das Vorhandensein katalytischer RNA ist entscheidend, aber es gibt auch andere Hinweise. Ein Anhaltspunkt dafür, dass RNA in frühen Lebensformen eine Rolle spielte, ist ihre Rolle in der Maschinerie, die Zellen für Replikation und Stoffwechsel nutzen (Crick 1966; White 1976, Abb. 2). Die konservierteste und universellste Komponente der Informationsverarbeitungsmaschinerie ist zum Beispiel der Apparat für die Übersetzung genetischer Informationen in Proteine: das Ribosom (Harris et al. 2003; Koonin 2003, Abb. 4). Dieser Apparat enthält zwar Proteine, aber auch ribosomale RNA (rRNA). Ribosomen enthalten nicht nur selbst RNA, sie benötigen auch RNA-Adaptoren (tRNAs), um ihre Aufgabe zu erfüllen. Außerdem ist es der RNA-Teil der Ribosomen, der die katalytischen Schritte der Proteinsynthese durchführt (Nissen et al. 2000; Steitz & Moore 2003). Ein weiteres Argument für das hohe Alter der RNA ist, dass die Basiswährung für biologische Energie die Ribonukleosidtriphosphate wie ATP und GTP sind (Joyce 1989). Diese Moleküle sind an fast allen Energieübertragungsvorgängen in allen Zellen beteiligt und sind sogar Bestandteile von Elektronentransfer Cofaktoren wie NAD (Nicotinamid-Adenin-Dinukleotid), FAD (Flavin Adenindinukleotid) und SAM (S-Adenosylmethionin). Wenden wir uns aber eher der Frage zu: Kann die RNA evolvieren?
Abb. 4: Ribosom
Die experimentelle Evolution der RNA
RNA-Sequenzen können einen Bauplan für ihre eigene Replikation liefern. Zu jeder RNA-Sequenz können wir durch Basenpaarung eine komplementäre Sequenz bilden. Wie die DNA hat auch die RNA die Fähigkeit, vererbbare Informationen zu speichern, die weitergegeben werden können. Ein gutes Beispiel ist der Lebenszyklus des HI-Virus (Savada et al. 2019, Kapitel 25, Campbell et al. 2014, Kapitel 19) . HIV verwendet das Proteinenzym Reverse Transkriptase, um seinen RNA-Strang in ein DNA-Komplement zu kopieren, das dann in doppelsträngige DNA umgewandelt werden kann. Da RNA genetische Informationen speichern kann, sollten Populationen von RNA-Molekülen in der Lage sein, sich weiterzuentwickeln. Wenn ein RNA-Molekül einen Phänotyp hat, der eine bestimmte chemische Reaktion katalysiert, können wir dann eine Selektion durchführen, um diesen Phänotyp zu verbessern oder zu verändern, und eine vererbbare Veränderung beobachten?
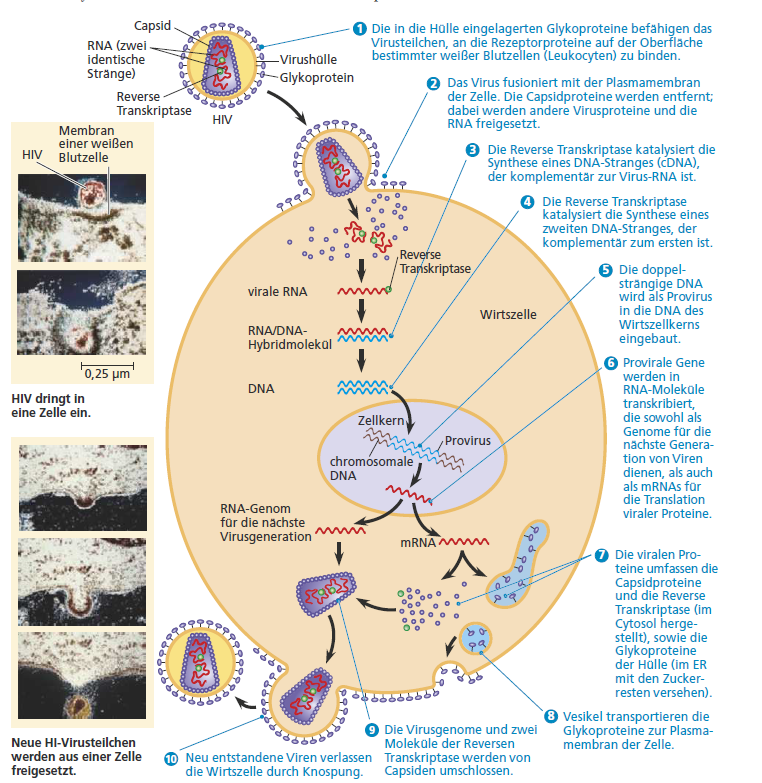
Abb. 5: Der Vermehrungszyklus von HIV, dem Retrovirus, das AIDS verursacht. Beachtet, wie in Schritt 5 die anhand des RNA-Genoms synthetisierte DNA in das Wirtsgenom (die chromosomale DNA im Zellkern) integriert wird – ein für Retroviren typisches Verhalten. Die Bilder am linken Rand (nachträglich gefärbte transmissionselektronenmikroskopische Aufnahmen) zeigen HIV beim Eintritt und beim Verlassen eines weißen Blutkörperchens (Leukocyt) des Menschen.
Experimente der katalytischen Kapazität des Tetrahymena-Ribozyms (Mills et al 1967, Beaudry & Joyce 1992, Abb. 6) konnten diese Frage beantworten. Dabei wurde zunächst eine große Population von RNA-Molekülen mit einer Mutationsrate von 5% im Tetrahymena-Ribozym hergestellt. Dann wurde diese Mutantenpopulation mit einer neuen Aufgabe konfrontiert, um bestimmte Genotypen auszuwählen (Abb. 6a). Die Aufgabe bestand Fall darin, dass das Substrat-Oligonukleotid in Form von DNA und nicht von RNA bereitgestellt wurde. Die natürlich vorkommende Sequenz des Tetrahymena-Ribozyms (der „Wildtyp“), mit dem diese Experimente begannen, konnte ein DNA-Substrat nur mit einer sehr langsamen Geschwindigkeit spalten. Entsprechend wurden in dem Mutanten-Pool Sequenzvarianten gesucht, die zufällig eine erhöhte Kapazität zur DNA-Spaltung aufwiesen. Die Forscher inkubierten die mutierte RNA-Population eine Stunde lang mit einem DNA-Substrat und vervielfältigten dann die Ribozym-RNA durch Zugabe von zwei Protein-Enzymen – der reversen Transkriptase und der RNA-Polymerase (Abb. 6b). Die Population neuer RNAs, die aus diesem Zyklus hervorgeht, enthält neue Variationen, die aus Kopierfehlern resultieren, und kann zum Keimen eines völlig neuen Zyklus verwendet werden.
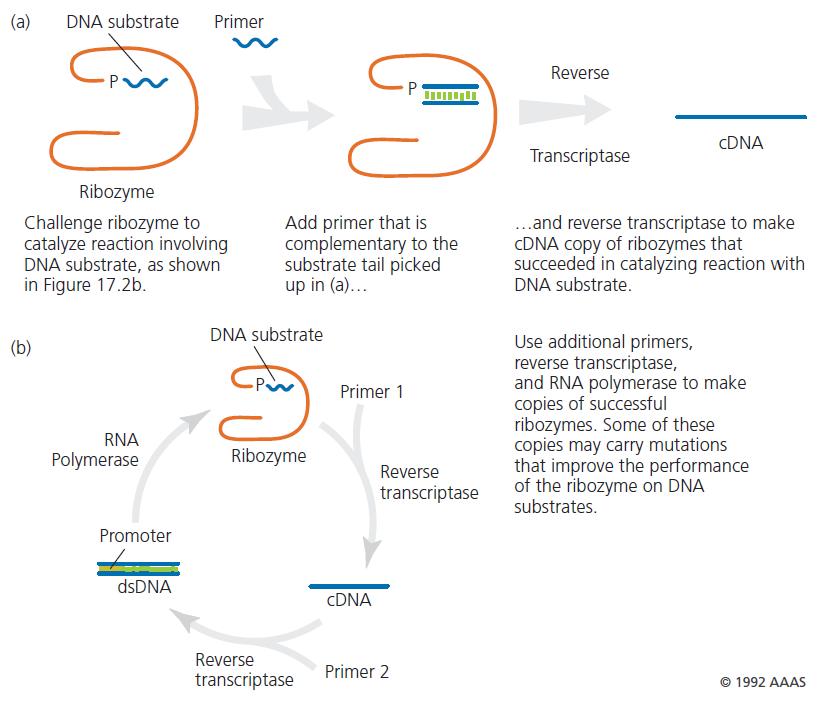
Abb. 6: Selektion und Vermehrung von RNA im Reagenzglas. (a) Auswahl. Ein Pool von RNAs (orange), die durch zufällige Mutagenese eines Ribozyms hergestellt wurden, wird aufgefordert, eine gewünschte chemische Reaktion durchzuführen. Nur diejenigen, die die Reaktion durchführen, erhalten einen kurzen „Schwanz“ von DNA-Nukleotiden an ihrem 3‘-Ende (blau). (b) Vermehrung. RNA-Sequenzen, die einen 3‘-Schwanz erworben haben (oben), binden Primer 1 durch komplementäre Basenpaarung und werden durch reverse Transkriptase in komplementäre DNA (cDNA) kopiert. Ein zweiter Primer bindet dann an die cDNA, so dass die reverse Transkriptase, die zusätzlich zur RNA auch DNA kopiert, die DNA doppelsträngig machen kann. Primer 2 enthält die Promotorregion für die RNA-Polymerase, so dass die doppelsträngige DNA wieder in RNA kopiert werden kann. Aus Beaudry & Joyce (1992).
Nach 10 solcher Generationen hatte sich die Aktivität der durchschnittlichen RNA in der Population um den Faktor 30 erhöht. Wichtig ist, dass diese phänotypische Verbesserung auf Veränderungen in der Nukleotidsequenz zurückführen sind (Abb. 7). Spezifische Mutationen an vier Nukleotidpositionen in der Sequenz des Ribozyms waren für den größten Teil der katalytischen Verbesserung verantwortlich. Einzelne Ribozyme, die Mutationen an den Positionen 94, 215, 313 und 314 trugen, wiesen eine mehr als 100-mal höhere katalytische Effizienz auf als die ursprüngliche Sequenz.
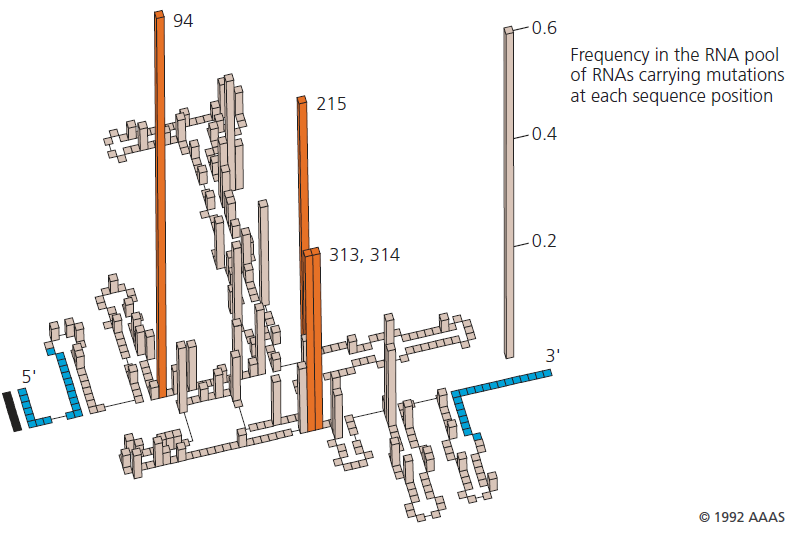
Abb. 7: Veränderungen der Allelhäufigkeit in einer sich entwickelnden RNA-Population. Dieses Histogramm, das auf einer groben Darstellung der gefalteten Sekundärstruktur des 413-Nukleotide umfassenden Tetrahymena-Ribozyms beruht, zeigt die Häufigkeit von Mutationen in der RNA-Population im Reagenzglas nach neun Selektions- und Reproduktionsrunden. Vier Mutationen, an den Nukleotiden 94, 215, 313 und 314, haben Häufigkeiten von über 50% erreicht und sind hauptsächlich für die neuen phänotypischen Eigenschaften der Population verantwortlich. Quelle: Beaudry & Joyce (1992).
Dieses Experiment hat gezeigt, dass RNA-Moleküle in Lösung Merkmale von lebenden Organismen aufweisen können, die es ihnen ermöglichen, sich weiterzuentwickeln. Jeder RNA kann eine bestimmte Fitness zugeschrieben werden, die sowohl vom Überleben als auch von der Vermehrung abhängig ist. Die Fitness des Moleküls ist ein Spiegelbild seines Phänotyps, der im Falle von Ribozymen unmittelbar durch ihre Primärsequenz spezifiziert ist. Es ist daher leicht, eine Parallele zwischen einer sich entwickelnden RNA-Population in einem Reagenzglas und einer sich entwickelnden Population moderner Organismen in der natürlichen Umgebung zu sehen (Lehman & Joyce 1993).
In Reagenzglas-Experimenten wie diesen haben Forscher viele Ribozyme mit entweder verbesserter oder völlig neuer Funktion entwickelt. Das katalytische Repertoire der RNA hat sich stark erweitert (Joyce 1998), und wir wissen jetzt, dass RNA Reaktionen wie Phosphorylierung (Lorsch & Szostak 1994), Aminoacyl-Transfer (Illangasekare et al. 1995), Bildung von Peptidbindungen (Zhang & Cech 1997) und Bildung von Kohlenstoff-Kohlenstoff-Bindungen (Tarasow et al. 1997; Fusz et al. 2005) katalysieren kann. Es wurden Ribozyme entwickelt, die allosterisch sind und zur Durchführung der Katalyse einen niedermolekularen Cofaktor benötigen (Tang & Breaker 1997). Es können Ribozyme ausgewählt werden, die eine Rolle bei der Ribonukleotidsynthese spielen (Unrau & Bartel 1998), die ihre Aktivität mit nur drei von vier Nukleotiden beibehalten (Rogers & Joyce 1999) und die ohne zweiwertige Metallionen-Cofaktoren arbeiten (Geyer & Sen 1997). RNA-Sequenzen, so genannte Aptamere, können so ausgewählt werden, dass sie sich eng an fast jedes beliebige andere Molekül binden (Ellington & Szostak 1990; Tuerk & Gold 1990), ähnlich wie die Immunglobulinproteine des Immunsystems von Säugetieren. Zusammengenommen deuten diese Entwicklungen darauf hin, dass die RNA ein mögliches lebendes System war, das den Zellen vorausging.
Selbst-Replikation
Nach dem, was wir bisher besprochen haben, fehlt ein entscheidender Teil des Beweises, dass die heutigen Organismen von Bewohnern einer RNA-Welt abstammen könnten. Wir wissen, dass die RNA ein vielseitiges Molekül ist und dass sie sich unter den richtigen Umständen entwickeln kann. In den von uns beschriebenen Experimenten wurde die RNA jedoch durch Proteinenzyme kopiert. Diese Proteine hätte es in der RNA-Welt nicht gegeben. Eine der Hauptprämissen der RNA-Welt-Hypothese ist, dass die RNA vor der Verwendung von Proteinen für die meisten biologischen Aufgaben existierte. Was uns fehlt, ist also der Nachweis, dass RNA sich selbst kopieren kann. Die „RNA-abhängige RNA-Autoreplikase“ bleibt ein schwer fassbarer Steinbruch für die Erforschung der Ursprünge des Lebens (Bartel & Unrau 1999; Müller 2006). Unabhängig davon, ob die RNA-Welt nur einen Typ selbstreplizierender RNA oder eine Reihe interagierender RNAs verwendete, wäre eine RNA mit einem Replikase-Phänotyp notwendig (Bartel 1999).
Die Hypothese, dass ein RNA-Molekül sich selbst replizieren und als einfacher Protoorganismus dienen könnte, ist überprüfbar. Wenn die Hypothese richtig ist, sollten wir in der Lage sein, ein sich selbst replizierendes RNA-Molekül im Labor herzustellen. Obwohl dies bis heute nicht gelungen ist, haben Forscher bedeutende Fortschritte gemacht. David Bartel und seine Mitarbeiter haben zum Beispiel mit Hilfe der Reagenzglas-Evolution nach Ribozymen gesucht, die RNA synthetisieren können (Bartel & Szostak 1993; Ekland et al. 1995, Abb. 8).
Die Forscher begannen mit einem großen Pool von RNA-Polynukleotiden. Jede Pool- hatte an ihren Enden die gleiche Sequenz (also am 3‘ und 5‘-Ende). Zwischen den Enden befand sich eine Sequenz von 220 Nukleotiden mit zufälliger Anordnung. Das 3‘-Ende der einzelnen Pool-RNAs verband sich mit Agarosekügelchen und wurde so fixiert. Anschließend wurde die fixierte Pool-RNA in Lösung mit Substrat-RNAs gegeben. Diese Substrat-RNAs banden sich an das freie 5‘-Ende der Pool-RNAs. Wenn der zufällige Teil der Sequenzen einer Pool-RNA über eine geeignete Ribozymaktivität verfügt, so bindet die Pool-RNA dauerhaft an das Substrat indem sie eine Phosphoesterbindung bildet. An ihrem 5’Ende hat die Substrat-RNA eine Markierungssequenz. Substrat-RNAs, die nicht durch Phosophoester-Bindungen gebunden sind, wurden weggespült, und die Pool-RNAs werden von ihren Agarosekügelchen freigesetzt. Schließlich ließen die Wissenschaftler die Pool-RNAs durch eine Affinitätssäule laufen. Die Affinitätssäule hielt die Markierungs-Sequenz auf der Substrat-RNA durch Basenpaarung fest. Somit fing die Säule jede Pool-RNA ein, deren 220-Nukleotid-Abschnitt der Zufallssequenz katalytische Aktivität aufwies. Nun lösten die Forscher die gefangenen Pool-RNAs von der Affinitätssäule, stellten viele Kopien von jeder her, indem sie Replikationsenzyme verwendeten, die einige Mutationen zuließen, und wiederholten den gesamten Vorgang. Die RNAs, die am ehesten von einer Generation zur nächsten überleben, sind diejenigen, die am effizientesten Phosphoesterbindungen katalysieren. Nach 10 Selektionsrunden hatte der RNA-Pool Ribozyme entwickelt, die die Bildung von Phosphoesterbindungen 7 Millionen Mal schneller katalysieren konnten als solche Bindungen ohne Katalysator (Abb. 9).
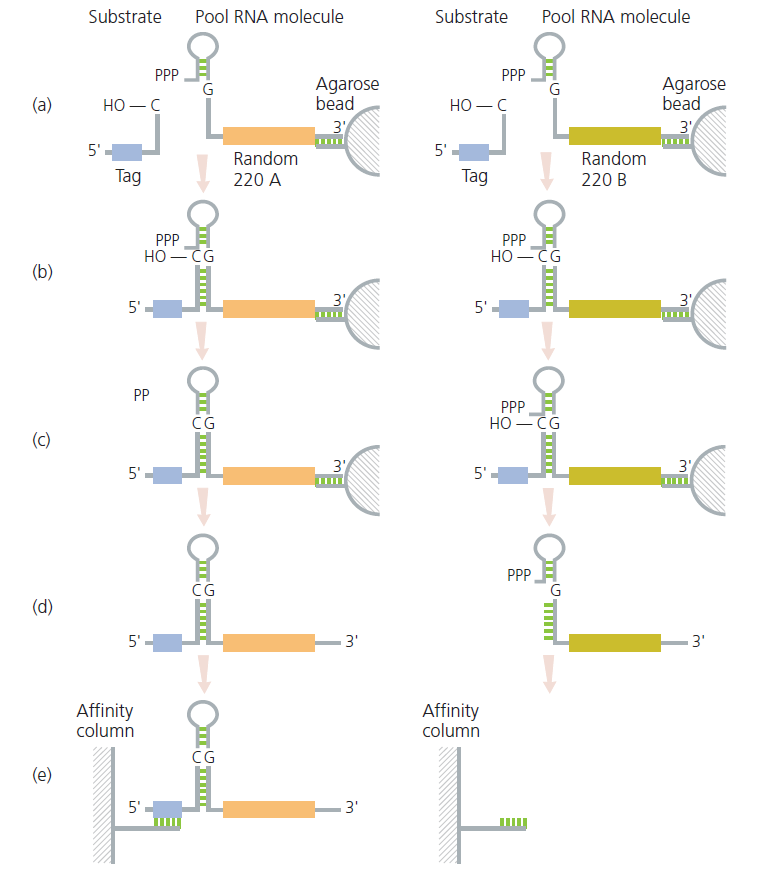
Abb. 8: Reagenzglas-Selektionsschema zur Identifizierung von Ribozymen, die RNA synthetisieren können. (a) Ein großer Pool von RNA-Molekülen mit zufälligen Sequenzen von 220 Nukleotiden wird durch Basenpaarung an Agarosekügelchen gebunden. Kopien einer kurzen Substrat-RNA werden hinzugefügt. (b) Das Substrat bindet durch Basenpaarung an das freie Ende der Pool-RNAs. (c) Wenn der zufällige Teil der Sequenz einer Pool-RNA eine geeignete Ribozymaktivität verleiht, bindet die Pool-RNA das Substrat dauerhaft an ihr eigenes freies Ende, indem sie eine Phosphoesterbindung bildet. (d) Substrat-RNAs, die nicht durch Phosphoesterbindungen gebunden sind, werden weggewaschen, und die Pool-RNAs werden von ihren Beads freigesetzt. (e) Die Pool-RNAs werden durch eine Affinitätssäule geleitet, die sie durch Basenpaarung mit einem Tag am freien Ende des Substrats einfängt. Nur Pool-RNAs mit gebundenem Substrat werden aufgefangen. Nach Bartel & Szostak (1993).
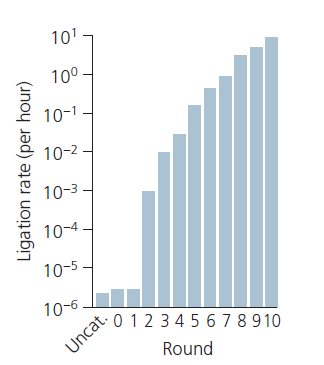
Abb. 9: Evolution der katalytischen Fähigkeit in einer Laborpopulation von Ribozymen. Das Diagramm zeigt die durchschnittliche Rate, mit der die Mitglieder des RNA-Pools von Bartel & Szostak (1993) die Bildung von Phosphoesterbindungen (Ligationsrate) katalysierten, als Funktion der Selektionsrunde. Beachtet die logarithmische Skala auf der vertikalen Achse. Aus Bartel & Szostak (1993).
Eine Arbeit von Wochner et al. (2011) führte ein vergleichbares Experiment mit anderen Ribozymen durch. Die Forscher begannen mit R18, einem von Johnston et al. (2001) erzeugten Ribozym, das eine RNA-Vorlage verwenden kann, um 14 RNA-Nukleotide an einen RNA-Primer anzuhängen. Die Forscher unterzogen eine Population von R18-Kopien einer Selektion auf die Fähigkeit, längere RNAs zu synthetisieren. Sie identifizierten bestimmte Sequenzveränderungen, die für die verbesserte Leistung verantwortlich waren, und bauten dann diese vorteilhaften Mutationen mit Hilfe der Gentechnik in R18 ein. Das Ergebnis war ein neues Ribozym, tC19, das 95 Nukleotide lange RNA-Moleküle synthetisieren kann – fast die Hälfte der Größe von tC19 selbst. Die Wissenschaftler unterzogen eine zweite Population von R18-Kopien einer Selektion auf die Fähigkeit, eine größere Vielfalt von Vorlagesequenzen zu kopieren, identifizierten Mutationen, die eine verbesserte Leistung bewirken, und bauten sie in tC19 ein. Das Endergebnis, tC19Z, war in der Lage, mit 99,8 %iger Genauigkeit funktionelle Kopien eines anderen Ribozyms zu synthetisieren (Abb. 10 & 11). Das Ribozym tC19Z ist nicht zur Selbstreplikation fähig. Aber es scheint, dass Biochemiker einer RNA-Sequenz oder einer Reihe von Sequenzen auf der Spur sind, die dies können (Lincoln & Joyce 2009).
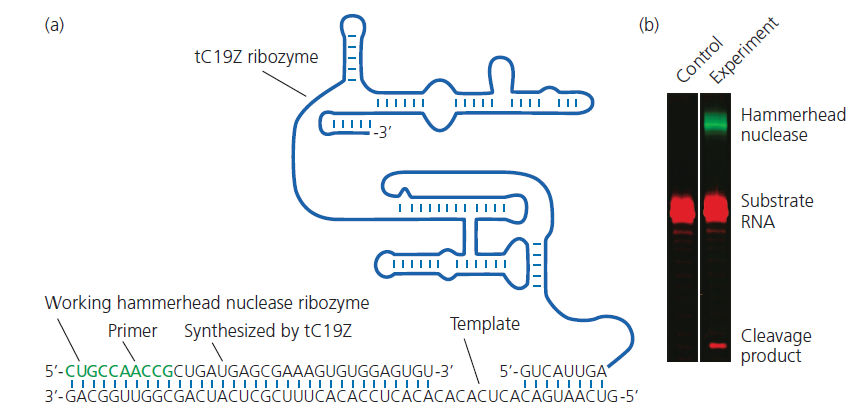
Abb. 10: Ein RNA-Polymerase-Ribozym, das ein anderes Ribozym synthetisieren kann. (a) Die Sekundärstruktur von tC19Z und die Vorlage für ein anderes Ribozym – eine so genannte Hammerhead-Nuklease -, die tC19Z mit Hilfe eines Primers kopieren kann. Nach Bentin (2011) und Wochner et al. (2011). (b) Elektrophorese-Gele, die zeigen, dass die von tC19Z synthetisierte Hammerhead-Nuklease ein RNA-Substrat spalten kann. Aus Wochner et al. (2011).
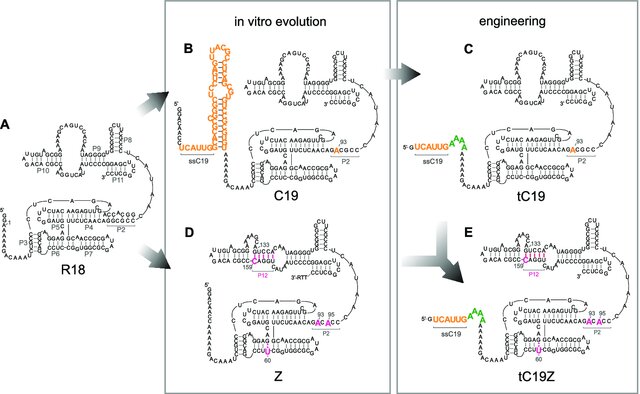
Abb. 11: Evolvierte und konstruierte Ribozyme. Sekundärstrukturen von (A) R18; (B) C19; (C) tC19; und vorgeschlagene Sekundärstrukturen für (D) Z; und (E) tC19Z. Die aus der Z-Selektion isolierten Mutationen sind in Magenta dargestellt, die aus der C19-Selektion isolierten Sequenzen in Orange und die konstruierten Reste in Grün. Aus Wochner et al. (2011).
2013 konnte der Nobelpreisträger Jack Szostak einen wichtigen Schritt zur Entstehung der ersten Protozellen experimentell nachvollziehen. Seinem Forscherteam gelang erstmals die Vervielfältigung von RNA innerhalb kleiner Fettsäure-Vesikeln (vgl. Adamala & Szostak 2013a, siehe auch Adamala & Szostak 2013b, Adamala et al. 2016, Izgu et al. 2016, Engelhart et al. 2016, Kamat et al. 2015, O’Flaherty et al. 2018, Abb. 12). Fettsäurevesikel sind für die Forschung deshalb so interessant, weil sie zum einen genau jene für die Replikation wichtige Reaktionsräume bieten und halbwegs selektiv nur bestimmten Molekülen den Durchtritt ins Innere gestatten. Dort können die erforderlichen Ausgangsstoffe in ausreichend hoher Konzentration zur Verfügung gestellt werden.
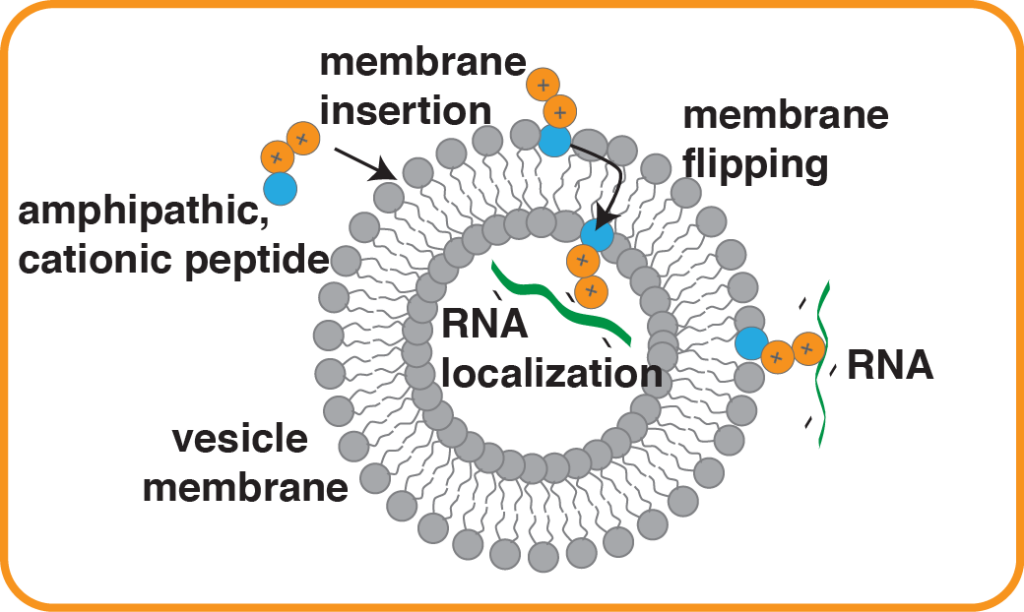
Abb. 12: In primitiven Zellen könnten Membranen eine katalytische Rolle gespielt haben, indem sie Reaktanten auf ihrer Oberfläche ko-lokalisierten. Zum Beispiel könnte die Lokalisierung von RNAs an Membranen den Zusammenbau von Ribozymen fördern und die Ribozymkatalyse erleichtern. Voraussetzung für ein solches Szenario ist ein einfacher Mechanismus zur Verankerung der RNA an der Protozellmembran. Studien haben weiterhin gezeigt, dass kurze, kationische, amphipathische Peptide die RNA-Bindung sowohl an zwitterionische Phospholipid- als auch an anionische Fettsäuremembranen fördern können. Die Assoziation dieser kationischen Peptide mit Phospholipidvesikeln kann die lokale positive Ladung einer Membran erhöhen und RNA-Polynukleotide anziehen. Bildquelle: https://molbio.mgh.harvard.edu/szostakweb/researchProtein.html
Die Hypothese von der Selbstvervielfältigung der RNA krankte aber auch daran, dass Magnesium-Ionen für den Kopiervorgang erforderlich sind (vgl. Adamala et al. 2016). Diese sind zwar im Meerwasser reichlich vorhanden, zerstören aber sowohl die einsträngige RNA als auch die Fettsäurehülle. Man konnte jedoch nachweisen, dass Citronensäure (Citrat) das Magnesium „maskiert“, sodass die ungünstigen Nebenreaktionen nicht mehr auftreten: Citrat bewahrt die Fettsäure-Membranen vor dem störenden Einfluss hoher Magnesiumkonzentrationen, ermöglicht zugleich die Vervielfältigung der RNA und schützt die einsträngige RNA vor dem durch Magnesium-Ionen beschleunigten Abbau. Die Teilung der Protozelle wird zwar von dem System nicht angestoßen, kann aber mechanisch (etwa durch Wellenbewegungen) erzwungen werden (vgl. O’Flaherty et al. 2018, Abb. 13).
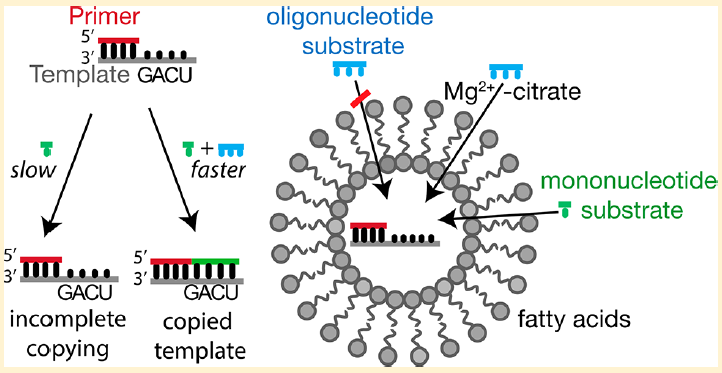
Abb. 13: Viele Jahre lang schien es eine grundlegende Inkompatibilität zwischen der RNA-Replikation, die hohe Mg2+-Konzentrationen erfordert, und Fettsäuremembranen zu geben, die durch moderate Mg2+-Konzentrationen zerstört werden. Man fand aber heraus, dass die Membranen geschützt sind, wenn Mg2+-Ionen durch Citrat chelatisiert werden, die RNA-Replikation aber trotzdem stattfinden kann. Dadurch konnten RNA-Kopierreaktionen in Fettsäurevesikeln durchgeführt werden, indem aktivierte Nukleotide an die Außenseite von Vesikeln gegeben wurden, die eingekapselte Primer-Template-Komplexe enthielten. Bildquelle: https://molbio.mgh.harvard.edu/szostakweb/researchVesicles.html
Allerdings bleiben spontan entstandene Lipidmembranen in der Regel nicht lange stabil. Da jedoch Ribozyme wiederum die kontrollierte Synthese von Lipidmembranen ermöglichen und zu deren Stabilisierung beitragen können, ist die Kombination von RNA und Lipidmembran von beider Vorteil; es entsteht eine Art der „Symbiose“, die zwangsläufig einen Evolutionsprozess in Gang setzt. Eine RNA, die sich rascher repliziert und die Fettsäurehülle besser stabilisiert, wird dadurch letztlich auch mehr Kopien von sich erzeugen.
Sowohl die Fettsäuren, wie auch die RNA können in den hydrothermalen Schloten entstanden sein. Der Geochemiker Mike Russel berichtete in einer Arbeit von 2007, dass sich große Mengen von Nukleotiden in Schloten ansammeln könnten (Baaske et al. 2007, Abb. 14). Der Grund dafür sind die ausgeprägten Temperaturgradienten, die sich dort entwickeln und die wir kennengelernt haben. Temperaturgradienten erzeugen zwei Arten von Strömungen, die durch die Poren zirkulieren: Konvektionsströme und Wärmediffusion. Dazwischen verfüllen diese beiden Wärmeströmungen die unteren Poren nach und nach mit vielen kleinen Molekülen, darunter auch Nukleotiden. Was noch besser ist: die Temperaturschwankungen treiben die RNA-Replikation ebenso an wie die bewährte Labormethode PCR.
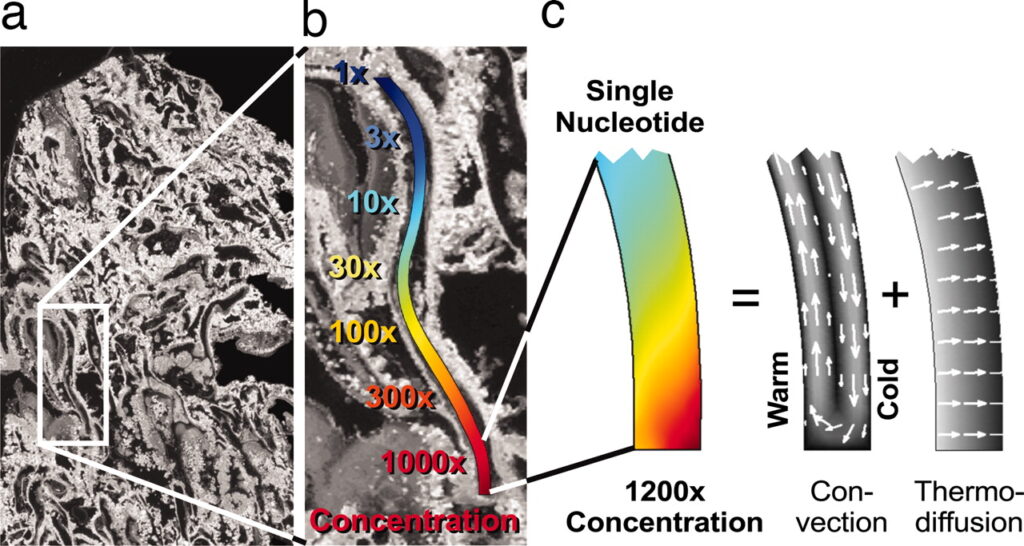
Abb. 14: Wärmegetriebene molekulare Akkumulation in hydrothermalen Poren. (a) Schnitt durch Aragonit (CaCO3) aus dem submarinen hydrothermalen Schlotfeld bei Lost City. (b) Simulation eines Teils des Porensystems. Bei einem horizontalen Temperaturgradienten von 30 K wird eine 1.200-fache Akkumulation einzelner Nukleotide erwartet (logarithmische Konzentrationsskala). Eine Verkettung von drei dieser Porenabschnitte führt zu einer 109-fachen Anreicherung. (c) Der Mechanismus der Akkumulation wird auf zweifache Weise durch Wärme angetrieben. Durch thermische Konvektion werden die Moleküle vertikal nach oben und unten transportiert, und durch Thermophorese werden die Moleküle horizontal nach rechts geschoben. Das Ergebnis ist eine starke molekulare Akkumulation von oben nach unten (lineare Konzentrationsskala).
Jedoch besteht das Problem, dass das Vorhandensein des schützenden Citrats auf der frühen Erde chemisch derzeit noch nicht plausibel gemacht werden. Andererseits ist Citrat ein wichtiges Molekül des Krebszyklus, einer wichtigen Drehscheibe des Stoffwechsels. Dessen Evolution wird in einem anderen Beitrag vorgestellt und macht dessen Existenz unter präbiotischen, hydrothermalen Bedingungen recht plausibel.
Weitere Studien zeigen außerdem, dass Modell-Protozellvesikel, die ein eingekapseltes Enzym enthalten, das die Synthese einfacher Fettsäurederivate fördert, durch Magnesium-Ionen stabilisiert werden, das für die Ribozymaktivität und RNA-Synthese erforderlich ist. Protozellen, die zu solchen katalytischen Umwandlungen fähig sind, hätten also in einer Umgebung mit hohem Magnesium-Gehalt einen Selektionsvorteil gegenüber anderen Protozellen (Adamala et al. 2016).
Interaktionen zwischen RNA und Vesikelmembranen auf der präbiotischen Erde könnten zudem zur Entstehung primitiver Zellen geführt haben, weil die Membranoberfläche Reaktionen katalysieren kann, an denen die RNA beteiligt ist. Aber dieses Szenario setzt voraus, dass es einen Mechanismus gibt, durch den sich RNA mit den Membranen von Urzellen verbinden kann. Eine Studie (Kamat et al. 2015) zeigt, dass elektrostatische Wechselwirkungen, die von kurzen, basischen Peptiden bereitgestellt werden, genutzt werden können, um die RNA-Bindung an Fettsäuremembranen anzutreiben (vgl. Abb. 12). So können die lokale positive Ladung einer Membran verstärkt werden und eine RNA elektrostatisch anziehen. Zudem können solche Peptide die Membran durchqueren, um eingekapselte RNA zu lokalisieren. Dieser Mechanismus des Einschlusses von Nukleinsäuren könnte für die primitive zelluläre Evolution von Bedeutung gewesen sein. Die physische Verbindung von RNA mit Vesikelmembranen könnte ein wichtiger erster Schritt zur Beschleunigung präbiotisch relevanter Reaktionen gewesen sein, einschließlich der RNA-Replikation und des Ribozymaufbaus, und könnte daher eine wichtige Rolle in frühen RNA-basierten Protozellen gespielt haben.
Zusätzlich zu diesen strukturellen Aufgaben können Peptide auch eine katalytische Rolle bei der Steuerung des Vesikelwachstums und ihrer Vermehrung spielen. Sie lokalisieren sich an der Membran von Protozellen und treiben das Vesikelwachstum voran und vermitteln zudem die Assoziation der Versikel mit RNA. Die Ergebnisse deuten auf einen möglichen Mechanismus hin, durch den aktivierte Aminosäuren die chemische Funktionalität von Fettsäuremembranen diversifizieren und RNA mit Vesikeln während der Bildung früher Protozellen kolokalisieren könnten (Izgu et al. 2016, Abb. 15).
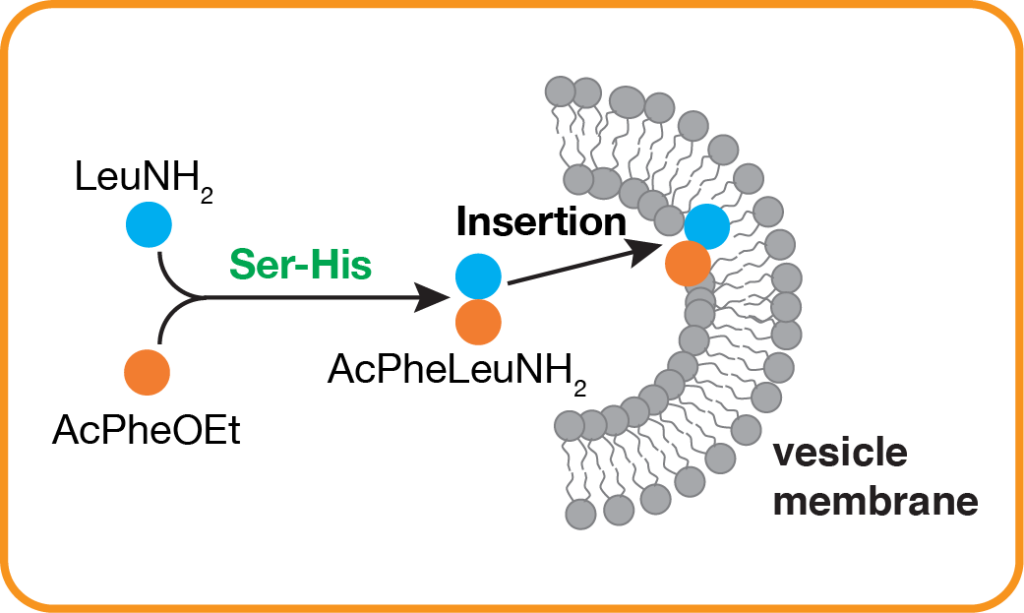
Abb. 15: Zusätzlich zu ihren strukturellen Aufgaben können Peptide auch eine katalytische Rolle bei der Steuerung des Vesikelwachstums spielen. Ein einfacher Dipeptid-Katalysator, hier Seryl-Histidin (Ser-His), kann das Vesikelwachstum durch die katalytische Synthese eines hydrophoben Dipeptids, N-Acetyl-L-Phenylalanin-Leucinamid, vorantreiben, das sich an der Membran von Modell-Protozellen lokalisiert und das kompetitive Vesikelwachstum in ähnlicher Weise vorantreibt, wie dies zuvor für Phospholipide gezeigt wurde.
Konkurrierende Protozellpopulationen, die jeweils aus einem sich replizierenden genetischen Polymer innerhalb eines replizierenden Vesikels bestehen, können evolvieren, wenn die molekularen Mechanismen eine vererbbare Variation der Fitness ermöglichen. Ein jedes genetisches Polymer in seiner Protozelle verschafft einen Selektionsvorteil, indem es beispielsweise für einen Katalysator kodiert, der einen nützlichen Metaboliten erzeugt. Eine Arbeitsgruppe berichtet über ein partielles Modell solcher im Entstehen begriffenen evolutionären Merkmale in einem System, das aus Fettsäurevesikeln besteht, die einen Dipeptidkatalysator enthalten, der die Bildung eines zweiten Dipeptids katalysiert. Das neu gebildete Dipeptid bindet an die Vesikelmembranen, was zu einer erhöhten Affinität für Fettsäuren führt und somit das Vesikelwachstum fördert. Die katalysierte Dipeptidsynthese verläuft in Vesikeln mit höherer Effizienz als in freier Lösung, was die Fitness weiter steigert. Die Beobachtungen legen nahe, dass in einer sich replizierenden Protozelle mit einem RNA-Genom die Ribozym-katalysierte Peptidsynthese ausgereicht haben könnte, um die darwinistische Evolution in Gang zu setzen (Adamala & Szostak 2013b).
Ein wichtiger Schritt in der präbiotischen Evolution ist, dass sich selbstreplizierende Moleküle zu komplexen lebenden Systemen entwickelt haben, indem sie ihre Informationen und Funktionen unbegrenzt erweiterten. Theoretisch könnte eine solche evolutionäre Komplexifizierung durch das sukzessive Auftreten neuartiger Replikatoren erfolgen, die miteinander interagieren und Replikationsnetzwerke bilden. Eine Studie aus dem Jahr 2022 führte Langzeitevolutionsexperimente mit RNA durch, die sich mit einer selbst kodierten RNA-Replikase repliziert (Mizuuchi et al. 2022, Abb. 16). Die RNA diversifiziert in mehrere koexistierende Wirts- und Parasitenlinien, deren Häufigkeiten in der Population zunächst schwanken und sich dann allmählich stabilisieren. Die endgültige Population, die fünf RNA-Stämme umfasst, bildet ein Replikator-Netzwerk mit vielfältigen Interaktionen, einschließlich der Zusammenarbeit zur Unterstützung der Replikation aller anderen Mitglieder (Abb. 17). Diese Ergebnisse belegen die Fähigkeit molekularer Replikatoren, durch Evolution spontan Komplexität zu entwickeln – ein entscheidender Schritt für die Entstehung von Leben. Kurzum: Ihre Arbeit zeigt, wie ein Molekül, das auch heute noch für das Überleben und die Fortpflanzung aller Lebewesen entscheidend ist, sich zu einem sich entwickelnden System entwickeln kann, wenn es als Team arbeitet. Das Problem war oft die Instabilität oder die Einfachheit einzelner RNA-Moleküle. Wenn jedoch mehrere replizierende auf Populationsebene interagieren ist das Informationsproblem zu lösen und die natürliche Auslese kann in Gang kommen. Das Forscherteam hat das richtige Design von RNA-Molekülen geknackt, um individuelle Replikatormoleküle zu schaffen, die gemeinsam nicht nur Informationen bewahren und sich im Laufe der Zeit verändern können, sondern dies auch so tun, dass die Lösung im Laufe aufeinander folgender Generationen komplexer wird.
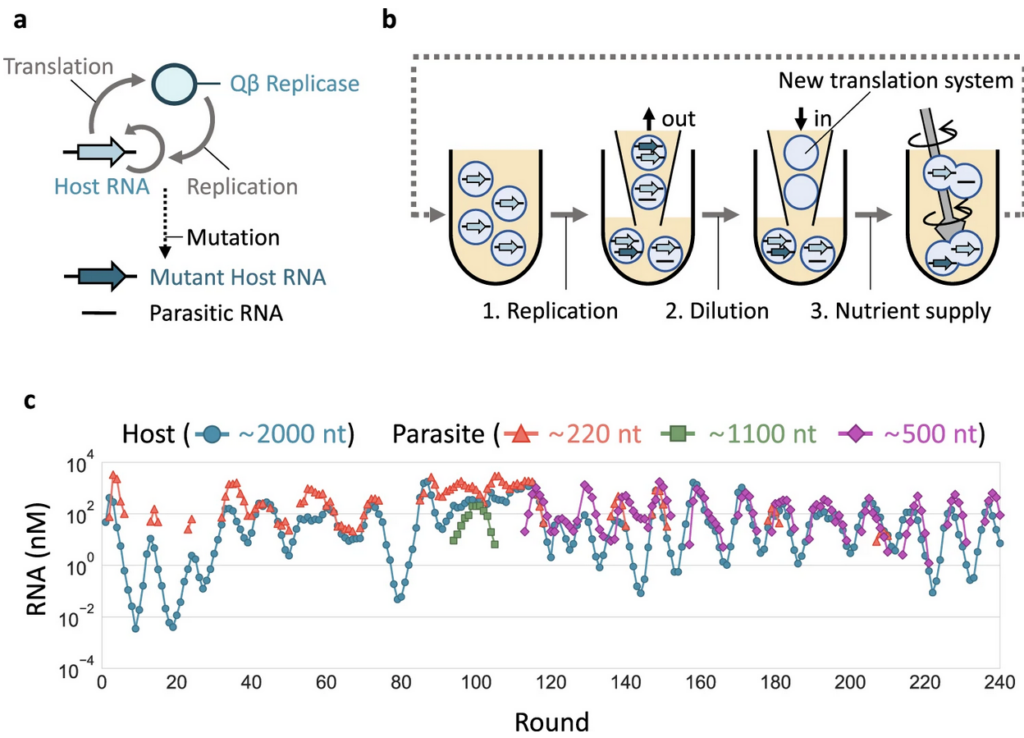
Abb. 16: a Das RNA-Replikationssystem. Die ursprüngliche Wirts-RNA repliziert durch Translation der selbst kodierten Replikase, durch die mutierte Wirts-RNAs und parasitäre RNAs erzeugt werden können. b Schematische Darstellung von Langzeit-Replikationsexperimenten in Wasser-in-Öl-Tröpfchen. (1) Die RNA-Replikation wurde 5 Stunden lang bei 37 °C durchgeführt. (2) Die Tröpfchen wurden 5-fach mit neuen Tröpfchen verdünnt, die das Übersetzungssystem enthielten. (3) Die Tröpfchen wurden kräftig gemischt, um ihre zufällige Fusion und Teilung zu induzieren. c Konzentrationsänderungen von Wirts- und Parasiten-RNAs unterschiedlicher Länge. Die Konzentrationen der Wirts-RNA wurden mittels RT-qPCR gemessen, und die Konzentrationen der parasitären RNA wurden anhand der entsprechenden Bandenintensitäten nach der Gelelektrophorese ermittelt. Die Konzentrationen der parasitären RNA wurden in Runden, in denen sie nicht nachweisbar waren, nicht aufgezeichnet. Quelle: Mizuuchi et al. (2022).
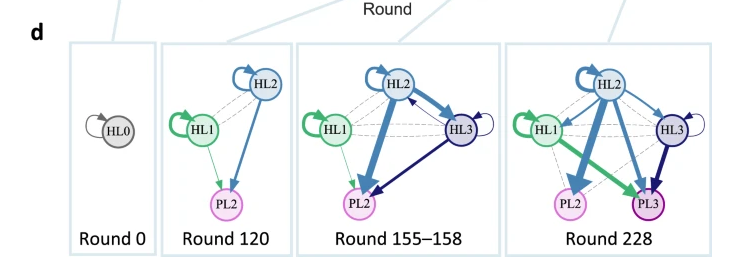
Abb. 17: Gerichtete Graphen zur Darstellung der interdependenten Replikation der RNA-Klone jeder ausgewählten Runde. Die Knoten stehen für die RNA-Klone der angegebenen Runden. Pfeile zeigen die direkte Replikation an, wobei die Breite proportional zum binären Logarithmus der gemessenen relativen Replikation ist. Graue gestrichelte Linien zeigen unentdeckte Replikationsereignisse von Wirts-RNA-Klonen an. Quelle: Mizuuchi et al. (2022).
Eine kürzlich erschienene Arbeit zeigt, wie sich ein entscheidendes RNA-Enzym, die RNA-Polymerase, evolutionär optimieren konnte (Papastavrou et al. 2024).[3] Erst diese Selbstoptimierung ermöglichte ein immer genaueres Kopieren des RNA-Erbguts und damit die zuverlässige Merkmalsweitergabe der ersten RNA-Zellen. Die RNA-Polymerase kann andere RNA-Moleküle kopieren und gilt damit als entscheidender Akteur bei der Vermehrung der frühen, RNA-basierten Lebensformen. Für ihre Studie verwendeten die Forscher zunächst eine sehr simple RNA-Polymerase, die nur kurze RNA-Stränge kopieren kann und dabei viele Fehler macht. Ausgehend davon wurde dieses RNA-Enzym dann schrittweise mit der Methode der gerichteten Evolution verändert. Durch Dutzende im Labor induzierte Runden der Mutation und Selektion entstanden so Enzymvarianten, die ihre Aufgabe immer besser und mit weniger Fehlern durchführen konnten. Untersucht wurde dabei auch, wie sich die Fehleranfälligkeit der Polymerase auf die kopierten Produkte auswirkte. Das Ergebnis: Ab einem gewissen Punkt war die RNA-Polymerase so weit optimiert, dass sie auch längere RNA-Moleküle zuverlässig kopierte. Es blieb jedoch eine Fehlerquote von etwa zehn Prozent, die Variationen und Mutationen im kopierten Objekt in geringem Ausmaß weiterhin zuließ. Die kopierte RNA-Sequenz veränderte sich dadurch im Zuge mehrerer Kopierrunden und häufte einige Mutationen an. Diese Mutationen waren jedoch offenbar sogar förderlich, sodass das RNA-Molekül seine eigene Funktion schließlich ebenfalls besser erfüllen konnte. Die Studie liefert damit weitere Indizien dafür, dass auf der Urerde tatsächlich einst eine „RNA-Welt“ existierte, in der die Evolution von RNA-Enzymen und anderen RNA-Molekülen parallel verlief. Ohne RNA-Polymerasen mit einer hohen Genauigkeit und geringen Fehlertoleranz wären demnach keine höher entwickelten RNA-Moleküle entstanden. In der Folge hätte es auch keine weiterentwickelte Zellen mit komplexer Zellmaschinerie und DNA-Erbgut gegeben und letztlich auch keine höheren Lebewesen.
Die RNA ist seit langem einer der Spitzenreiter bei der Suche nach den ersten Replikationssystem. Sie ist heute in der gesamten Biosphäre allgegenwärtig, könnte durch nicht-biologische Prozesse bereits auf der Urerde vorhanden gewesen sein, kann eine große Menge an Informationen speichern und fungiert als dynamische physikalische Einheit. Das bedeutet, dass es potenziell Strukturen bilden kann, die physisch neue Moleküle aufbauen können, die wiederum neue Strukturen bilden können. Wenn dieser Prozess unvollkommen ist, werden einige der “Replikator”-Strukturen die Aufgabe schneller oder effizienter erledigen als andere und zur dominierenden Form der RNA werden … zumindest, bis etwas noch Besseres auftaucht, wie z. B. die DNA.
Gerald Joyce (1996) drückte es wie folgt aus: „Sobald ein RNA-Enzym mit RNA-Replikase-Aktivität in der Hand ist, hört das Träumen auf und der Spaß beginnt.“ Es sind noch längst nicht alle Fragen der RNA-Welt geklärt und die Forschung wird weitergehen. Uns interessiert aber: Ist ein Übergang von der RNA- zur DNA-Welt überhaupt möglich? Denn alle Lebewesen speichern ihr Genom in Form von DNA. Lediglich einige Viren haben RNA als alleinigen Informationsspeicher. Die ganze RNA-Welt-Hypothese steht und fällt mit der Möglichkeit eines Übergangs zu DNA-Welt. Würde eine Spezies selbstreplizierender RNA ein DNA-Genom mit DNA-Replikation und Transkription hervorbringen? Wie beginnt die RNA Proteine zu synthetisieren und DNA als Speichermedium zu nutzen? Mögliche Antworten dazu kommen im nächsten Beitrag.
Literatur
Adamala KP, Engelhart AE, Szostak JW (2016): Collaboration between primitive cell membranes and soluble catalysts. Nat Commun. 7:11041. doi: 10.1038/ncomms11041.
Adamala K, Szostak JW. (2013a): Nonenzymatic template-directed RNA synthesis inside model protocells. Science. 342(6162):1098-100.
Adamala K, Szostak JW (2013b): Competition between model protocells driven by an encapsulated catalyst. Nat Chem. 5(6):495-501. doi: 10.1038/nchem.1650.
Altman, S, Baer, M F, Bartkiewicz, M, Gold, H, Guerrier-Takada, C, Kirsebom, LA, Lumelsky, N, Peck, K (1989): Catalysis by the RNA subunit of RNase P—a minireview“. Gene. 82 (1): 63–4 b
Baaske P, Weinert FM, Duhr S, Lemke KH, Russell MJ, Braun D. (2007): Extreme accumulation of nucleotides in simulated hydrothermal pore systems. Proc Natl Acad Sci U S A. 29,104(22):9346-51.
Bartel, D. P. (1999): Recreating an RNA replicase. In The RNA World (2nd ed.), ed. R. F. Gesteland, T. R. Cech, and J. F. Atkins. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 143–162.
Bartel, D. P., Szostak, J. W. (1993): Isolation of new ribozymes from a large pool of random sequences. Science 261: 1411–1418.
Bartel, D. P., Unrau, P. J. (1999): Constructing an RNA World. Trends in Cell Biology 9: M9–M13.
Beaudry, A. A., Joyce, G. F. (1992): Directed evolution of an RNA enzyme. Science 257: 635–641.
Bentin, T. (2011): A ribozyme transcribed by a ribozyme. Artificial DNA, PNA & XNA 2: 40.
Campbell, N. A., et al. (2014): Campbell Biologie, 10., aktualisierte Auflage
Crick, F. H. C. (1966): The genetic code—yesterday, today, and tomorrow. Cold Spring Harbor Symposia on Quantitative Biology 31: 1–9.
Ekland, E. H., Szostak, J. W., Bartel, D. P. (1995): Structurally complex and highly active RNA ligases derived from random RNA sequences. Science 269: 364–370.
Ellington, A. D., Szostak, J. W. (1990): In vitro selection of RNA molecules that bind specific ligands. Nature 346: 818–822.
Engelhart, A., Adamala, K., Szostak, J. (2016): A simple physical mechanism enables homeostasis in primitive cells. Nature Chem 8, 448–453
Fusz, S., Eisenführ, A. et al. (2005): A ribozyme for the aldol reaction. Chemistry & Biology 12: 941–950.
Gesteland, R. F., Cech, T. R., Atkins, J. F. (eds. 1999): The RNA World. 2nd ed. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.
Geyer, C. R., Sen, D. (1997): Evidence for the metal-cofactor independence of an RNA phosphodiester-cleaving DNA enzyme. Chemistry & Biology 4: 579–593.
Gilbert, W. (1986): The RNA world. Nature 319: 618.
Harris, J. K., Kelley, S. T., et al. (2003): The genetic core of the universal ancestor. Genome Research 13: 407–412.
Illangasekare, M., Sanchez, G. et al. (1995): Aminoacyl-RNA synthesis catalyzed by an RNA. Science 267: 643–647.
Izgu EC, Björkbom A, Kamat NP, Lelyveld VS, Zhang W, Jia TZ, Szostak JW (2016): N-Carboxyanhydride-Mediated Fatty Acylation of Amino Acids and Peptides for Functionalization of Protocell Membranes. J Am Chem Soc. 138(51):16669-16676. doi: 10.1021/jacs.6b08801.
Johnston, W. K., Unrau, P. J. et al. (2001): RNA-catalyzed RNA polymerization: Accurate and general RNA-templated primer extension. Science 292: 1319–1325.
Joyce, G. F. (1989): RNA evolution and the origins of life. Nature 338: 217–224.
Joyce, G. F. (1996): Ribozymes: Building the RNA world. Current Biology 6: 965–967.
Joyce, G. F. (1998): Nucleic acid enzymes: Playing with a fuller deck. Proceedings of the National Academy of Sciences, USA 95: 5845–5847.
Kamat NP, Tobé S, Hill IT, Szostak JW (2015): Electrostatic Localization of RNA to Protocell Membranes by Cationic Hydrophobic Peptides. Angew Chem Int Ed Engl. 54(40):11735-9. doi: 10.1002/anie.201505742.
Koonin, E. V. (2003): Comparative genomics, minimal gene-sets, and the last universal common ancestor. Nature Reviews Microbiology 1: 127–136.
Kruger K, Grabowski PJ, Zaug AJ, Sands J, Gottschling DE, Cech TR (1982): Self-splicing RNA: autoexcision and autocyclization of the ribosomal RNA intervening sequence of Tetrahymena. Cell. 31 (1): 147–157.
Lehman, N., Joyce, G. F. (1993): Evolution in vitro of an RNA enzyme with altered metal dependence. Nature 361: 182–185.
Lincoln, T. A., Joyce, G. F. (2009): Self-sustained replication of an RNA enzyme. Science 323: 1229–1232.
Lorsch, J. R., Szostak, J. W. (1994): In vitro evolution of new ribozymes with polynucleotide kinase activity. Nature 371: 31–36.
Mills, D. R., Peterson, R. L., Spiegelman, S. (1967): An extracellular Darwinian experiment with a self-duplicating nucleic acid molecule. Proceedings of the National Academy of Sciences, USA 58: 217–220.
Mizuuchi, R., Furubayashi, T., Ichihashi, N. (2022): Evolutionary transition from a single RNA replicator to a multiple replicator network. Nat Commun 13, 1460
Müller, U. F. (2006): Re-creating an RNA world. Cellular and Molecular Life Sciences 63: 1278–1293.
Nissen, P., Hansen, J., et al. (2000): The structural basis of ribosome activity in peptide bond synthesis. Science 289: 920–930.
O’Flaherty DK, Kamat NP, Mirza FN, Li L, Prywes N, Szostak JW. (2018): Copying of Mixed-Sequence RNA Templates inside Model Protocells. J Am Chem Soc. 140(15):5171-5178. doi: 10.1021/jacs.8b00639.
Papastavrou N, Horning DP, Joyce GF. (2024): RNA-catalyzed evolution of catalytic RNA. Proc Natl Acad Sci U S A. 121(11):e2321592121.
Rogers, J., Joyce, G. F. (1999): A ribozyme that lacks cytidine. Nature 402: 323–325.
Sadava, D., Hillis, D., Heller, H. C., Hacker, S. (2019): Purves, Biologie, 10. Auflage, Springer-Verlag Berlin Heidelberg.
Steitz, T. A., Moore, P. B. (2003): RNA, the first macromolecular catalyst: The ribosome is a ribozyme. Trends in Biochemical Sciences 28: 411–418.
Tang, J., Breaker, R. R. (1997): Rational design of allosteric ribozymes. Chemistry and Biology 4: 453–459.
Tarasow, T. M., Tarasow, S. L., Eaton, B. E. (1997): RNA-catalyzed carbon–carbon bond formation. Nature 389: 54–57.
Tuerk, C., Gold, L. (1990): Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science 249: 505–510.
Unrau, P. J., Bartel, D. P. (1998): RNA-catalyzed nucleotide synthesis. Nature 395: 260–263.
Visser CM (1984): Evolution of biocatalysis 1. Possible pre-genetic-code RNA catalysts which are their own replicase. Origins of Life. 14 (1–4): 291–300.
White, H. B. (1976): Coenzymes as fossils of an earlier metabolic state. Journal of Molecular Evolution 7: 101–104.
Wochner, A., Attwater, J. et al. (2011): Ribozyme-catalyzed transcription of an active ribozyme. Science 332: 209–212.
Woese C (1967): The Genetic Code. New York: Harper and Row.
Zaug, A. J., Cech, T. R. (1986): The intervening sequence RNA of Tetrahymena is an enzyme. Science 231: 470–475.
Zhang, B., Cech, T. R. (1997): Peptide bond formation by in vitro selected ribozymes. Nature 390: 96–100.
[1] https://internet-evoluzzer.de/synthese-von-nukleinsaeuren-zuckern-fetten/
[2] https://internet-evoluzzer.de/polymerisation-von-biomolekuelen/
[3] Siehe auch: https://www.scinexx.de/news/biowissen/neuer-einblick-in-die-rna-welt/