Die RNA-Welt und Spiegelmans Monster
Text als pdf
Mitte der 1980er Jahre machte man die überraschende Entdeckung, dass RNA sich wie ein Katalysator verhält. RNA bildet nur selten eine Doppelhelix, stattdessen aber kleiner kompliziert geformte Moleküle, die sich gegenseitig einander zur Katalyse bereitstellen. In einer hypothetischen RNA-Welt übernimmt diese gleichzeitig die Rolle der Proteine und der DNA und katalysiert, neben vielen anderen Reaktionen, ihre eigene Synthese (Gilbert 1986, Joyce 1989; Abb. 1). Aber kann die RNA mit Proteinen agieren? Wie Entstand der Weg der Informationsspeicherung von der RNA zur DNA und wie entwickelte sich der genetische Code?
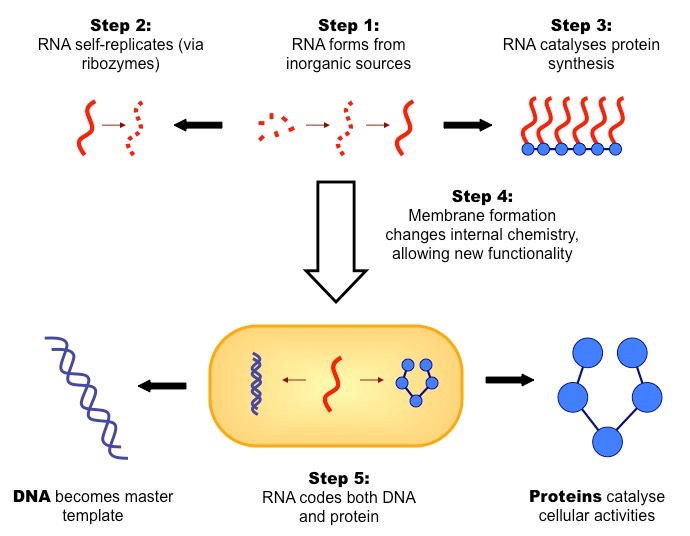
Abb. 1: RNA-Welt-Hypothese
Geben wir RNA zusammen mit nötigen Rohmaterialien und Energie (ATP) in ein Reagenzglas, wird sie sich replizieren. Tatsächlich wird sie sich nicht nur replizieren, sondern sie wird entstehen, wie der Molekularbiologe Sol Spiegelman und seine Kollegen in den 1960ern herausfanden (Spiegelman et al. 1965, Mills et al. 1967; Abb. 2). Über Reaganzglasgenerationen hinweg repliziert sich die RNA schneller und schneller und wird letztendlich enorm effizient. Egal wie die Ausgangsbeschaffenheiten sind, es besteht immer die gleiche Tendenz einen sich replizierenden RNA-Stand mit 50 Buchstaben zu erzeugen. Aber diese RNA-Moleküle werden nicht komplexer. Der Grund weshalb sie auf einer Strecke von 50 Buchstaben stehen bleiben ist, dass dies die Bindungssequenz für das Replikase-Enzym ist, ohne dass sich ein Strang nicht replizieren könnte. Die RNA kann gewissermaßen nicht über ihren eigenen Tellerrand schauen und wird in einer Lösung niemals Komplexität entwickeln. Wie und warum begann die RNA nun, auf Kosten ihrer eigenen Replikationsgeschwindigkeit, Proteine zu codieren? Der einzige Ausweg aus diesem Teufelskreis ist, dass die Selektion auf einer höheren Stufe erfolgt und die RNA Teil eines größeren Ganzen wird, dass nun die Einheit der Selektion darstellt – beispielsweise eine Zelle. Das Problem besteht aber auch hier, dass eine Zelle viel zu komplex ist, um einfach so, ohne Evolution, in Erscheinung zu treten, was bedeutet, dass für die Merkmale einer Zelle, viel mehr noch als für die Replikationsgeschwindigkeit der RNA, Selektion eintreten muss.
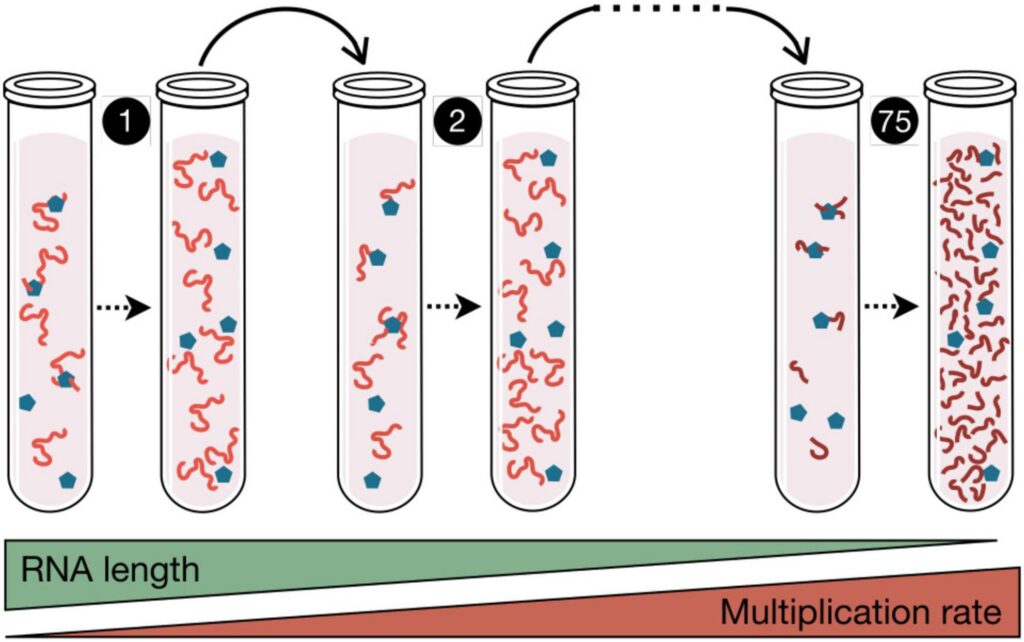
Abb. 2: Spiegelmans Monster. In diesem 1967 veröffentlichten Experiment wurden die RNA des Phagen Qβ (rote Linien) und das für seine Vervielfältigung verantwortliche Enzym (blaue Fünfecke) in Gegenwart von Nukleotiden inkubiert und einer In-vitro-Replikation unterzogen. Die Produkte jeder Polymerisationsreaktion wurden nacheinander in neue Röhrchen übertragen. Nach 75 dieser Transfers waren 83 % des RNA-Genoms eliminiert, was wiederum die Vermehrungsrate der RNA-Population erhöhte. Diese bahnbrechende Arbeit von Mills et al. (1967) zeigte, dass die darwinistische Evolution in einem Reagenzglas stattfinden kann. Ähnliche Experimente mit seriellen Transfers werden bei der Untersuchung der Virusevolution eingesetzt.
Sind RNA-Moleküle in großer Menge vorhanden, verbinden sich die Nukleotide spontan zu langen Ketten. Die ist Konzentration niedrig, zerfällt die RNA wieder in die Nukleotide. Das Problem besteht darin, dass eine RNA jedes Mal, wenn sie sich selbst repliziert, Nukleotide aufnimmt und so deren Konzentration verringert. Sofern der Pool mit Nukleotiden nicht kontinuierlich aufgefüllt wird, und zwar schneller als er ausgebraucht wird, könnte die RNA-Welt niemals funktionieren. Wie kann eine RNA-Welt also funktionieren? Die Antwort sticht uns direkt ins Auge: es sind die gebrauchsfertigen anorganischen Zellen in den Hydrothermalquellen, die wir kennengelernt haben.
Der Geochemiker Mike Russel berichtete in einer Arbeit von 2007 (Baaske et al. 2007), dass sich große Mengen von Nukleotiden in Schloten ansammeln könnten. Der Grund dafür sind die ausgeprägten Temperaturgradienten, die sich dort entwickeln und die wir kennengelernt haben. Temperaturgradienten erzeugen zwei Arten von Strömungen, die durch die Poren zirkulieren: Konvektionsströme und Wärmediffusion. Dazwischen verfüllen diese beiden Wärmeströmungen die unteren Poren nach und nach mit vielen kleinen Molekülen, darunter auch Nukleotiden. Was noch besser ist: die Temperaturschwankungen treiben die RNA-Replikation ebenso an wie die bewährte Labormethode PCR. Längere RNA- oder DNA-Moleküle häufen sich theoretisch stärker an als einzelne Nukleotide. Mit ihrer größeren Größe sind sie besser dazu geeignet, die Poren zu verfüllen.
Solche anorganischen Schlot-Zellen besitzen dieselbe Größe wie organische Zellen und werden in den Schloten ständig gebildet. Sind die Organellen einer Zelle also besonders gut darin, die Rohmaterialien zu erneuern, die für ihre eigene Replikation benötigt werden, beginnt die Zelle sich selbst zu replizieren und bildet Knospen neuer anorganischer Zellen. Im Gegensatz werden dazu „eogistische“ RNAs, die sich so schnell wie möglich selbst replizieren, verdrängt, da sie nicht in der Lage sind, die Rohmaterialien zu erneuern, die sie zur Fortsetzung ihrer eigenen Replikation brauchen. Mit anderen Worten verschiebt sich in der Umgebung der Schlote die Selektion graduell von der Replikationsgeschwindigkeit einzelner RNA-Moleküle hin zum allgemeinen Stoffwechsel der Zellen, die als individuelle Einheiten fungieren. Und vor allen anderen sind die Proteine Meister des Stoffwechsels. Es war unvermeidlich, dass sie die RNA ersetzen würden. Selbstverständlich entstanden Proteine nicht ganz plötzlich, wahrscheinlich trugen Minerale, Nukleotide, RNAs, Aminosäuren und Molekülkomplexe zum Prototyp des Stoffwechsels bei. Was als einfache Verschmelzung von Molekülen begann, wurde in dieser Welt aus sich natürlich vermehrenden Zellen zur Selektion der Fähigkeit, die Bestandteile ganzer Zellen zu erneuern. Es wurde zur Selektion der Selbstversorgung und letztlich eines unabhängigen Daseins. Und ironischerweise finden wir in dem unabhängigen Dasein der heutigen Zellen den letzten Schlüssel zum Ursprung der DNA.
Von der RNA-Welt zur DNA-Welt
Warum ist aber die DNA überhaupt entstanden? Argumentiert wird, dass die RNA durch DNA ersetzt wurde, weil letztere stabiler ist und zuverlässiger repliziert werden kann. Das ermöglicht die Evolution größerer Genome, die für mehr und komplexere Proteine codieren (Poole et al. 2001, Lazcano et al. 1988). Ein solcher Selektionsdruck konnte jedoch nur im Rahmen des Wettbewerbs zwischen Populationen von RNA- und DNA-Organismen wirken. Um die Vorteile eines DNA-Genoms in Bezug auf Stabilität zu nutzen, hätten diese ersten Organismen bereits über effiziente DNA-Reparatur- und Replikationssysteme verfügen müssen, wie z. B. einen spezifischen Mechanismus zur Entfernung von Uracil aus der DNA (Poole et al. 2000).
Betrachten wir den Stammbaum des Lebens, so erkennen wir, dass die drei Domänen des Lebens: Bakterien, Archaeen und Eukaryoten (Woese et al. 1990) viele Gemeinsamkeiten in der Proteinbiosynthese haben: genetischer Code, Transkription, Translation und so weiter verlaufen ähnlich bzw. lassen sich homologisieren. Aber es gibt gleichzeitig einen tiefen Riss zwischen Bakterien und Archaeen: sie lernten völlig unabhängig voneinander ihre DNA zu vervielfältigen (Edgell & Doolittle 1997, Leipe et al. 1999, Forterre 1999, 2001, 2002, 2005, 2013, Koonin et al. 2020, Abb. 3). Anders ausgedrückt: Viele Proteine, die am DNA-Stoffwechsel beteiligt sind erfüllen ähnliche Funktionen, sind aber nicht zu einender homolog, sind also unabhängig voneinander entwickelt worden (Arezi & Kuchta 2000, Filee et al. 2002, Steitz 1999, Buhler et al. 2001, Champoux 2001, Keck et al. 2001, Nichols et al. 1999, Garcia et al. 2000, Lakshminarayan et al. 2001, Leipe et al. 2000, Yoshida et al. 1983).
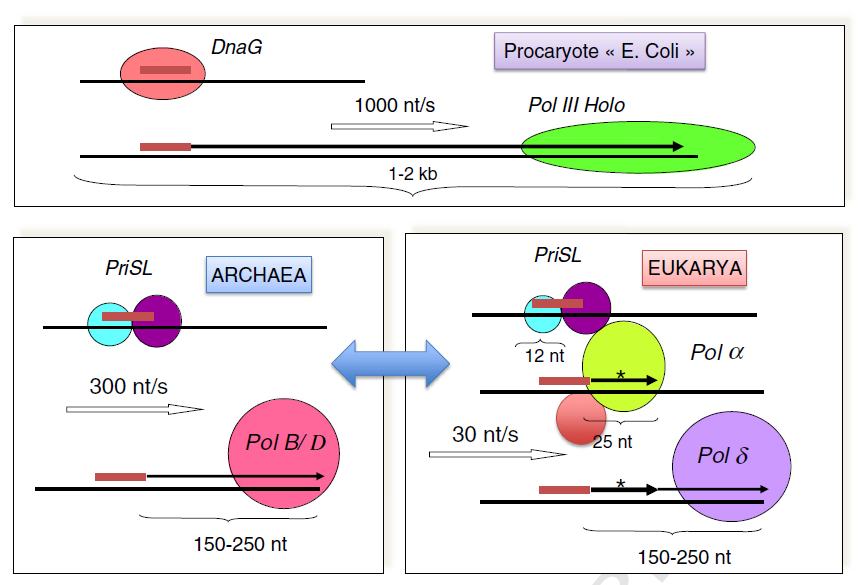
Abb. 3: schematische Darstellung der Replikationssysteme bei den drei Domänen im Hinblick auf die Synthese der Okazaki-Fragmente.
Bemerkenswert ist zudem, dass die meisten DNA-Replikationsproteine zwischen Archaeen und Eukaryoten zueinander homolog sind. Diese phylogenetische Verbindung zwischen den Replikationsmechanismen von Archaeen und Eukaryonten ist umso bedeutsamer, als sich der Replikationsmodus und die Chromatinstruktur, die in Archaea und Bakterien ähnlich sind, in Archaea und Eukarya (Matsunaga et al. 2001, Myllykallio et al. 2000) unterscheiden. Eukaryoten entstanden aus der Verschmelzung einer urtümlichen Archaeenzelle mit einem Bakterium – letzterer wurde dann zu den Mitochondrien. Ein Mechanismus, der unter der Endosymbiontentheorie zusammengefasst wird und wir sehr bald behandeln werden.
Unsere vorherigen Beiträge haben gezeigt, dass sich LUCA bildete, bevor sich Bacteria und Archaea voneinander divergierten und als freilebende Zellen lebten (Koga et al. 1998, Martin & Russel 2003). Folgerichtig haben sie auch ein eigenes Replikationssystem für ihre Genome entwickelt.
Die Replikation der DNA, der physikalische Mechanismus der Vererbung in allen lebenden Zellen – entwickelte sich zweimal (Edgell & Doolittle 1997, Leipe et al. 1999, Forterre 1999, 2001, 2002, 2005, 2013, Koonin et al. 2020). Trotz ihrer Unterschiede in der Replikation sprechen die Gemeinsamkeiten in der Proteinsynthese dafür, dass sie auf einen gemeinsamen Vorfahren zurückgehen. Wieso entwickelte sich dann aber die Vermehrung der DNA unabhängig voneinander?
Bill Martin und Mike Russel verfassten 2003 (Martin & Russel 2003, 2007, Martin et al. 2014, Lane 2010, 2017, Lane & Martin 2012, Lane et al. 2010) hierfür ihre eigene Theorie, die wir ebenfalls kennengelernt haben: der gemeinsame Vorfahre von Bakterien und Archeen war keineswegs ein freilebender Organismus, sondern ein mittelmäßiger Replikator, der an löchriges Felsgestein gebunden war und die mineralischen Zellen, die die heißen Hügel durchsiebten, noch nicht verlassen hatte. So zeigt sich z. B., dass beide Gruppen völlig verschiedene Zellmembranen und Zellwände haben, was darauf hindeutet, dass sie diesen Unterschied unabhängig voneinander innerhalb derselben steinigen Grenzen entwickelt haben (Abb. 4).
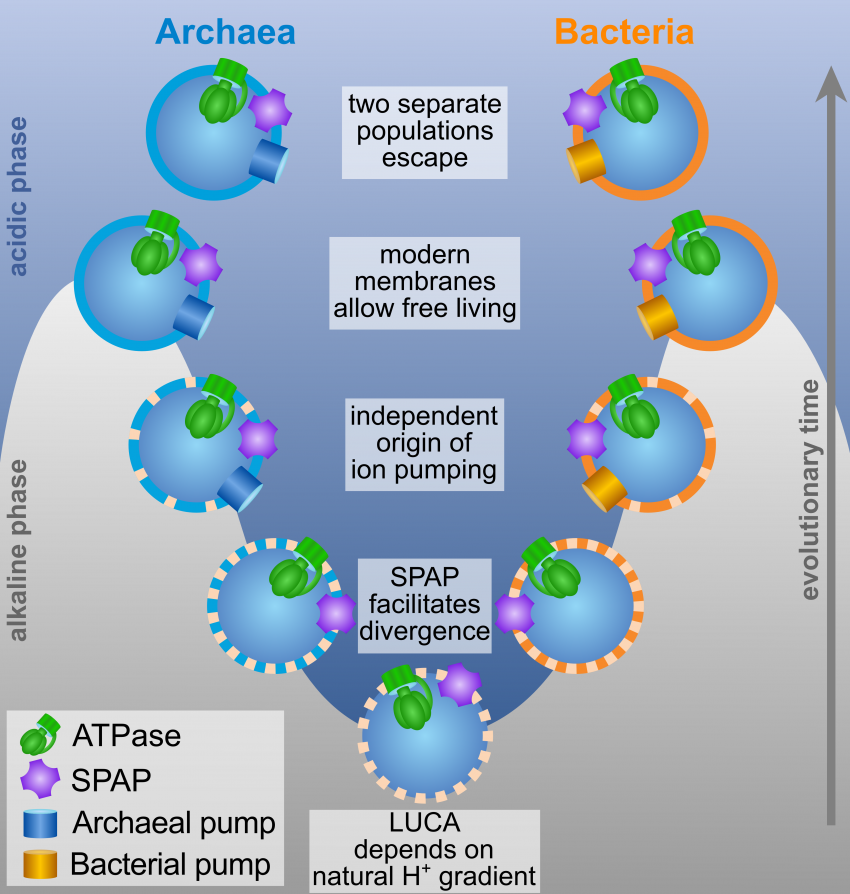
Abb. 4: Entstehung der Bacteria und Archaea.
Bill Martin und der Evolutionsbiologe Eugene Koonin veröffentlichten 2005 zudem ihre Theorie über den Ursprung von Genen und Genomen in hydrothermalen Quellen (Koonin & Martin 2005, Abb. 5). Sie nahmen an, dass der Lebenszyklus mineralischer Zellen dem moderner Retroviren ähnelte. Retroviren besitzen ein winziges Genom, dass sich lieber in RNA als DNA verschlüsselt. Wenn sie eine Zelle befallen, kopieren Retroviren ihre RNA in DNA, wobei sie ein Enzym namens reverse Transkriptase benutzen. Die neue DNA wird zunächst im Genom des Wirtes eingepflanzt und dann zusammen mit den eigenen Genen der Wirtszellen eingelesen. Bei der Herstellung zahlreicher Kopien arbeitet das Virus also von der DNA aus, während es sich selbst für die nächste Generation verpackt, verlässt es sich auf die RNA, um die Erbinformationen zu vermitteln. Was ihm bemerkenswerterweise fehlt ist die Fähigkeit DNA selbst zu replizieren, was üblicherweise eine ziemlich mühselige Prozedur darstellt, für die zahlreiche Enzyme notwendig sind. Der Vorteil eines solchen Lebenszyklus ist die schnelle Vermehrung: das Genom ist einfach und kurz und damit die Vermehrung schneller. Der Nachteil ist aber, dass man auf die Existenz von echten Zellen angewiesen ist. Ein zweiter Nachteil ist, dass die Speicherkapazität der RNA im Vergleich zur DNA gering ist. RNA ist chemisch instabiler, dafür reaktionsfreudiger, Die Reaktionsfreudigkeit katalysiert zwar viele biochemische Reaktionen, aber macht sie instabil, wodurch große RNA-Genome nicht machbar sind, was eine unabhängige Existenz schier unmöglich macht.
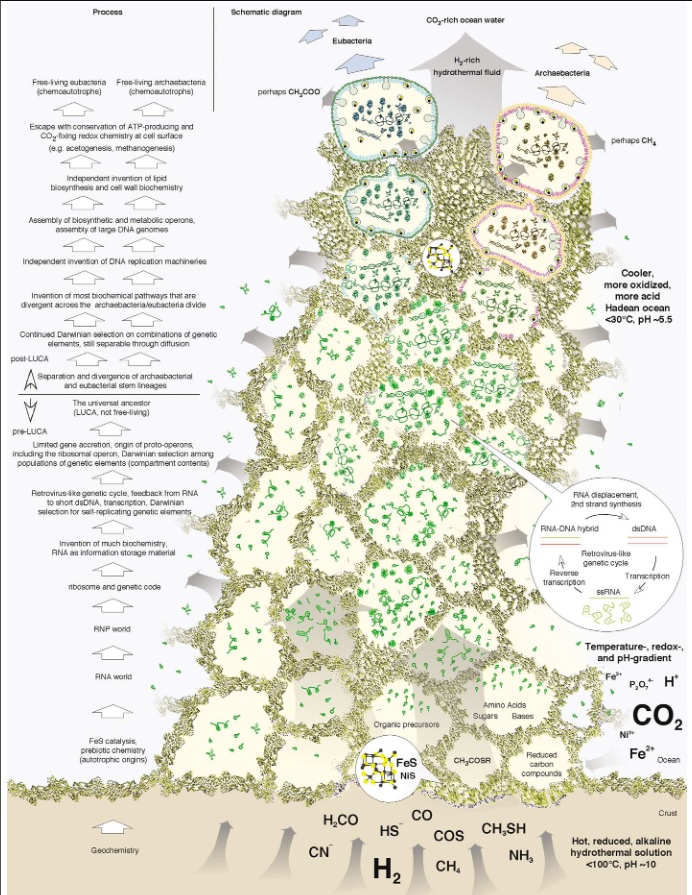
Abb. 5: Ein Evolutionsszenario vom Ursprung der RNA-Moleküle bis zum unabhängigen Entweichen von archaebakteriellen und eubakteriellen Zellen innerhalb sich natürlich bildender anorganischer Kompartimente, die in erster Linie aus FeS bestehen, in einem hydrothermalen Schlot aus dem Erdaltertum (>3,8 Ga alt). Die linke Seite der Abbildung zeigt die vorgeschlagene Abfolge der im Haupttext beschriebenen Ereignisse (von unten nach oben); der rechte Teil ist eine sehr schematische Zeichnung, die zunehmend komplexere Ebenen der molekularen Organisation innerhalb der Kompartimente entlang des Weges von anorganischem Kohlenstoff zu LUCA und von LUCA zu freilebenden chemoautotrophen Prokaryoten veranschaulicht (modifiziert von Ref. [24]). Das vergrößerte Kompartiment zeigt den vorgeschlagenen retrovirusartigen genetischen Zyklus von LUCA. Es wird angenommen, dass die Synthese des RNA-DNA-Hybrids und der dsDNA durch Reverse Transkriptase katalysiert wird, wobei RNase H (ein ubiquitäres, wahrscheinlich uraltes Enzym) an der Entfernung des RNA-Strangs während des letzten Schritts beteiligt ist. Die Transkription von RNA aus dsDNA wurde durch die DNA-abhängige RNA-Polymerase (ein weiteres universelles Enzym) katalysiert. Die Teile des Replikationssystems, die Archaebakterien und Eubakterien gemeinsam haben (Sliding Clamp, Clamp Loader ATPase und DNA-Ligase), könnten an den dsDNA-Synthese- und/oder Transkriptionsschritten beteiligt gewesen sein. Die Abbildung ist schematisch und nicht maßstabsgetreu gezeichnet. Die Kompartimente könnten einen Durchmesser von <1-100 μm haben, wie in fossilen und modernen Schloten.
Mineralische Zellen bilden jedoch einen Vorteil für die Entstehung komplexerer RNA-Lebensformen: viele der Eigenschaften, die für ein unabhängiges Leben benötigt werden, ist in den Schloten vorhanden: die Mineralzellen stellen bereits begrenzende Membranen, Energie usw. Der zweite Vorteil ist, dass sich RNA-Schwärme fortlaufend mischen und durch die miteinander in Verbindung stehenden Zellen passen. Gruppen, die gut miteinander kooperieren, können sich zusammenfinden, indem sie sich gemeinsam ausbreiten, um neu gebildete Zellen zu besiedeln. So können Populationen kooperativer RNAs entstehen, die mineralischen Zellen entsprangen, wobei jede RNA eine Handvoll verwandter Gene codierte. Der Nachteil einer solchen Vermischung ist jedoch, dass die RNA-Populationen anfällig dafür wären, sich erneut in andere, möglicherweise ungeeignete Verbindungen zu mischen. Eine Zelle, die es schaffen würde, ihr Genom zusammenzuhalten, indem sie eine kleine Gruppe kooperativer RNAs in ein einzelnes DNA-Molekül konvertiert, würde all diese Vorteile behalten. Ihre Replikation würde dann der eines Retrovirus ähneln: Ihre RNA würde in einen RNA-Schwarm überschrieben werden, der benachbarte Zellen infiziert und ihnen dieselbe Fähigkeit verleiht, nämlich die Informationen wieder in einer DNA-Bank abzulegen.
Die Umwandlung von RNA in DNA ist zudem auch nicht schwer, es gibt zwei entscheidende Unterschiede: Die erste ist die Entfernung eines Sauerstoffatoms aus der Ribose, um eine Desoxyribose zu erzeugen. Diese Reaktion läuft auch heute noch in den Schloten ab. Die zweite Veränderung ist die Anlagerung einer Methylgruppe an Uracil um Thymin herzustellen. Methylgruppen entstehen in den Schloten ebenfalls. Dadurch wird die DNA stabiler, kann größer werden und sich anreichern.
Ein möglicher Schritt auf dem Weg zu einem DNA-Genom ist die Verfügbarkeit von DNA-Vorläufern, den sogenannten Desoxyribonukleotidtriphosphaten (dNTPs), also den einzelnen Nukleotiden (Forterre 2002, Abb. 6). In modernen Zellen werden diese in zwei aufeinanderfolgenden Schritten hergestellt: Der erste Schritt ist die Reduktion von RNA-Vorläufern durch Ribonukleotid-Reduktasen, was zu Uracil-haltiger U-DNA führt. Der zweite Schritt ist die Herstellung von Desoxythymidin-5′-Monophosphat (dTMP) aus Desoxyuridin-5′-Monophosphat (dUMP) durch Thymidylat-Synthasen. Dies führt zur Bildung von Thymin-haltiger DNA (T-DNA).
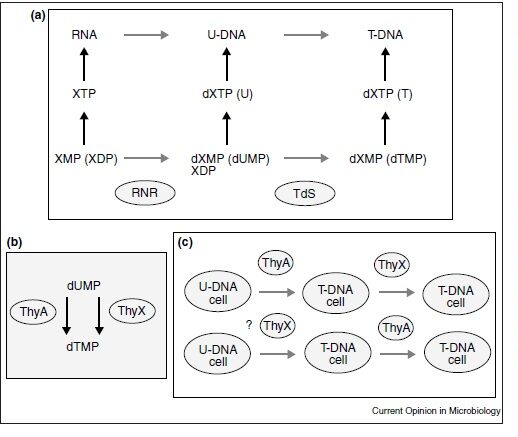
Abb. 6: Umwandlung von RNA in DNA. (a) In Analogie zum heutigen Mechanismus der Biosynthese von DNA-Vorläufern kann man folgern, dass der Übergang von RNA zu DNA in zwei Schritten erfolgte: erstens Reduktion von XMP oder XDP zu dXMP oder dXDP durch Ribonukleotid-Reduktase (RNR), was zu Uracil-haltiger U-DNA führt; dann zweitens Methylierung von Uracil in dUMP durch Thymidylat-Synthase (Tds) zur Herstellung von dTMP, was zu Thymidin-haltiger DNA (T-DNA) führt. (b) Die Umwandlung von dUMP in dTMP kann über zwei verschiedene Mechanismen erfolgen, die von zwei evolutionär nicht verwandten Enzymen, ThyA und ThyX, katalysiert werden. (c) Zwei Hypothesen für die “Erfindung” der T-DNA. Man kann sich entweder eine unabhängige “Erfindung” durch ThyA und ThyX in zwei verschiedenen Linien von U-DNA-Zellen vorstellen oder eine sekundäre “Erfindung” von ThyA in einer T-DNA-Zelle, die ThyX enthält (oder von ThyX in einer T-DNA-Zelle, die ThyA enthält). Die nicht-orthologe Verdrängung von ThyA durch ThyX und umgekehrt erfolgte in jedem Fall während der späteren Evolution der T-DNA-Zellen. RNR, Ribonukleotid-Reduktase; TdS, Thymidylat-Synthase.
Es ist auch bekannt, dass Cytosin sich spontan durch einen chemischen Prozess namens „hydrolytische Deamination“ in Uracil umwandeln kann. Cytosin und Guanin sind komplementäre Basen, d. h. bei einer Doppelstrangbildung verpaaren sich beide miteinander. Die komplementäre Base von Uracil ist Adenin. Wenn sich nun Cytosin ungewollt in ein Uracil wandelt, so würde bei der Replikation ebenfalls ein Guanin durch ein Adenin ersetzt werden, was u. U. zu nachhaltigen Veränderungen der genetischen Information führen würde. Hier kann die natürliche Selektion als „Qualitätskontrolle“ wirken. Denn Dank dieser kleinen Modifikationen ist die DNA stabiler und kann daher als besseres Speichermedium fungieren. Da die DNA langlebiger ist als die RNA besteht aus Sicht der natürlichen Selektion ein starker Selektionsdruck in Richtung Erhaltung der genetischen Information. Durch die „genetische Machtübernahme“ durch die DNA ist die RNA nicht mehr für die Weitergabe der genetischen Information verantwortlich. Dafür werden ihre katalytischen Eigenschaften immer bedeutungsvoller.
Die Rolle der Viren
Es zeigt sich, wie vorhin erwähnt, dass Viren ggfs. eine mögliche Schlüsselposition bei der Entstehung von einer RNA-basierten Protozelle zu einer DNA-basierten spielten (Forterre & Prangishvili 2013, Koonin & Dolja 2014, Moelling & Broecker 2021, Abb. 7), wobei das Vorhandensein von U-DNA eine Rolle spielen könnte (Forterre 2001, 2002).
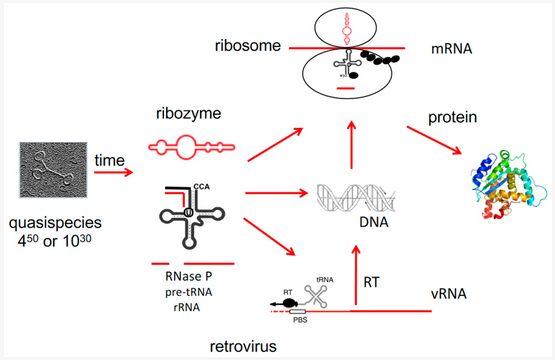
Abb. 7: Viroide/viroidähnliche RNAs/Ribozyme zu Beginn der Evolution. Das Bild zeigt ein Viroid. Viroide/Ribozyme und tRNAs können sich spontan aus einer Quasispezies vieler RNA-Moleküle bilden, beide weisen enzymatische Aktivitäten auf. Das Ribozym RNase P verarbeitet prä-tRNA zur kürzeren tRNA, DNA kann ohne reverse Transkriptase (RT) aus RNA entstehen, die Proteinsynthese in Ribosomen benötigt Ribozyme als wesentliche Bestandteile (mRNA ist Boten-RNA, vRNA ist virale RNA, PBS ist die Primer-Bindungsstelle für tRNA-Bindung).
Ein Organismus, genauer ein Virus, der auf das Vorhandensein von U-DNA selektiert wurde, hätte einen Selektionsvorteil, weil das Genom des Virus vor RNA-Inaktivierungsmechanismen des Wirtes geschützt wäre. Interessanterweise enthalten einige moderne Viren immer noch U-DNA, während andere, wie z. B. T4-Bakteriophagen, modifizierte Formen von T-DNA zum Schutz des Genoms entwickelt haben (Abb. 8, Poole et al. 2000, Forterre 2001, Kiljunen et al. 2005, Uchiyama et al. 2014). Die T-DNA könnte also auch zuerst in einem Virus selektiert worden sein, und zwar als modifizierte Form der U-DNA. Viele DNA-Viren kodieren ihre eigene Ribonukleotid-Reduktase und/oder Thymidylat-Synthasen, was mit dieser Hypothese übereinstimmt. Schließlich kodieren viele DNA-Viren auch für ihre eigenen DNA-Replikationsproteine (Arezi & Kuchta 2000, Balter 2000, Filee et al. 2002, Forterre 1992, 1999, Garcia et al. 2000, Lakshminarayan et al. 2001, Takemura 2001, Villarreal & DeFilippis 2000), so dass die Mechanismen der DNA-Replikation selbst in den Viren entstanden sein können, bevor sie auf die Zellen übertragen wurden. Beispielsweise können virale DNA-Polymerasen ein Bindeglied zwischen zellulären DNA-Replikasen und viralen RNA-Replikasen sein. So gibt es z. B. Ähnlichkeiten viraler RNA-Replikasen mit reversen Transkriptasen und einigen DNA-Polymerasen (Butcher et al. 2001, Ng et al. 2002, Steitz 1999).
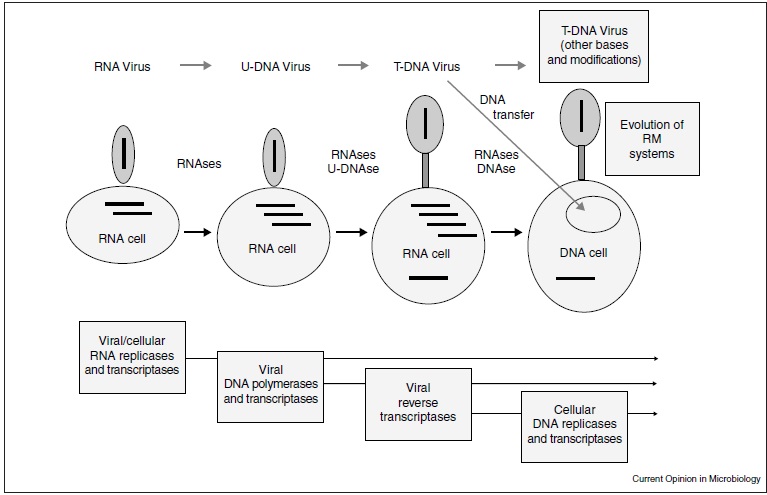
Abb. 8: Durch Wettrüsten angetriebene Koevolution zellulärer und viraler Genome. Nach dieser Hypothese haben RNA-Viren bereits in der zweiten Stufe der RNA-Welt (nach der “Erfindung” der Proteinsynthese) mit RNA-Zellen koexistiert. Die U-DNA wurde zuerst in einem RNA-Virus als modifizierte Form der RNA selektiert, um das Genom vor RNAsen zu schützen, und die T-DNA wurde später in einer U-DNA als modifizierte Form der U-DNA selektiert, um das Genom vor U-DNAsen zu schützen. DNA und DNA-Replikationsproteine wurden von mehreren T-DNA-Viren in eine RNA-Zelle übertragen, um eine DNA-Zelle zu erhalten. An diesem Übergang könnten virale reverse Transkriptasen beteiligt gewesen sein. Einige der heutigen T-DNA-Viren haben ihre DNA modifiziert, um ihre Genome vor DNA-Seuchen zu schützen. Restriktionsmodifikationssysteme (RM) sind die jüngste Ergänzung zu diesen Mechanismen im Zusammenhang mit dem Konflikt zwischen Zellen und Viren.
Man kann sich vorstellen, dass es einer RNA-Zelle gelungen ist, ihr Genom durch Retrotranskription in DNA umzuwandeln, um gegen virale RNasen resistent zu werden, oder dass ein DNA-Virus, das in einer RNA-Zelle lebt, die volle Kontrolle über die Zelle übernommen hat und das ursprüngliche RNA-Genom verdrängt hat. Diese Szenarien implizieren, dass DNA-Viren bereits im zweiten Zeitalter der RNA-Welt vorhanden waren (Forterre 1999). Tatsächlich deuten vergleichende virale Genomik und Strukturanalysen von Viren, die entfernt verwandte Wirte infizieren, stark darauf hin, dass zumindest einige Virengruppen uralt sind (Lakshiminarayan et al. 2001, Butcher et al. 2001, Hendrix et al. 2000) und möglicherweise schon vor der Divergenz der drei Domänen von LUCA vorhanden waren. Interessanterweise sind die meisten Replikationsproteine, die von DNA-Viren kodiert werden, oft nur sehr entfernt mit ihren zellulären funktionellen Analoga in ihren Wirten verwandt (Arezy & Kuchta 2000, Filee et al. 2002, Garcia et al. 2000, Takemura 2001, Villarreal & DeFilippis 2000), obwohl einige Fälle von kürzlich erfolgten Übertragungen von Zellen auf Viren durch phylogenetische Analysen identifiziert werden können (Filee et al. 2002, Martin & MacNeil 2002, Moreira 2000).
Ursprung der DNA-Polymerase
Ein für die Vermehrung, also Replikation, der DNA wichtiges Enzym ist die DNA-Polymerase. Hierbei handelt es sich jedoch nicht um ein einziges Enzym, sondern um verschiedene Enzyme, die an der Replikation der DNA beteiligt sind. Die DNA-Polymerasen der einzelnen Domänen bestehen aus verschiedenen Proteinfamilien, mit andersartigen katalytischen Zentren. Dass sie so unterschiedlich sind, spricht für eine unabhängige Entwicklung des Replikationssysems bei den einzelnen Domänen. Die verschiedenen DNA-Polymerasen der Domäne der Bacteria werden mit römischen Zahlen benannt. Die bei der Replikation der DNA wichtigsten sind die Polymerase III (Abb. 9), aber auch I. Polymerase II, IV und V haben wahrscheinlich eine Rolle in der Reparatur der DNA (Burgers et al. 2001, Pavlov et al. 2006, Raia et al. 2019, 2019a Koonin 2006, Makarvoa et al. 2014, Cann & Ishino 1999, Banach-Orlowska et al. 2005, Goodman 2002, Patel et al. 2010, Sutton & Walker 2001, Raychaudhury & Basu 2011, Munk 2009).
Abb. 9: Schema der DNA-Polymerase III.
Die DNA-Polymerasen der Eukaryoten werden nach griechischen Buchstaben benannt. Die Polymerase alpha (α), delta (δ) und epsilon (ε) sind für die Kettenverlängerung in der DNA-Replikation bei den eukaryotischen Lebewesen zuständig. Polymerase beta (β) ist an der Reparatur der DNA beteiligt. Weiterhin kommt in den Plastiden und Mitochondrien eukaryotischer Zellen die Polymerase gamma (γ) vor Yamitch & Sweasy 2010, Chung et al. 1991, Pursell et al. 2007, Lujan et al. 2016, Johnson et al. 2015, Doublie & Zahn 2014, Edwards et al. 2003, Bienstock et al. 2014, Prasad et al. 2017, Stumpf & Copeland 2011, Munk 2009).
Retro-Viren verfügen ebenfalls über eigene DNA-Polymerasen, diese sind aber RNA-abhängig, man spricht also von RNA-abhängigen DNA-Polymerasen. Sie nutzen einen RNA-Strang zur Synthese von DNA. Das Enzyme Reverse Transkriptase der Retroviren ist ein solches Beispiel. Eine RNA-abhängige DNA-Polymerase in Eukaryoten sind die Telomerasen, welche aus einem Protein- (TERT) und einem langen RNA-Anteil (TR) bestehen und die Endstücke der Chromosomen, die sogenannten Telomere, wieder herstellen (Hu & Temin 1990, Munk 2009).
Trotz ihrer funktionellen Unterschiede lassen sich die Polymerasen in verschiedene Familien einteilen, die mit Großbuchstaben benannt werden, da einzelne Untereinheiten der jeweiligen Polymerasen ihre Homologien haben (Filee et al. 2002, Abb. 10). Zu den Polymerasen der Familie A gehören z. B. die bakterielle Polymerase I und die eukaryotische, in Mitochondrien Polymerase gamma (γ). Zu den Polymerasen der Familie B gehören die bakterielle Polymerase II und die eukaryotischen Polymerasen alpha (α), delta (δ) und epsilon (ε). Zur Familie C gehört die bakterielle Polymerase III. Hier sehen wir, dass die für die Replikation wichtigste Polymerase der Bakterien einer anderen Familie angehört, als jene der Eukaryoten. Polymerasen der Familie X kommen nur bei Eukaryoten vor, z. B. Polymerase beta (β). Die bakteriellen Polymerasen IV und V gehören in die Familie Y. die RNA-abhängigen DNA-Polymerasen werden in die Familie RT eingegliedert.
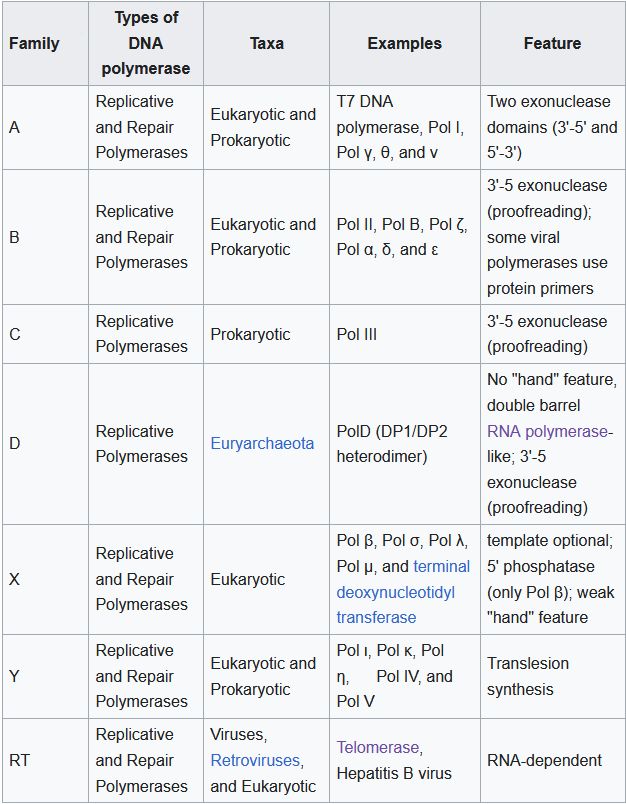
Abb. 10: die verschiedenen Polymerasen.
Die DNA-Polymerasen der Archaeen weisen Homologien zu jenen der Eukaryoten auf, sind aber einfacher gebaut (Munk 2009, Makarova et al. 2014). Es gibt aber innerhalb dieser Domäne eine DNA-Polymerase, die für einige Archaeen einzigartig ist, die Polymerase D.
Sie finden sich z. B. bei Pyrococcus furiosus und Methanococcus jannaschii (Ishino et al. 1998). Diese Klasse der Polymerasen ist für den Ursprung der Polymerasen-Enzyme und somit für den Ursprung der Replikation von LUCA von Bedeutung (Koonin et al. 2020).
DNA-Polymerasen der Familie D bestehen aus zwei Proteinketten: DP1 und DP2. Im Gegensatz zu anderen DNA-Polymerasen ähneln die Struktur und der Mechanismus des katalytischen Kerns von DP2 dem von RNA-Polymerasen mit mehreren Untereinheiten (Saugert 2019, Saugert et al. 2016, Raia et al. 2019a; Abb. 11). RNA-Polymerasen sind für die Synthese von RNA-Polymeren verantwortlich, so auch für die Synthese der mRNA bei der Proteinsynthese.
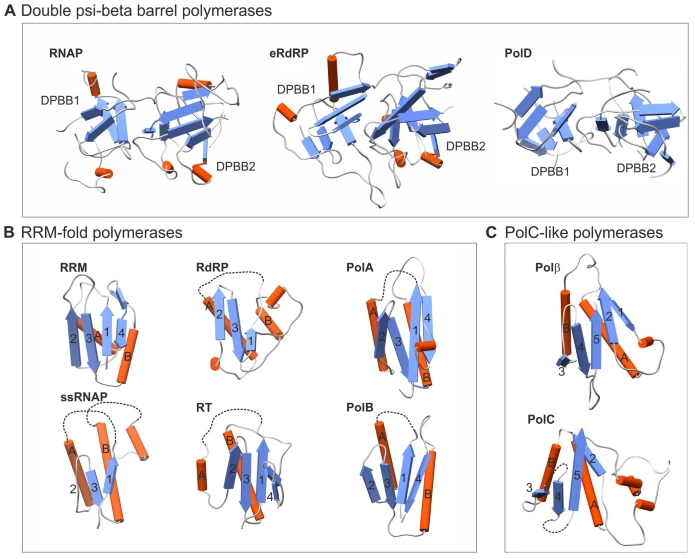
Abb. 11: Die zentralen katalytischen Domänen von DNA- und RNA-Polymerasen. a Double-psi beta-barrel (DPBB) Polymerasen. RNAP, mehrgliedrige DNA-abhängige RNA-Polymerase aus Thermus thermophilus (PDB ID: 1iw7); eRdRP, eukaryotische RNA-abhängige RNA-Polymerase aus Neurospora crassa (PDB ID: 2j7n); PolD, DP2-Untereinheit der DNA-Polymerase der Familie D aus Pyrococcus abyssi (PDB ID: 5ijl). b RRM-gefaltete Polymerasen. RRM, RNA-Erkennungsmotiv-enthaltende RNA-Bindungsdomäne des menschlichen Nucleolysins TIAR (PDB ID: 2cqi); RdRP, RNA-abhängige RNA-Polymerase des Poliovirus Typ 1 (PDB ID: 1ra7); PolA, DNA-Polymerase der Familie A aus Thermus aquaticus (PDB ID: 1taq); ssRNAP, DNA-abhängige Einzeluntereinheit-RNA-Polymerase des Bakteriophagen T7 (PDB ID: 1msw); RT, reverse Transkriptase des Moloney-Mausleukämievirus (PDB ID: 1mml); PolB, DNA-Polymerase der Familie B aus Thermococcus gorgonarius (PDB ID: 1tgo). c PolC-ähnliche Polymerasen. Polβ, DNA-Polymerase β aus Rattus norvegicus (PDB ID: 1bpb); PolC, DNA-Polymerase der Familie C aus Thermus aquaticus (PDB ID: 2hpi). In b und c sind die wichtigsten Sekundärstrukturelemente, die den Kern der Palmdomäne bilden, mit Zahlen (für β-Stränge) und Großbuchstaben (für α-Helices) angegeben. Gestrichelte Linien zeigen Regionen an, in denen Einfügungen in die Core Palm Domänen stattgefunden haben; diese wurden aus Gründen der Visualisierung weggelassen.
Die DP1-DP2-Schnittstelle ähnelt dem Zinkfinger der eukaryotischen Klasse-B-Polymerase und ihrer kleinen Untereinheit (Raia et al. 2019a, Abb. 12). Zinkfingerproteine sind eine Klasse von nukleinsäurebindenden Proteinen, die eine bestimmte Proteindomäne besitzen: die Zinkfingerdomäne, bei der ein Zinkion (Zn2+) koordinativ gebunden ist (Thiel & Lietz 2004, Munk 2009; Abb. 13). Die Polypeptidkette nimmt durch den Einbau des Zinkatoms eine schleifenförmige Struktur – den sogenannten Zinkfinger – ein, welche spezifisch mit der DNA oder auch RNA interagieren kann.
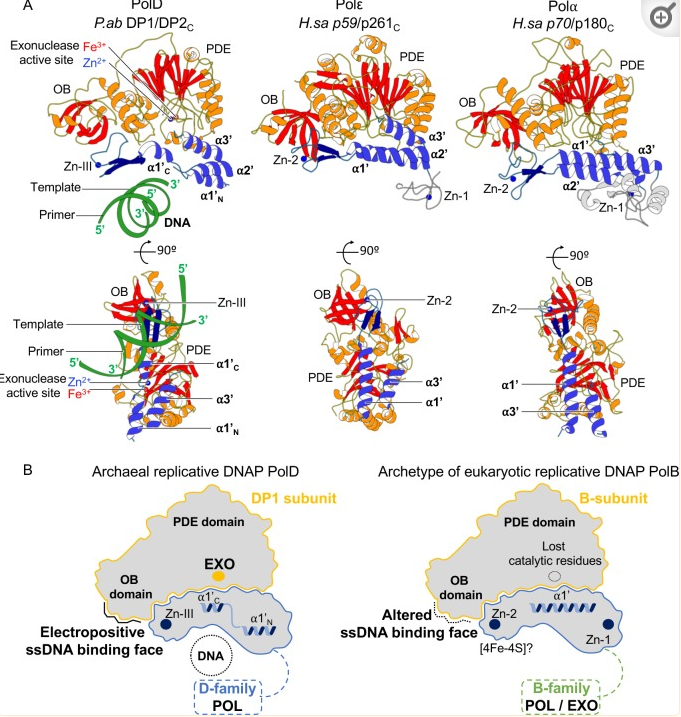
Abb. 12: Struktureller Vergleich der Intersubunit-Schnittstelle von PolD der Archaeen und eukaryotischen replikativen DNAPs. (A) Vergleich der DP1/DP2-CTD-Region der PolD-Kryo-EM-Struktur von P. abyssi mit den Kristallstrukturen des menschlichen Polε p59/p261C (PDB ID: 5VBN [24]) und des menschlichen Polα p70/p180C (PDB ID: 4Y97 [58]). Die eukaryotische Zn-1-Bindungsdomäne, die in PolD nicht konserviert ist, ist grau eingefärbt. (B) Schematische Darstellung der DP1-DP2-Schnittstelle von PolD und der A-Untereinheit/B-Untereinheit-Schnittstelle eukaryotischer DNAPs, die die Unterschiede zwischen beiden Strukturen hervorhebt. Kryo-EM, Kryo-Elektronenmikroskopie; CTD, C-terminale Domäne; DNAP, DNA-Polymerase; EXO, aktive Stelle der Exonuklease; H.sa, H. sapiens; OB, Oligonukleotidbindung; PDB, Protein Data Bank; PDE, Phosphodiesterasedomäne; ssDNA, einzelsträngige DNA.
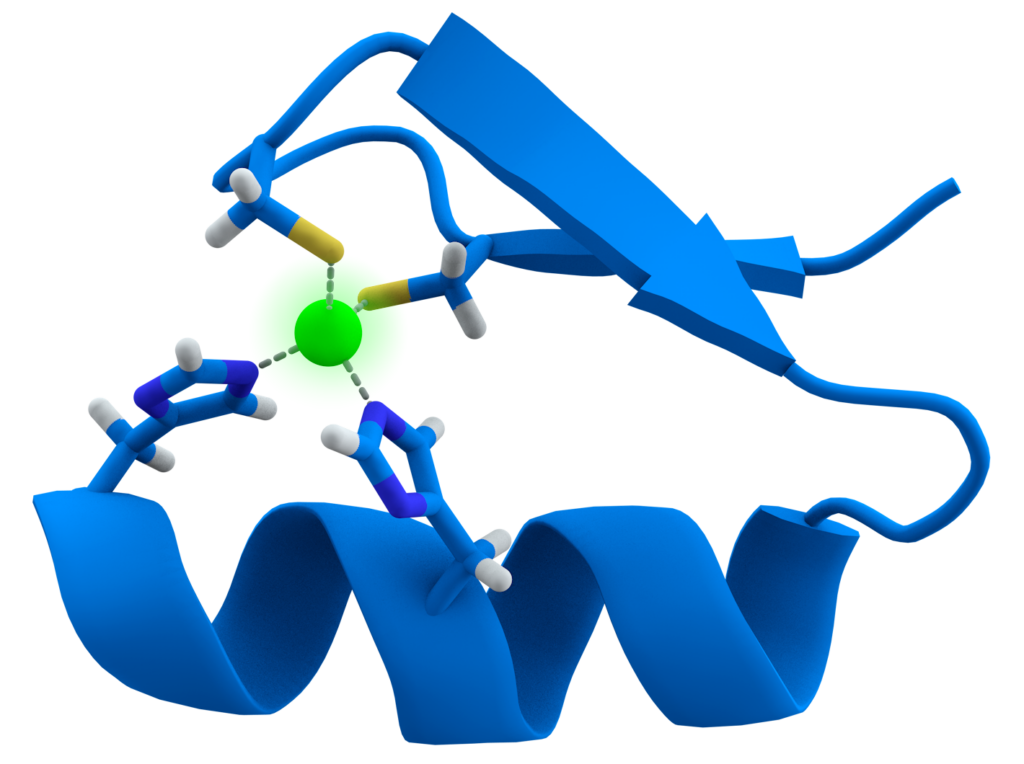
Abb. 13: Darstellung eines Zinkfingerproteins, das aus einer α-Helix und einer antiparallelen β-Faltblatt-Struktur besteht. Das grün dargestellte Zinkion wird von je zwei Histidin- und Cystein-Resten koordinativ gebunden.
DP1 ist wahrscheinlich der Vorläufer der kleinen Untereinheit von Polmerase α und ε der Eukaryoten und bietet Korrekturlesefähigkeiten, die in Eukaryoten verloren gegangen sind. Weiterhin haben einige Domänen von DP1 Ähnlichkeiten mit einer Untereinheit der Bakterien Polymerase III (Makarova et al. 2014, Raia et al. 2019, 2019a, Koonin et al. 2020, Madru et al. 2020, Abb. 14 und 15). Es wurde aufgrund dieser Homologien mit den Polymerasen anderer Domänen vorgeschlagen, dass die DNA-Polymerase der Familie D die erste war, die sich in zellulären Organismen entwickelt hat, und dass die replikative Polymerase von LUCA zur Familie D gehörte.
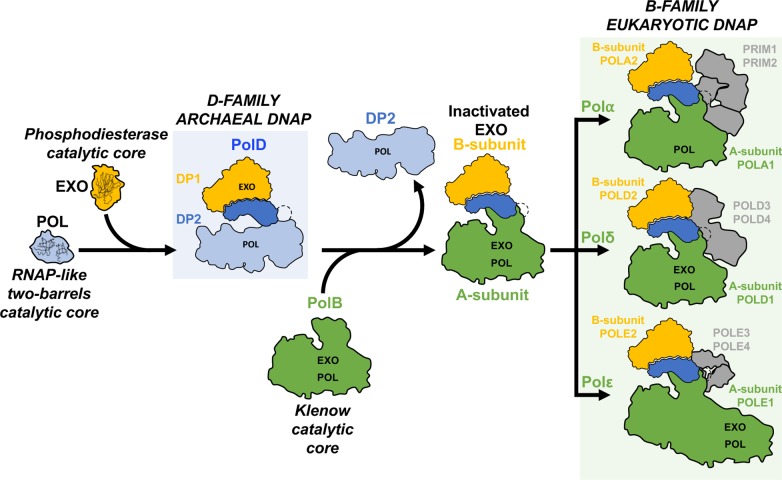
Abb. 14: Hypothetisches Szenario, das die Ursprünge der PolD und ihre evolutionären Beziehungen zu den eukaryotischen replikativen DNAPs erklärt. Eukaryotische replikative DNAPs zeigen eine komplexe Beziehung zu ihren Archaeen-Vorfahren, einschließlich Beiträgen sowohl der B- als auch der D-Familie der Polymerasen. Im Laufe der Evolution wurde der katalytische Kern der D-Familie DP2 durch einen katalytischen Kern der B-Familie in allen eukaryotischen replikativen DNAPs ersetzt, die somit im Vergleich zu ihren archäischen Homologen chimär sind. Der Vergleich der PolD-Kryo-EM-Struktur mit den eukaryotischen DNAPs zeigt, dass der eukaryotische A-Untereinheit-CTD/B-Untereinheit-Komplex mehrere Merkmale verloren hat, die für die Funktion des DP2-CTD/DP1-Komplexes in PolD kritisch sind, was auf das folgende hypothetische Szenario hindeutet, das die evolutionären Beziehungen zwischen dem archäischen PolD und den eukaryotischen replikativen DNAPs erklärt PolD hat einen RNAP-ähnlichen katalytischen Zwei-DPBB-Kern für die DNA-Replikation rekrutiert. Im Laufe der Evolution wurde der RNAP-ähnliche katalytische Kern von DP2 durch eine katalytische Untereinheit aus der B-Familie ersetzt. Die aktive Nuklease-Stelle von DP1 wurde inaktiviert, da die Proofreading-Aktivität nun von der katalytischen Untereinheit (A) übernommen wird. Während in PolD der DP1/DP2-CTD-Komplex aktiv zur DNA-Bindung beiträgt, sind die CTD/B-Untereinheit-Komplexe der eukaryotischen A-Untereinheit in Polα, Polδ und Polε an der Interaktion mit anderen Mitgliedern der Replikationsgabel beteiligt. cryo-EM, Kryo-Elektronenmikroskopie; CTD, C-terminale Domäne; DNAP, DNA-Polymerase; EXO, Exonuklease-Aktivstelle; RNAP, RNA-Polymerase.
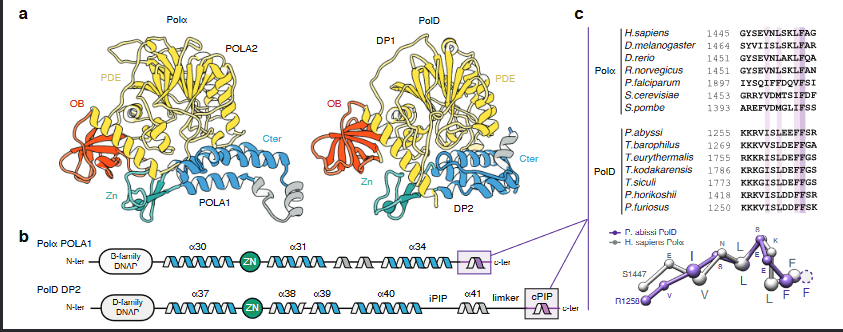
Abb. 15: Strukturen von PolD und eukaryotischem Polα. Nicht nur die allgemeine Topologie ist konserviert, die beiden teilen auch eine bifunktionelle Primase- und PCNA-bindende PIP-Box-Sequenz am C-Terminus, die sowohl dem eukaryotischen Polα als auch dem Polε ähnlich ist.
Hier wird postuliert (Koonin et al. 2020, Abb. 16), dass die DNA-Polymerase der Familie D wegen ihrer katalytischen Ähnlichkeiten zu RNA-Polymerasen aus diesen hervorgegangen ist und es zu einer Trennung zwischen der Transkriptions- und Replikationsmaschinerie kam, die durch die Anhäufung zusätzlicher Proteindomänen und deren Rekombination beschleunigt wurde. Hier könnten Viren, die ihre eigenen DNA-Polymerasen haben und die Wirtszellen befallen haben, wie erwähnt, eine wichtige Rolle gespielt haben. So wurde die Polymerase der Familie C bei Bakterien zur dominierenden Polymerase für die Vermehrung der DNA, während bei den Archaeen die Polymerase der Familie D für die Replikation sorgte. Die Evolution der Archaeen war aber mit dem Erwerb mehrerer Polymerasen der B-Familie verbunden. Bei den meisten Archaeen ist diese Familie nicht an der Replikation beteiligt, bei den Crenarchaeota haben jedoch einige Polymerasen der Familie B, die angestammte Polymerase D für die Replikation ersetzt. Eine ähnliche Verdrängung fand zu Beginn der Evolution der Eukaryoten statt. In diesem Fall hat sich Polymerase B offenbar mit Polymerade D rekombiniert, wobei die. Nachfolgende Duplikationen von Polymerase B zu Beginn der Evolution der Eukaryoten führten zu den verschiedenen Polymerasen wie Polymerasen alpha (α), delta (δ) und epsilon (ε). Die Evolution von Polymerase B in Eukaryonten beinhaltete auch die Inaktivierung der Untereinheit DP1, die eine strukturelle Rolle behielt.
Der Ursprung der Familie A, das in fast allen Bakterien konserviert ist und eindeutig ein Vorfahre im bakteriellen Bereich ist, bleibt ungewiss. Eine Möglichkeit ist, dass Polymerasen der Familie A von einem Virus abstammt und dann von einem bakteriellen Vorfahren übernommen wurde. In Bakterien ist Polymerase der Familie A Reparaturenzyme, die nicht direkt an der Replikation beteiligt sind, aber in einigen Viren und in eukaryotischen Mitochondrien fungiert es als replizierende Polymerase. Bemerkenswerterweise wurde Polymerase A von einigen Bakteriophagen PolA als RNA-Polymerase eingefangen und anschließend in gleicher Funktion von eukaryotischen Mitochondrien rekrutiert, höchstwahrscheinlich von einem Phagen (Falkenberg et al. 2007, Filee & Forterre 2005). Die Rekrutierung viraler Polymerasen, die oft katalytisch effizienter sind als zelluläre Gegenstücke (Menendez-Arias & Andino 2017, Salas & de Vega 2016), durch zelluläre Organismen scheint also ein wiederkehrendes Thema in der Evolution zu sein, wobei der postulierte Ersatz von PolD durch PolB bei der Entstehung der Eukaryonten nur ein Beispiel ist (wenn auch eines von großer Bedeutung). Es sind zwar noch viele Fragen ungeklärt, aber die Entdeckung der Polymerase D liefert einige spannenden Hinweise über den Ursprung der Replikation des Genoms.
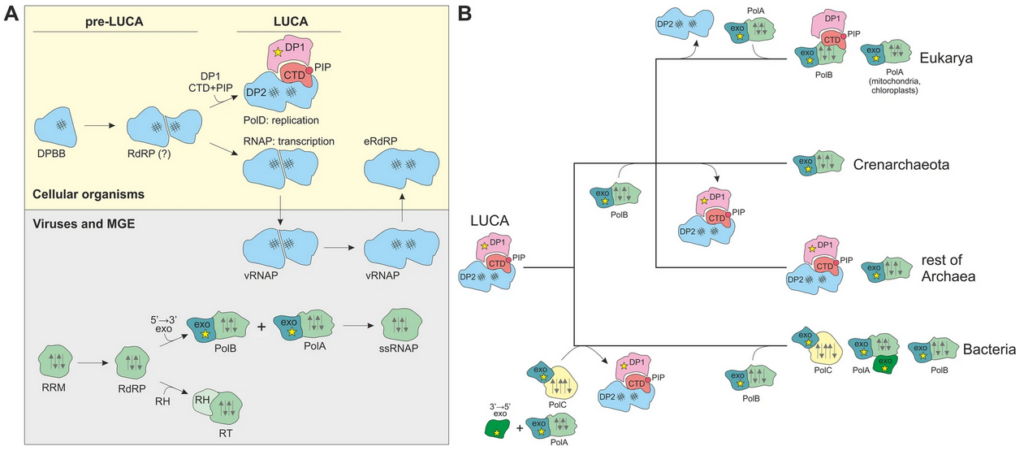
Abb. 16: Vorgeschlagenes Szenario für den Ursprung und die frühe Evolution der DNA-Replikation und -Transkription. a Evolution der zellulären (oben) und viralen (unten) Polymerasen aus einem Doppel-psi-Beta-Fass (DPBB) bzw. RNA-Erkennungsmotiv (RRM)-haltigen Proteinen. Die ersten DPBB- und RRM-basierten Polymerasen sind wahrscheinlich in den frühesten Stadien der Evolution in Protozellen entstanden, vor der Entstehung des Letzten Universellen Zellulären Vorfahren (pre-LUCA); die für die Genomreplikation und Transkription von LUCA verantwortlichen Polymerasen entwickelten sich aus einem gemeinsamen Vorfahren. DPBB-basierte RNAPs wurden zwischen der zellulären und der viralen Welt in beide Richtungen ausgetauscht. b Szenario für die Evolution der DNA-Replikationsmechanismen in den drei Lebensbereichen. Die verschiedenen Formen von PolB, die sowohl in Archaeen als auch in Eukaryonten vorkommen, sind der Einfachheit halber nicht dargestellt. Die verschiedenen Domänen und Untereinheiten sind durch verschiedene Formen und Farben gekennzeichnet. Der gelbe Stern zeigt eine aktive Exonuklease-Domäne an. Beachten Sie, dass die DP1-Untereinheit in den eukaryotischen DNAPs eine inaktivierte Exonuklease ist. DPBB ist mit einem dreifachen Hashtag-Symbol gekennzeichnet, während die Palm-(RRM)-Domänen durch Pfeile dargestellt sind. (e)RdRP, (eukaryotische) RNA-abhängige RNA-Polymerase; (ss)RNAP, (einteilige) DNA-abhängige RNA-Polymerase; RT, Reverse Transkriptase; PolA, B, C, und D, DNA-Polymerasen der Familien A, B, C und D; DP1, kleine Untereinheit von PolD mit Exonuklease-Aktivität; DP2, große Untereinheit von PolD mit DNA-Polymerase-Aktivität; RH, Ribonuklease-H-Domäne; Exo, Exonuklease-Domäne; CTD, C-terminale Domäne; PIP, PCNA-interagierendes Motiv; MGE, mobile genetische Elemente.
Das Modell vom Hyperzyklus
Dass die RNA-Welt-Hypothese bei weitem mehr ist als eine Spekulation, dafür sprechen auch weitere indirekte Belege. Das wohl gewichtigste Indiz förderte Manfred Eigen im Rahmen seines Modells vom Hyperzyklus zutage, das im Folgenden vorgestellt werden soll. Der „Hyperzyklus“ wurde von Eigen als erstes evolutionsfähiges Replikationssystem postuliert. Im einfachsten Hyperzyklus finden RNA-Moleküle zusammen, die sich in gegenseitiger Wechselwirkung aus einer Substratlösung hervorbringen und „vermehren“. Dabei koppeln sich entweder zwei oder mehrere selbstreproduzierende RNA-Stränge (Ribozyme) oder aber Ribozyme und Enzyme zu einem stabilen Autozyklus, der sich selbst unterhält und repliziert (Eigen 1971, Eigen & Winkler 1973/1974, Eigen & Schuster 1977, 1979; Abb. 17).
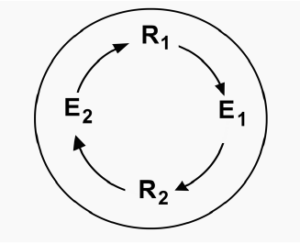
Abb. 17: Hyperzyklus nach Eigen: Ein intermediäres Molekül R (1,2,…,n-1), z. B. RNA, repliziert sich selbst; dann wird ein Produkt davon abgelesen und synthetisiert, in diesem Fall ein Enzym E (1,2,…,n-1), welches die Synthese des nächsten Moleküls R (2…n) katalysiert usw. Im Gegensatz zu einem einfachen katalytischen Zyklus kann sich der Hyperzyklus evolutionär weiter entwickeln; er wächst hyperbolisch. Experimentell sind bisher einige RNA Moleküle mit Hilfe der Qß-Replikase auf eine solche Art in einem Flow-Reactor synthetisiert worden. Mit Hilfe chromatographischer Auftrennung kann man Moleküle mit vorher bestimmten Eigenschaften isolieren (Schuster 2007; dort weitere Literatur).
EIGEN hatte das Modell vom Hyperzyklus ersonnen, um zu berechnen, ab welcher Sequenzlänge die bei der Vermehrung selbstreproduzierender Polynukleotide (Ribozyme) auftretenden Kopierfehler die „Information“ auseinanderdriften lassen und wie groß die Fehlerquote mindestens sein muss, damit das System überhaupt evolviert. Die bei jedem Replikationsschritt auftretenden Fehler kommen durch Mutationen zustande, die bewirken, dass aus einer Ursequenz allmählich ein „Kometenschweif“ bestehend aus ähnlichen Ribozymen entsteht. Es handelt sich dabei also um ein Mutantenensemble, die so genannte Quasispezies, deren Mitglieder hinsichtlich Kopiergenauigkeit, Stabilität und Replikations-geschwindigkeit miteinander in Konkurrenz stehen. Wenn keine Fehler auftreten würden, gäbe es keine Entwicklung und auch keine Evolution. Die Fehler dürfen aber auch nicht zu groß sein, sonst endet der Prozess in einer so genannten „Fehler-Katastrophe“ (Eigen & Schuster 1979, Swetina & Schuster 1982, Schuster & Stadler 1999, Loeb et al. 1999).
Für Polynukleotide, die ausschließlich aus stabilen Guanin-Cytosin-(GC-) Basenpaarungen bestehen, beträgt die maximal erlaubte Fehlerquote nur etwa 1%, die Kette dürfte maximal 100 Nukleotide (nt) lang sein. Für Polynukleotide, die ausschließlich aus Adenin-Uracil-Basenpaaren bestehen, beträgt die maximale Ablesefehlerquote bei der Replikation etwa das Zehnfache, die Kettenlänge könnte maximal nur 10 nt betragen.
Um eine stabile Reproduktion ohne „Informationsauflösung“ über beliebig viele Generationen hinweg zu gewährleisten, ist es unmöglich, das „Ur-Genom“ auf einem einzelnen Molekül unterzubringen. Bilden sich in einer Quasispezies jedoch zwei oder mehrere Mutanten heraus, die ihre Reproduktion gegenseitig katalysieren und stabilisieren, entstehen kooperative Systeme, die sich über lange Zeiten stabil reproduzieren und gegenüber allen anderen Konkurrenten im Mutantenensemble einen entscheidenden Vorteil besitzen. Als Bedingung muss jedoch gelten, dass jedes Ribozym aus 50–100 nt besteht. Für das „Ur-Gen“ muss man also einen GC-Anteil von 50–100 % annehmen, da die RNA-Matrizen lediglich dann eine hinreichend kleine Fehlerquote besitzen.
Das eigentlich Spektakuläre ist, dass es tatsächlich gelang zu zeigen, dass die tRNA-Moleküle heutiger Organismen einer Quasispezies-Verteilung entsprungen sind, wie dies nach dem Modell des Hyperzyklus zu erwarten ist (Eigen & Winkler-Oswatitsch 1981a, b). Zudem weisen die tRNAs exakt die angesprochenen Verhältnisse (hoher GC-Anteil von ca. 80% und eine durchschnittliche Kettenlänge von 76 Nukleotiden) auf, wie es die Theorie mathematisch erwarten ließ. Überdies konnte anhand der variablen Sequenzen die Ursequenz mathematisch rekonstruiert werden, wodurch die Hypothese der RNA-Welt empirisch gestützt wird (Kämpfe 1992: S. 201).
Das Chemoton
Ausgehend vom Modell des Hyperzyklus entwarfen und bauten einige weitere Arbeitsgruppen Synthese- bzw. Evolutionsautomaten; sie werden als „Automaton“ (mathematischer Modellautomat), „Chemoton“ (Ganti 1975, 2003a, b, Munteanu & Sole 2006, Sole et al. 2007) oder einfach als „Flow-Reactor“ (Schuster 2007) bezeichnet, welche als Nachfolger des legendären Millerschen Versuchs gelten können. Ein sehr interessantes Modell ist sicher das viel zitierte „Chemoton“ des ungarischen Wissenschaftlers Ganti (Abb. 18). Es wird in der einschlägigen Literatur als Modell für das kleinste sich selbst replizierende System angesehen („minimal unit of life“) und ist relativ unkompliziert.
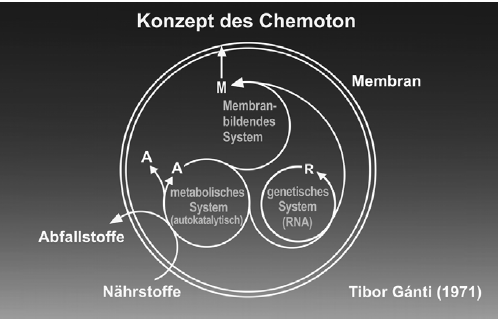
Abb. 18: Das Chemoton von Ganti: Drei selbst-produzierende Subsysteme sind an-einander gekoppelt. Diese Kopplung führt zur Proliferation und zum Programm-kontrollierten, „fließenden Automaton“, dem Chemoton.
Für Wissenschaftler, die versuchen, diesen Funken des Lebens zu rekonstruieren, bietet das Chemoton ein attraktives Ergebnis für Experimente. Wenn es gelingt, Chemikalien dazu zu bringen, sich selbst zu einem Chemoton zusammenzusetzen, zeigt das einen Weg auf, auf dem das Leben entstanden sein könnte. Gánti argumentierte, dass in jedem lebenden Organismus zwei Schlüsselprozesse ablaufen müssen. Erstens muss er seinen Körper aufbauen und erhalten; das heißt, er braucht einen Stoffwechsel. Zweitens muss er eine Art von Informationsspeicher haben, zum Beispiel ein Gen oder Gene, die kopiert und an die Nachkommen weitergegeben werden können.
Gántis erste Version seines Modells bestand im Wesentlichen aus zwei autokatalytischen Gruppen mit unterschiedlichen Funktionen, die sich zu einer größeren autokatalytischen Gruppe zusammenschlossen. Damit unterschied es sich gar nicht mal so sehr von Eigens Hyperzyklus. Im folgenden Jahr wurde Gánti jedoch von einem Journalisten auf einen entscheidenden Fehler hingewiesen: Gánti nahm an, dass die beiden Systeme auf im Wasser schwimmenden Chemikalien basierten. Doch sich selbst überlassen, würden sie auseinanderdriften – und das Chemoton würde „sterben“.
Die einzige Lösung bestand darin, ein drittes System hinzuzufügen: eine äußere Barriere, um sie einzudämmen. In lebenden Zellen ist diese Barriere eine Membran, die aus fettähnlichen Chemikalien besteht, den Lipiden. Das Chemoton musste eine solche Barriere haben, um sich selbst zusammenzuhalten. Gánti schloss daraus, dass sie ebenfalls autokatalytisch sein musste, damit sie sich selbst erhalten und wachsen konnte.
Und so kam das Endkonzept des Chemotons zustande – Gántis Konzept des einfachsten möglichen lebenden Organismus: Gene, Stoffwechsel und Membran, alles miteinander verbunden. Der Stoffwechsel produziert Bausteine für die Gene und die Membran, und die Gene üben Einfluss auf die Membran aus. Zusammen bilden sie eine selbstreplizierende Einheit: eine Zelle, die so einfach ist, dass sie nicht nur auf der Erde relativ leicht entstehen, sondern sogar alternative Biochemien auf fremden Welten erklären könnte.
Ein Argument gegen die Idee eines Chemotons als erste Form des Lebens war, dass es so viele chemische Komponenten benötigt, darunter Nukleinsäuren, Proteine und Lipide. Viele Experten hielten es für unwahrscheinlich, dass diese Chemikalien alle aus denselben Ausgangsmaterialien am selben Ort entstehen würden, daher der Reiz von abgespeckten Ideen wie der RNA-Welt.
Doch kürzlich haben Biochemiker Beweise dafür gefunden, dass alle Schlüsselchemikalien des Lebens aus denselben einfachen Ausgangsmaterialien entstehen können. In einer im September 2020 veröffentlichten Studie hat eine Forschergruppe eine Datenbank mit Experimenten aus mehreren Jahrzehnten zusammengestellt, in denen versucht wurde, die chemischen Bausteine des Lebens herzustellen (Wolos et al. 2020, Abb. 19). Sie fanden heraus, dass mit nur sechs einfachen Ausgangsmaterialien wie Wasser und Methan Zehntausende von entscheidenden Bestandteilen hergestellt werden können, darunter auch die Grundbausteine von Proteinen und RNA.
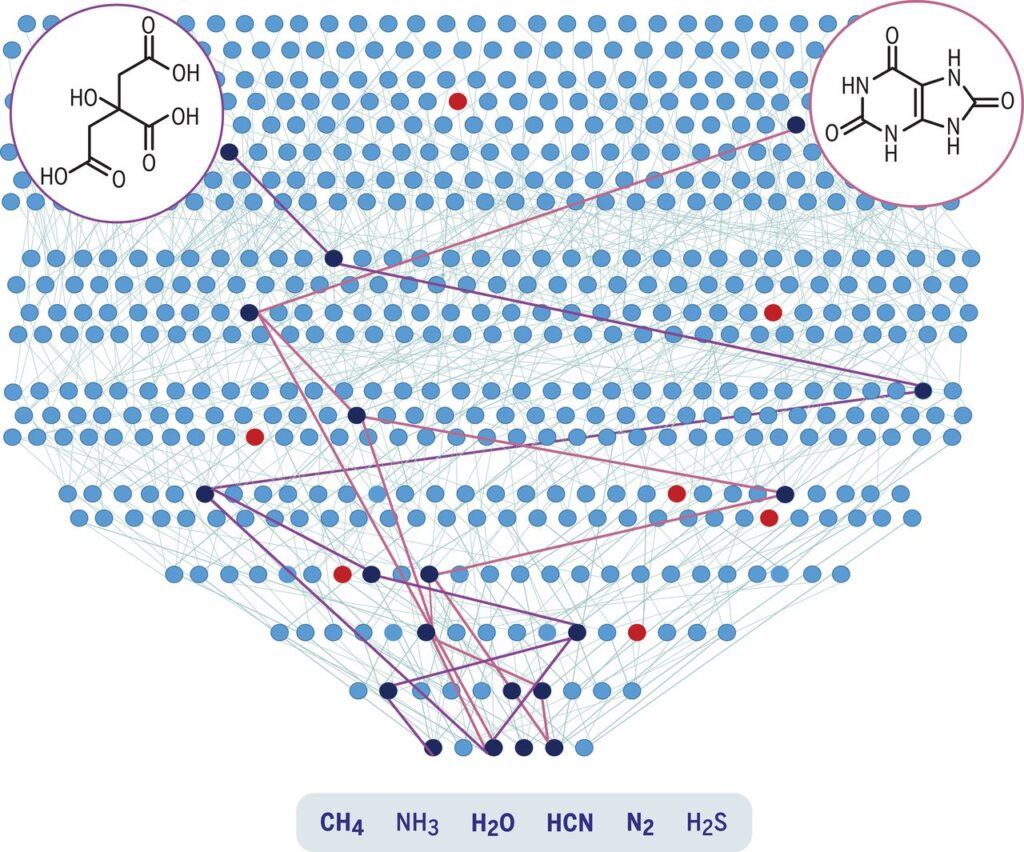
Abb. 19: Die Computersimulation plausibler präbiotischer Reaktionen schafft ein Netzwerk von Molekülen, die aus präbiotischen Rohstoffen synthetisiert werden können, und stellt mehrere bisher unbekannte – jetzt aber experimentell bestätigte – Synthesen präbiotischer Ziele sowie selbstregenerierende Zyklen her. In dieser schematischen Darstellung stehen hellblaue Knoten für abiotische Moleküle, dunkelblaue Knoten für Moleküle entlang der neu entdeckten präbiotischen Synthesen von Harnsäure und Zitronensäure und rote Knoten für andere biotische Moleküle.
Doch aus keinem dieser Experimente ging bisher ein funktionierendes Chemoton hervor. Das mag einfach daran liegen, dass es kompliziert ist, womöglich ist aber auch Gántis Modell keine exakte Entsprechung des ersten Lebens auf Erden. Dennoch gibt uns das Chemoton eine Möglichkeit, darüber zu spekulieren, wie die einzelnen Komponenten des Lebens zusammenarbeiten. Gleichzeitig bildet die Hypothese, dass das Leben in den Hydrothermalquellen der Tiefsee entstand plausible Antworten zur Entstehung des Lebens und wie die verschiedenen Komponenten des Lebens: Stoffwechsel, Informationsspeicherung und Begrenzung. Auch die im letzten Beitrag vorgestellte Arbeit von Szostak und Kollegen aus dem Jahr 2013 ist ein Beispiel, welches dem Chemoton recht nahekommt (Adamala et al. 2016, Adamala & Szostak 2013a, b). Die Forscher waren in der Lage RNA innerhalb einer Protozelle sich selbst zu vermehren (Abb. 20).
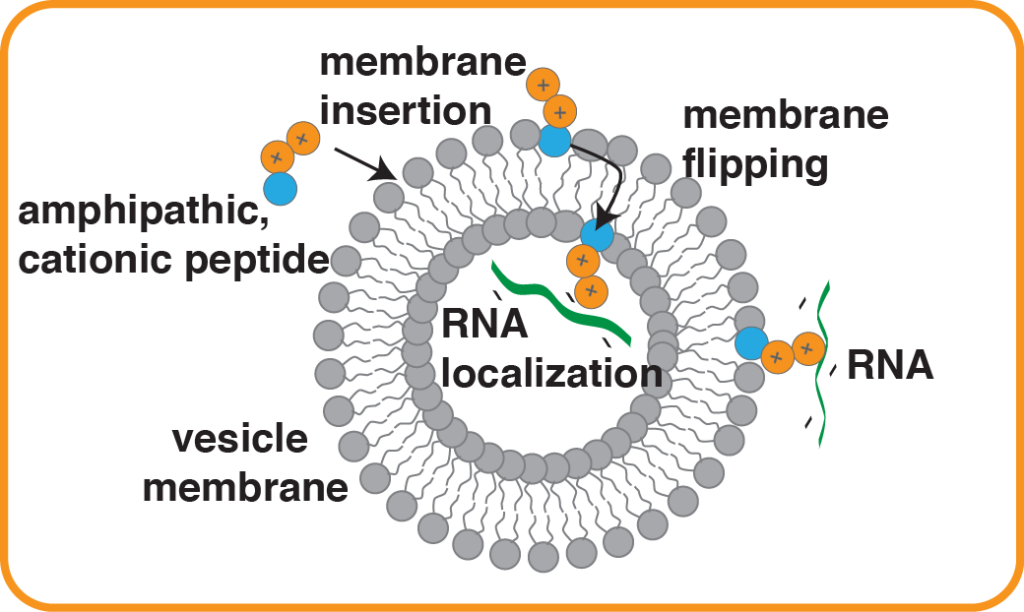
Abb. 20: In primitiven Zellen könnten Membranen eine katalytische Rolle gespielt haben, indem sie Reaktanten auf ihrer Oberfläche ko-lokalisierten. Zum Beispiel könnte die Lokalisierung von RNAs an Membranen den Zusammenbau von Ribozymen fördern und die Ribozymkatalyse erleichtern. Voraussetzung für ein solches Szenario ist ein einfacher Mechanismus zur Verankerung der RNA an der Protozellmembran. Studien haben weiterhin gezeigt, dass kurze, kationische, amphipathische Peptide die RNA-Bindung sowohl an zwitterionische Phospholipid- als auch an anionische Fettsäuremembranen fördern können. Die Assoziation dieser kationischen Peptide mit Phospholipidvesikeln kann die lokale positive Ladung einer Membran erhöhen und RNA-Polynukleotide anziehen.
Die DNA wurde zum Speichermedium, die RNA spielt dagegen eine wichtige Rolle im Zellstoffwechsel. Es entwickelt sich etwas, was oft als das „genetische Dogma“ der Molekularbiologie bezeichnet wird. „Dogma“ ist zweifelsohne ein unpassendes Wort, aber im Grunde genommen geht es darum, dass der Informationsfluss von der DNA zur RNA zum Protein fließt. Aber mittlerweile hat dieses „zentrale Dogma“ einige Revidierungen erfahren. So ist bei manchen Viren bekannt, dass die RNA in DNA umwandeln können, wenn sie das Enzym Reverse Transkriptase besitzen. Und mittlerweile ist auch bekannt, dass einige Proteine die Information zur Bildung von Proteinen haben. Das ist bei Prionen der Fall. Prionen, die eine spezielle Faltung im Vergleich zum ursprünglichen Normalprotein haben, sind in der Lage andere Proteine in Prionen umzuwandeln. Rinderwahn und die Creutzfeldt-Jakob-Krankheit werden von Prionen verursacht. Proteine haben zwar nicht die Information DNA zu bilden, können aber mittels Genregulation Einfluss auf die Genexpression nehmen.
Der entscheidende Knackpunkt ist wie die Informationen für Proteine in der DNA gespeichert sind, bzw. in RNA übertragen werden. Dies ist mit dem sog. genetischen Code möglich, den wir im nächsten Video behandeln werden.
Literatur
Adamala KP, Engelhart AE, Szostak JW (2016): Collaboration between primitive cell membranes and soluble catalysts. Nat Commun. 7:11041. doi: 10.1038/ncomms11041.
Adamala K, Szostak JW. (2013a): Nonenzymatic template-directed RNA synthesis inside model protocells. Science. 342(6162):1098-100.
Adamala K, Szostak JW (2013b): Competition between model protocells driven by an encapsulated catalyst. Nat Chem. 5(6):495-501. doi: 10.1038/nchem.1650.
Agnieszka W. et al. (2020): Synthetic connectivity, emergence, and self-regeneration in the network of prebiotic chemistry. Science 369, eaaw1955.
Arezi, B., Kuchta, R. D. (2000): Eukaryotic DNA primase. Trends Biochem Sci, 25, pp. 572-576
Baaske P, Weinert FM, Duhr S, Lemke KH, Russell MJ, Braun D. (2007): Extreme accumulation of nucleotides in simulated hydrothermal pore systems. Proc Natl Acad Sci U S A. 29,104(22):9346-51.
Balter, M. (2000): Evolution on life’s fringes. Science, 289, pp. 1866-1867
Banach-Orlowska, M. et al. (2005): DNA polymerase II as a fidelity factor in chromosomal DNA synthesis in Escherichia coli. In: Molecular Microbiology. Band 58 (1): 61–70
Bienstock RJ, Beard WA, Wilson SH (2014): Phylogenetic analysis and evolutionary origins of DNA polymerase X-family members. DNA Repair. 22: 77–88.
Buhler, C., Lebbink, J. H., Bocs, C., Ladenstein, R., Forterre, P. (2001): DNA topoisomerase VI generates ATP-dependent double-strand breaks with two-nucleotide overhangs. J Biol Chem, 276, pp. 37215-37222
Burgers PM, Koonin EV, Bruford E, Blanco L, Burtis KC, Christman MF, Copeland WC, Friedberg EC, Hanaoka F, Hinkle DC, et al. (2001): Eukaryotic DNA polymerases: proposal for a revised nomenclature. J Biol Chem 276(47):43487–90.
Butcher, S. J. et al. (2001): A mechanism for initiating RNA-dependent RNA polymerization. Nature, 410, pp. 235-240
Cann IK, Ishino Y. (1999): Archaeal DNA replication: identifying the pieces to solve a puzzle. Genetics 152(4):1249–67.
Champoux, J. J. (2001): DNA topoisomerases: structure, function and mechanism. Annu Rev Biochem, 70, pp. 369-413
Chung DW, Zhang JA, Tan CK, Davie EW, So AG, Downey KM (1991): Primary structure of the catalytic subunit of human DNA polymerase delta and chromosomal location of the gene. Proceedings of the National Academy of Sciences of the United States of America. 88 (24): 11197–11201.
Doublié S, Zahn KE (2014): Structural insights into eukaryotic DNA replication. Frontiers in Microbiology. 5: 444.
Edgell, D. R., Doolittle, W. F. (1997): Archaea and the origin(s) of DNA replication proteins. Cell, 89, pp. 995-998
Edwards S, Li CM, Levy DL, Brown J, Snow PM, Campbell JL (2003): Saccharomyces cerevisiae DNA polymerase epsilon and polymerase sigma interact physically and functionally, suggesting a role for polymerase epsilon in sister chromatid cohesion. Molecular and Cellular Biology. 23 (8): 2733–2748.
Eigen, M. (1971): Selforganization of matter and the evolution of biological mac-romolecules. Naturwissenschaften 58, 465–533.
Eigen, M., Schuster, P. (1977): The hypercycle. A principle of natural self-organization. Naturwissenschaften 64, 541–566.
Eigen, M., Schuster, P. (1979): The hypercycle – a principle of natural self-Organization. Berlin.
Eigen, M., Winkler, R. (1973/74): Ludus Vitalis. Mannheim, 53–140.
Eigen, M., Winkler-Oswatitsch, R. (1981a): Transfer RNA the early adapter. Na-turwissenschaften 68, 217–228.
Eigen, M., Winkler-Oswatitsch, R. (1981b): Transfer RNA an early gene? Naturwissenschaften 68, 282–292.
Falkenberg M, Larsson NG, Gustafsson CM. (2007): DNA replication and transcription in mammalian mitochondria. Annu Rev Biochem. 76:679–99.
Filee J, Forterre P. (2005): Viral proteins functioning in organelles: a cryptic origin? Trends Microbiol. 13(11): 510–3.
Filee, J., Forterre, P., Sen-Lin, T., Laurent, J. (2002): Evolution of DNA polymerase families: evidences for multiple gene exchange between cellular and viral proteins. J Mol Evol, 54, pp. 763-773
Forterre P. (1992): New hypotheses about the origins of viruses, prokaryotes and eukaryotes. In Frontiers of Life. Edited by Trân Thanh Vân JK, Mounolou JC, Shneider J, McKay C. Editions Frontières; Gif-sur-Yvette, France; 221-234.
Forterre, P. (1999): Displacement of cellular proteins by functional analogues from plasmids or viruses could explain puzzling phylogenies of many DNA informational proteins. Mol Microbiol, 33, pp. 457-465
Forterre P. (2001): Genomics and early cellular evolution. The origin of the DNA world. C R Acad Sci III. 324 (12): 1067-76.
Forterre P. (2002): The origin of DNA genomes and DNA replication proteins. Curr Opin Microbiol. 5(5):525-32.
Forterre P.(2005): The two ages of the RNA world, and the transition to the DNA world: a story of viruses and cells. Biochimie. 87(9-10):793-803.
Forterre P. (2013): Why are there so many diverse replication machineries? J Mol Biol. 425(23):4714-26.
Forterre, P., Prangishvili, D. (2013): The major role of viruses in cellular evolution: facts and hypotheses. Current Opinion in Virology, 3(5), 558–565.
Ganti, T. (2003a): The principles of life. Oxford.
Ganti, T. (2003b): The chemoton theory, vol. 1: Theoretical foundations of fluid machineries. New York.
Gánti T. Organization of chemical reactions into dividing and metabolizing units: the chemotons. Biosystems. 1975;7:189–195.
Garcia, A. D., Aravind, L., Koonin, E. V., Moss, B. (2000): Bacterial-type DNA Holliday junction resolvases in eukaryotic viruses Proc Natl Acad Sci USA, 97, pp. 8926-8931
Gilbert, W. (1986): The RNA world. Nature 319: 618.
Goodman MF (2002): Error-prone repair DNA polymerases in prokaryotes and eukaryotes. Annual Review of Biochemistry. 71: 17–50. doi:10.1146/annurev.biochem.71.083101.124707
Hendrix, R. W. et al. (2000): The origins and ongoing evolution of viruses. Trends Microbiol 8, pp. 504-508
Hu WS, Temin HM (1990): Retroviral recombination and reverse transcription. Science. 250 (4985): 1227–1233.
Ishino Y, Komori K, Cann IK, Koga Y (1998): A novel DNA polymerase family found in Archaea. Journal of Bacteriology. 180 (8): 2232–2236.
Johnson RE, Klassen R, Prakash L, Prakash S (2015): A Major Role of DNA Polymerase δ in Replication of Both the Leading and Lagging DNA Strands. Molecular Cell. 59 (2): 163–175.
Joyce, G. F. (1989): RNA evolution and the origins of life. Nature 338: 217–224.
Kämpfe, L. (1992): Evolution und Stammesgeschichte der Organismen. Stuttgart.
Keck, J. L., Roche, D. D., Lynch, A. S., Berger, J. M. (2000): Structure of the RNA polymerase domain of E. coli primase. Science, 287, pp. 2482-2486
Kiljunen, S. et al. (2005): Yersiniophage phiR1-37 is a tailed bacteriophage having a 270 kb DNA genome with thymidine replaced by deoxyuridine. Microbiology. 151 (12): 4093–4102.
Koga Y, Kyuragi T, Nishihara M, Sone N. (1998): Did archaeal and bacterial cells arise independently from noncellular precursors? A hypothesis stating that the advent of membrane phospholipid with enantiomeric glycerophosphate backbones caused the separation of the two lines of descent. Journal of Molecular Evolution 46: 54–63.
Koonin EV. (2006): Temporal order of evolution of DNA replication systems inferred by comparison of cellular and viral DNA polymerases. Biol Direct. 1:39.
Koonin, E.V.; Dolja, V.V. (2014): Virus world as an evolutionary network of viruses and capsidless selfish elements. Microbiol. Mol. Biol. Rev. 78, 278–303
Koonin, E.V., Krupovic, M., Ishino, S. et al. (2020): The replication machinery of LUCA: common origin of DNA replication and transcription. BMC Biol 18, 61
Koonin, E.V., Martin, W. (2005): On the origin of genomes and cells within inorganic compartments. Trends Genet. 21, 647–654.
Lakshminarayan, M. I., Aravind, L., Koonin, E. V. (2001): Common origin of four large families of large eukaryotic RNA viruses. J Virol, 75, pp. 11720-11734
Lane, N. (2010): Why Are Cells Powered by Proton Gradients? Nature Education 3(9):18
Lane, N. (2017): Der Funke des Lebens Energie und Evolution. Konrad Theis Verlag
Lane, N., Allen, J.F., Martin, W. (2010): How did LUCA make a living? Chemiosmosis in the origin of life. Bioessays 32, 271–280.
Lane, N, Martin, W. (2012): The origin of membrane bioenergetics. Cell 151: 1406–16.
Lazcano, A., Guerrero, R., Margulis, L., Oro, J. (1988): The evolutionary transition from RNA to DNA in early cells. J Mol Evol, 27, pp. 283-290
Leipe, D. D., Aravind, L., Koonin, E. V. (1999): Did DNA replication evolve twice independently? Nucleic Acids Res, 27, pp. 3389-3401
Leipe, D.D. et al. (2000): The bacterial replicative helicase DnaB evolved from a RecA duplication. Genome Res. 10, 5–16
Loeb, L. A. et al. (1999): Lethal mutagenesis of HIV with mutagenic nucleoside analogs. PNAS 96, 1492–1497.
Lujan SA, Williams JS, Kunkel TA (2016): DNA Polymerases Divide the Labor of Genome Replication. Trends in Cell Biology. 26 (9): 640–654.
Madru C, Henneke G, Raia P, Hugonneau-Beaufet I, Pehau-Arnaudet G, England P, et al. (2020): Structural basis for the increased processivity of D-family DNA polymerases in complex with PCNA. Nature Communications. 11 (1): 1591.
Makarova KS, Krupovic M, Koonin EV. (2014): Evolution of replicative DNA polymerases in archaea and their contributions to the eukaryotic replication machinery. Front Microbiol. 5:354.
Martin IV, MacNeill SA (2002): ATP-dependent DNA ligases. Genome Biol 3:REVIEWS3005.
Martin, W., Russell, M.J. (2003): On the origins of cells: a hypothesis for the evolutionary transitions from abiotic geochemistry to chemoautotrophic prokaryotes, and from prokaryotes to nucleated cells. Philos. Trans. R. Soc. Lond. B Biol. Sci. 358, 59–83, discussion 83–85.
Martin, W., Russell, M.J. (2007): On the origin of biochemistry at an alkaline hydrothermal vent. Philos. Trans. R. Soc. Lond. B Biol. Sci. 362, 1887–1925.
Martin, W., Sousa, F. L., Lane, N. (2014): Energy at life’s origin. Science 344: 1092–93.
Matsunaga, F., Forterre, P., Ishino, Y., Myllykallio, H. (2001): In vivo interactions of archaeal Cdc6/Orc1 and minichromosome maintenance proteins with the replication origin. Proc Natl Acad Sci USA, 98, pp. 11152-11157
Menendez-Arias L, Andino R. (2017): Viral polymerases. Virus Res. 234:1–3.
Mills, D. R., Peterson, R. L., Spiegelman, S. (1967): An extracellular Darwinian experiment with a self-duplicating nucleic acid molecule. In: Proceedings of the National Academy of Sciences 58 (1), 217–224
Moelling, K., Broecker, F. (2021): Viroids and the Origin of Life. International Journal of Molecular Sciences 22, no. 7: 3476. https://doi.org/10.3390/ijms22073476
Moreira, D. (2000): Multiple independent horizontal transfers of informational genes from bacteria to plasmids and phages implications for the origin of bacterial replication machinery. Mol Microbiol, 35, pp. 1-5
Munk, K. (2009, Hrsg.): Taschenlehrbuch Biologie: Genetik. Stuttgart: Thieme Verlag
Munteanu, A., Sole, R.V. (2006): Phenotypic diversity and chaos in a minimal cell model. Journal of Theoretical Biology 240, 434–442.
Myllykallio, M. et al. (2000): Bacterial mode of replication with eukaryotic-like machinery in a hyperthermophilic archaeon. Science, 288, pp. 2212-2215
Ng, K. K. et al. (2002): Crystal structures of active and inactive conformations of a caliciviral RNA-dependent RNA polymerase. J Biol Chem, 277, pp. 1381-1387
Nichols, M. D., DeAngelis, K., Keck, J. L., Berger, J. M. (1999): Structure and function of an archaeal topoisomerase VI subunit with homology to the meiotic recombination factor Spo11. EMBO J, 18, pp. 6177-6188
Patel M, Jiang Q, Woodgate R, Cox MM, Goodman MF (2010): A new model for SOS-induced mutagenesis: how RecA protein activates DNA polymerase V. Critical Reviews in Biochemistry and Molecular Biology. 45 (3): 171–184.
Pavlov YI, Shcherbakova PV, Rogozin IB. (2006): Roles of DNA polymerases in replication, repair, and recombination in eukaryotes. Int Rev Cytol. 255:41–132.
Poole, A., Penny, D., Sjöberg, B.-M. (2000): Methyl-RNA: an evolutionary bridge between RNA and DNA? Chem Biol, 7, pp. R207-R216
Poole, A., Penny, D., Sjöberg, B.-M. (2001): Confounded cytosine! Tinkering and the evolution of DNA. Nat Rev Mol Cell Biol, 2, pp. 147-151
Prasad R, Çağlayan M, Dai DP, Nadalutti CA, Zhao ML, Gassman NR, et al. (2017): DNA polymerase β: A missing link of the base excision repair machinery in mammalian mitochondria. DNA Repair. 60: 77–88.
Pursell ZF, Isoz I, Lundström EB, Johansson E, Kunkel TA (2007): Yeast DNA polymerase epsilon participates in leading-strand DNA replication. Science. 317 (5834): 127–130.
Raia P, Carroni M, Henry E, Pehau-Arnaudet G, Brule S, Beguin P, Henneke G, Lindahl E, Delarue M, Sauguet L. (2019a): Structure of the DP1-DP2 PolD complex bound with DNA and its implications for the evolutionary history of DNA and RNA polymerases. PLoS Biol. 17(1):e3000122.
Raia P, Delarue M, Sauguet L. (2019): An updated structural classification of replicative DNA polymerases. Biochem Soc Trans. 47(1):239–49.
Raychaudhury P, Basu AK (2011): Genetic requirement for mutagenesis of the G[8,5-Me]T cross-link in Escherichia coli: DNA polymerases IV and V compete for error-prone bypass. Biochemistry. 50 (12): 2330–2338.
Salas M, de Vega M. (2016): Protein-primed replication of bacteriophage Phi29 DNA. Enzymes 39: 137–67.
Sauguet L. (2019): The extended “two-barrel” polymerases superfamily: structure, function and evolution. J Mol Biol. 431(20):4167–83.
Sauguet L, Raia P, Henneke G, Delarue M. (2016): Shared active site architecture between archaeal PolD and multi-subunit RNA polymerases revealed by X-ray crystallography. Nat Commun. 7:12227.
Schuster, P. (2007): Molekulare Grundlagen der Evolution. Kann man die Wahrscheinlichkeit der Lebensentstehung abschätzen? Abendkolloquium: Frontiers of Science, Düsseldorf, 1.02.2007, 51. www.tbi.univie.ac.at/~pks. Zugr. a. 25.12.2007.
Schuster, P., Stadler, P.F. (1999): Nature and evolution of early replicons. In: DOMINGO, E./WEBSTER, R.G./HOLLAND, J.J. (Hg.) Origin and evolution of viruses. San Diego, 1–24.
Solé RV, Munteanu A, Rodriguez-Caso C, Macía J. (2007): Synthetic protocell biology: from reproduction to computation. Philos Trans R Soc Lond B Biol Sci. 29;362(1486):1727-39.
Spiegelman, S., Haruna, I., Holland, I. B., Beaudreau, G., Mills, D. R. (1965): The synthesis of a self-propagating and infectious nucleic acid with a purified enzyme. Proceedings of the National Academy of Sciences 54 (3), 919–927
Steitz, T. A. (1999): DNA polymerases: structural diversity and common mechanisms. J Biol Chem, 74, pp. 17395-17398
Stumpf JD, Copeland WC (2011): Mitochondrial DNA replication and disease: insights from DNA polymerase γ mutations. Cellular and Molecular Life Sciences. 68 (2): 219–233.
Sutton MD, Walker GC (2001): Managing DNA polymerases: coordinating DNA replication, DNA repair, and DNA recombination. Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8342–8349.
Swetina, J., Schuster, P. (1982): Self-replication with errors. A model for polynu-cleotide replication. Biophysical Chemistry 16, 329–345.
Takemura, M. (2001): Poxviruses and the origin of the eukaryotic nucleus. J Mol Evol, 52, pp. 419-425
Thiel, G., Lietz, M. (2004): Regulator neuronaler Gene: Zinkfingerprotein REST. Biologie in unserer Zeit. 34, 96–101.
Uchiyama, J. et al. (2014): Intragenus generalized transduction in Staphylococcus spp. by a novel giant phage. The ISME Journal 8 (9): S. 1949–1952.
Villarreal, L. P., DeFilippis, A. (2000): Hypothesis for DNA viruses as the origin of eukaryotic replication proteins. J Virol, 74, pp. 7079-7084
Woese C., Kandler O., Wheelis M. (1990): Towards a natural system of organisms : proposal for the domains Archae, Bacteria, and Eucarya, Proc. Natl. Acad. Sci. USA 87, 4576–4579.
Yamtich J, Sweasy JB (2010): DNA polymerase family X: function, structure, and cellular roles. Biochimica et Biophysica Acta (BBA) – Proteins and Proteomics. 1804 (5): 1136–1150.
Yoshida S, Suzuki R, Masaki S, Koiwai O. (1983): DNA primase 922 associated with 10S DNA polymerase alpha from calf thymus. 923. Biochim Biophys Acta 741:348–57.
Zhang L, Chan SS, Wolff DJ (2011): Mitochondrial disorders of DNA polymerase γ dysfunction: from anatomic to molecular pathology diagnosis. Archives of Pathology & Laboratory Medicine. 135 (7): 925–934.