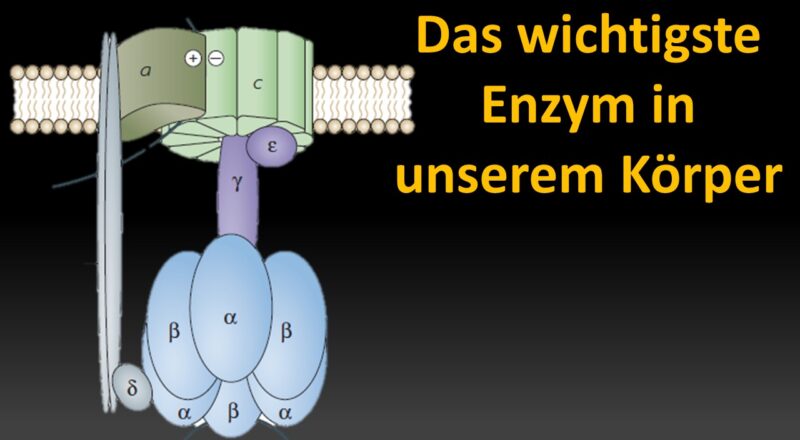
ATP ist sowas wie die Währung des Stoffwechsels (Abb. 1). Ohne ATP läuft nichts. ATP steht für Adenosin-Tri-Phosphat und speichert quasi die Energie, die wir zum Leben brauchen. Synthetisiert wird ATP mittels der ATP-Synthase, einem Transmembranprotein. ATP ist eine energiereiche Verbindung, deren Bildung der Zufuhr von Energie bedarf. Um diese Energie aufzubringen, koppelt die ATP-Synthase die ATP-Bildung mit dem energetisch begünstigten Transport von Protonen (oder anderen Ionen) entlang eines Protonengradientens über eine Membran. Das Enzym spielt im Stoffwechsel fast aller bekannten Organismen eine zentrale Rolle, da ATP ununterbrochen als Energieüberträger benötigt wird.
Text als pdf
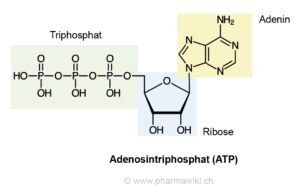
Abb. 1: ATP
Die Typen der ATP-Synthase
Es gibt drei Haupttypen der ATP-Synthasen (Abb. 2; Mulkidjanian et al. 2007, Stewart et al. 2014, Müller & Gruber 2003, Cross & Müller 2004, Kühlbrandt 2019, Song et al. 2013): Die häufigste ist die F-ATP-Synthase, die in der Zellmembran von Bakterien, Mitochondrien und Chloroplasten vorkommt. Dementsprechend wird angenommen, dass eukaryotische ATP-Synthasen vom F-Typ von den bakteriellen Vorläufern dieser Organellen abstammen (Hilario & Gogarten 1993, Gogarten & Taiz 1992, Mulkidjanian et al. 2008, Mahendrarajah et al. 2023)
Die ATP-Synthase setzt sich aus 8 bis 20 verschiedenen Untereinheiten zusammen. Sie gruppieren sich zu zwei Komplexen: Der wasserlösliche Komplex F1 katalysiert die Bildung von ATP. Der wasserunlösliche, in eine Membran eingebaute Komplex Fo transportiert Protonen.
Etwas abweichend vom Aufbau ist die V-ATP-Synthase, die sich in der Plasmamembran zahlreicher Zellorganellen befindet. Sie wird im Allgemeinen als das polare Gegenstück der F-ATP-Synthase angesehen. Nutzt die F-ATP-Synthase den Protonengradienten zur Herstellung von ATP, ist die V-ATP-Synthase hingegen eine Protonenpumpe, die die Energie der ATP-Hydrolyse nutzt, um einen Protonengradienten zu erzeugen (Nelson et al. 2000).
V-ATP-Synthasen finden sich in den Membranen vieler Organellen, wie Endosomen, Lysosomen und sekretorischen Vesikeln, wo sie eine Vielzahl von Aufgaben erfüllen, die für die Funktion dieser Organellen entscheidend sind. So treibt beispielsweise der von V-ATP-Synthasen erzeugte Protonengradient durch die Vakuolenmembran von Hefe die Kalziumaufnahme in die Vakuole durch ein H+/Ca2+-Antiportersystem an (Ohya et al. 1991). Bei der synaptischen Übertragung in Nervenzellen säuert die V-ATPSynthase synaptische Vesikel an (Wienisch & Klingauf 2006).
V-ATP-Synthasen kommen auch in den Plasmamembranen einer Vielzahl von Zellen vor, z. B. in den Interkalationszellen der Niere, in Osteoklasten (knochenresorbierenden Zellen), Makrophagen, Neutrophilen, Spermien, Mitteldarmzellen von Insekten und bestimmten Tumorzellen (Izumi et al. 2003). Plasmamembran-V-ATP-Synthasen sind an Prozessen wie der pH-Homöostase beteiligt. V-ATP-Synthasen in Spermien säuern das Akrosom an. Durch diese Ansäuerung werden Proteasen aktiviert, die zum Durchbohren der Plasmamembran der Eizelle erforderlich sind. V-ATPasen in der Plasmamembran von Osteoklasten pumpen Protonen auf die Knochenoberfläche, was für die Knochenresorption notwendig ist. In den Interkalationszellen der Niere pumpen V-ATPasen Protonen in den Urin und ermöglichen so die Rückresorption von Bikarbonat in das Blut. Darüber hinaus spielen V-ATPasen eine wichtige Rolle bei einer Vielzahl von biologischen Prozessen, wie z. B. bei der Freisetzung von Toxinen, dem Eindringen von Viren, dem Membran-Targeting, dem kontrollierten Zelltod und der Regulierung des pH-Werts des Zellplasmas (Emma et al. 1997).
Der dritte Typ, die A-ATP-Synthase, ähnelt der V-ATP-Synthase und kommt hauptsächlich bei Archaeen vor. Da sie in Stammbäumen zusammen clustern und sich strukturell ähneln, werden sie oft als Untergruppe des V-Typs angesehen. Das außergewöhnliche Vorkommen einiger Archaeenlinien mit F-Typ und einiger Bakterienlinien mit A-Typ wird als Ergebnis eines horizontalen Gentransfers angesehen (Hilario & Gogarten 1993, Cross & Müller 2004, Mulkidjanian et al. 2008, Williams et al. 2017, Nelson-Sathi et al. 2015).
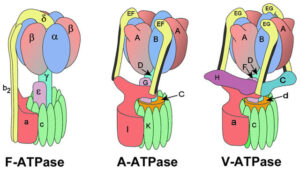
Abb. 2: Organisation der Untereinheiten in der Familie der rotierenden ATPasen. Bei der V-ATPase ist die D/F/d/c-Ringstruktur der Rotor, (AB)3/(EG)3/C/H/a ist der Stator. Äquivalente Untereinheiten innerhalb jedes Komplexes sind in derselben Farbe dargestellt.
Homologe Strukturen der ATP-Synthasen
Zwischen diesen Typen der ATP-Synthase gibt es auffällige Homologien, aber auch einige Untereinheiten, die nicht homolog sind (vgl. Abb. 3). Der Komplex Fo des F-Typs, also der hydrophobe Teil in der Lipidmembran, hat strukturelle Ähnlichkeiten mit jenen des V/A-Typs. Dies betrifft vor allem die in die Membran eingebettete c-Untereinheit.
Die Homologie zwischen der δ-Untereinheit des sowie dem hervorstehenden Teil der b-Untereinheit des F-Typs und den E- und G-Untereinheiten des V-Typs wurde aus schwacher Sequenzähnlichkeit abgeleitet (Supekova et al. 1995, Pallen et al. 2006).
Die a-Untereinheit des F-Typs hat hingegen keine signifikante Sequenzähnlichkeit mit dem des V/A-Typs, es bestehen aber strukturelle und mechanistische Analogien (Lolkema et al. 2003, Perzov et a. 2001).
Beide Typen der a-Untereinheit enthalten einen streng konservierten Argininrest in der Mitte der vorletzten Transmembranhelix. Die elektrostatische Wechselwirkung dieses Arginins mit den konservierten Carboxylateinheiten, die in den rotierenden c-Untereinheiten enthalten sind, ist für den Ionentransport entscheidend (Kawano et al. 2002, Kawasaki-Nishi et al. 2001). Hinsichtlich der Domänenorganisation ähnelt die a-Untereinheit des V/A-Typs einer Fusion der a-Untereinheit des F-Typs mit seiner b-Untereinheit. Eine Homologie der a-Untereinheit bei den ATP-Synthasetypen ist nicht ganz sicher (Mulkidjanian et al. 2007).
Die Untereinheiten α und β des F-Typs bilden die Kopfstruktur des F1-Komplex und sind ein Hexamer mit 6 Bindungsstellen. Sie entsprechen den A und B-Untereinheiten des V/A-Typs und diese Untereinheit ist in beiden Typen hochgradig konserviert (Schafer et al. 2006, Gibbons et al. 2000).
Im Gegensatz dazu sind die Untereinheiten der zentralen Drehachse (beim F-typ die γ-Untereinheit), der die Kopfstruktur mit der Membraneinheit verbindet, nicht homolog und strukturell verschieden. Dies wird nicht nur durch das Fehlen signifikanter Sequenzähnlichkeiten deutlich, sondern auch durch das Vorhandensein unterschiedlicher Faltungen in diesen Untereinheiten (Gibbons et al. 2000, Drory & Nelson 2006).
Die zentrale Drehachse des F- und V/A-Typs ist für die Rotationskatalyse unerlässlich, und so hat der deutliche Mangel an Sequenzähnlichkeiten zwischen beiden Typen erhebliche Auswirkungen auf deren evolutionären Ursprung.
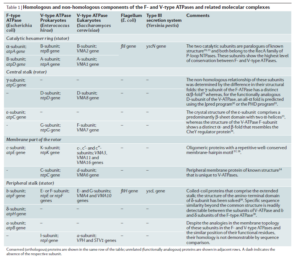
Abb. 3: Homologe und nicht-homologe Komponenten der F- und V-Typ-ATPasen und verwandter molekularer Komplexe
Viele Untereinheiten der ATP-Synthasen haben Homologien zu anderen Proteinen:
Die katalytischen Hexamere aus den α und β- bzw. A und B-Einheiten der ATP-Synthasen sind homolog zu hexameren Helicasen (Walker 1998), genauer gehören diese katalytischen Hexamere zur selben Familie wie Rho, einer bakteriellen RNA-Helikase, die als Transkriptionsterminationsfaktor fungiert (Abb. 4; Adelman et al. 2006, Kaplan & O’Donnell 2003, Gocheva et al. 2015, Molodtsov et al. 2023). Die Struktur von Rho besteht aus einem hexameren Ring, der der Struktur der ATP-Synthasen ähnelt und zwei RNA-Bindungsmotive enthält, die beide auf das Zentrum der Ringstruktur ausgerichtet sind (Skordalakes & Berger 2003, Kaplan & O’Donnell 2003). Der hexamere Ring ist charakteristisch für eine Vielzahl von DNA- und RNA-Helikasen (Patel & Picha 2000) sowie für verschiedene nukleinsäureabhängige ATPasen, die in Nukleinsäure- und Proteintranslokationssystemen funktionieren (Gomis-Ruth et al. 2001, Cabezon & de la Cruz 2006, Aussel et al. 2002, Iyer et al. 2004, Juuti et al. 1998, Pirttimaa et al. 2002, Kainov et al. 2003, 2006, Wall & Kaiser 1999, Merz, et al. 2000, Kaplan & O’Donnell 2003, Gocheva et al. 2015, Molodtsov et al. 2023). Die Rotationsbewegung mehrerer Helikasen beim Abwickeln von DNA oder RNA wurde nachgewiesen (Laskey & Madine 2003, Lee & Yang 2006, Skordalakes & Berger 2006).
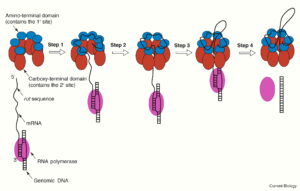
Abb. 4: rho-Helicase, die bei der Termination der Transkription fungiert und die Homologien zur α und β- bzw. A und B-Einheiten der ATP-Synthasen aufweist. In Schritt 1 bindet die aminoterminale Domäne des Rho-Faktors (blau), die die 1° mRNA-Bindungsstelle enthält, an die “rut”-Sequenz des mRNA-Transkripts. Der Rho-Faktor ist als offener Ring dargestellt, kann sich aber in diesem Schritt im Gleichgewicht zwischen einem offenen und einem geschlossenen Zustand befinden. In Schritt 2 bindet die carboxyterminale Domäne von Rho (orange), die die 2°-mRNA-Bindungsstelle enthält, die mRNA stromabwärts von rut, und der hexamere Ring von Rho schließt sich. In Schritt 3 bindet die carboxyterminale Domäne von Rho zyklisch ATP und hydrolysiert es, um sich entlang der mRNA in Richtung 5′ zu 3′ zu bewegen. Die aminoterminale Domäne kann sich während dieses Schritts von der Rut ablösen. In Schritt 4 agiert der Rho-Faktor als Helikase, um die ATP-Bindung und -Hydrolyse mit der Abspulung der Nukleinsäure zu verbinden. Dies führt zu einer Loslösung der mRNA von der genomischen DNA und der RNA-Polymerase (rosa). Abbildung aus Kaplan & O’Donnell (2003).
Zweitens gibt es eine homologe Beziehung zwischen den ATP-Synthasen und den Untereinheiten der bakteriellen Flagellenmotoren und Typ-III-Sekretionssysteme (T3SS, Abb. 5 & 6 Cornelis 2006, Deng et al. 2017, Diepold & Armitage 2015, Minamino et al. 2022). Diese Beziehung lässt sich über die katalytischen Untereinheiten (Vogler et al. 1991, Diepold & Armitage 2015, Minamino et al. 2022) und die Untereinheiten des peripheren Stiels der ATP-Syntasen vom F- und V-Typ (Pallen et al. 2006) nachverfolgen. Es wurde vorgeschlagen, der Flagellum-Motor und das Typ-III-Sekretionssystem durch die Rekrutiertung der ATP-Synthase für deren Funktion entwickelt hat (Pallen et al. 2006, Aizawa 2001, Blocker et al. 2003, Minamino et al. 2022). In diesen beiden Systemen fehlt jedoch ein Gegenstück der zentralen Drehachse der ATP-Synthasen, was darauf hindeutet, dass die rekrutierte Einheit ursprünglich eine Proteintranslokase und keine ATP-Synthase gehandelt haben könnte.
Die Verbreitung von Flagellenmotoren und Typ-III-Sekretionssystem (T3SS) ist außerdem auf eine taxonomisch verstreute Gruppe von Bakterien beschränkt, was in deutlichem Gegensatz zum universellen Sec-System der Proteintranslokation steht (Pohlschroder et al. 2005). Dies könnte auf einen späten Ursprung der Flagellarmotoren und der T3SS-Maschinerie hindeuten.
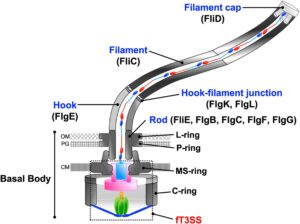
Abb. 5: Schematische Darstellung des bakteriellen Flagellums. Das bakterielle Flagellum besteht aus basalen Körperringen, nämlich dem C-Ring, dem MS-Ring, dem L-Ring und dem P-Ring, sowie aus einer axialen Struktur, die aus dem Stab, dem Haken, der Haken-Filament-Verbindung, dem Filament und der Filamentkappe besteht. Um die axiale Struktur jenseits der Zytoplasmamembran aufzubauen, werden die axialen Flagellarproteine durch das fT3SS transloziert, diffundieren durch einen schmalen zentralen Kanal und sammeln sich an der Spitze der wachsenden Struktur. OM, äußere Membran; PG, Peptidoglykanschicht; CM, zytoplasmatische Membran. Abbildung aus Minamina et al. (2022).
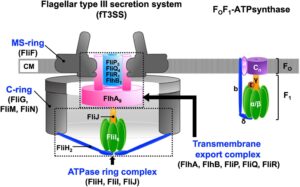
Abb. 6: Schematische Darstellung des flagellaren Typ-III-Exportapparats und der FOF-ATP-Synthase. Das flagellare Typ-III-Sekretionssystem (fT3SS) besteht aus fünf Membranproteinen, FlhA, FlhB, FliP, FliQ und FliR, und drei zytoplasmatischen Proteinen, FliH, FliI und FliJ. FlhA, FlhB, FliP, FliQ und FliR bilden einen transmembranen Exportkomplex im MS-Ring des Basalkörpers der Geißel. FliH, FliI und FliJ bilden einen cytoplasmatischen ATPase-Ring. Der FliI6-FliJ1-Ringkomplex ist strukturell dem α3β3γ1-Ringkomplex der FOF1-ATP-Synthase ähnlich. Die N-terminalen und C-terminalen Domänen von FliH ähneln strukturell den b- bzw. δ-Untereinheiten der FOF1-ATP-Synthase. Das FliH-Dimer fungiert als peripherer Stiel, der den FliI6-FliJ-Ringkomplex auf ähnliche Weise an der Basis des Flagellums verankert, wie die b- und δ-Untereinheiten der FOF1-ATP-Synthase den α3β3γ-Ringkomplex mit dem membrangebundenen FO verbinden. Die Stöchiometrie des c-Rings variiert dramatisch von c8 bis mindestens c15. CM, Zytoplasmamembran. Abbildung aus Minamina et al. (2022).
Diese Systeme funktionieren auch nach anderen Prinzipien als die Sec-Systeme (Abb. 7; Mori & Ito 2001, Driessen et al. 1998). Die Sec-Maschinerie interagiert z. B. mit dem Signalerkennungspartikel (SRP) und transloziert Proteine sowohl während als auch nach der Translation (Pohlschroder et al. 2005). Im Gegensatz dazu ist die Sekretion mit Hilfe von Flagellenmotoren und T3SS SRP-unabhängig und transloziert hauptsächlich posttranslationale Proteine (Wilharm et al. 2004, 2007). Daher ist es sinnvoll vorzuschlagen, dass beide Arten von Sekretionssystemen Nachkommen ursprünglicher Protein-Translokationsmaschinen sind. Eine auffällige Analogie in der Evolution dieser Systeme ist, dass beide offenbar RNA-Helikasen rekrutiert haben, die die ATP-Hydrolyse mit der Proteintranslokation koppeln. SecA, die universelle Kopplungs-ATPase der bakteriellen Sec-Systeme, ist ein Homolog der Helikasen der Superfamilie 2 (Koonin & Gorbalenya 1992, Keramisanou et al. 2006).
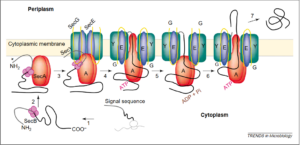
Abb. 7: Das Sec-Proteintransport-System. Sec-Faktoren und Translokationsprozesse. Das Präprotein ist durch eine schwarze Linie dargestellt, der graue Bereich zeigt die Signalsequenz. Schritte 1-3, Targeting. Eine Signalsequenz und ihr unmittelbarer carboxy-terminaler Bereich bilden eine Initiationsdomäne, die von der Sec-Maschinerie erkannt wird. SecB, das Sec-System-spezifische Chaperon, schleust das Präprotein in den Sec-Translokationsweg ein und lenkt den gebundenen Vorläufer durch seine Fähigkeit, SecA zu binden, aktiv auf die Translokase. Das SecA-tragende Präprotein bindet dann an die Membran an einer hochaffinen SecA-Bindungsstelle. SecY, SecE und SecG bilden einen hetero-trimeren Komplex, SecYEG, der einen Weg (“Kanal”) für die Polypeptidbewegung darstellt. Schritte 4 und 5, Initiierung. Der Initiierungsschritt erfordert ATP, aber nicht dessen Hydrolyse. Schritt 6, Fortführung. Die Fortsetzung der Translokation erfordert Zyklen der ATP-Hydrolyse und/oder der protonenmotorischen Kraft durch die Membran. Man geht davon aus, dass die Translokation schrittweise erfolgt, mit einem Schritt von 20-30 Aminosäureresten. Schritt 7, Vollendung. Bislang ist nur wenig über den Vervollständigungsprozess bekannt, der auf der periplasmatischen Seite stattfindet und zur Freisetzung und/oder Faltung des Substratproteins in den periplasmatischen Raum führt. Abbildung aus Mori & Ito (2001).
Entstehung der ATP-Synthase
Wie könnte die ATP-Synthase entstanden sein? Folgendes Szenario ist denkbar und wird durch das Datenmaterial unterstützt (Abb. 8):
Die Kombination einer RNA-Helikase mit einem Membrantransporter führte nicht sofort zur Bildung einer Ionen-übertragenden ATP-Synthase. Stattdessen wurde als Zwischenschritt eine ATP-getriebene Membran-RNA-Translokase gebildet. Diese Translokase könnte durch direktes Andocken der RNA-Helikase an den Transmembrankanal funktioniert haben. In der Tat ähnelt die strukturelle Organisation von Nukleinsäure-Translokationsvorrichtungen (wie dem Trw-Komplex) stark der einer Protonen-übertragenden ATP-Syynthase, bei der die zentrale Drehachse entfernt wurde. Interessanterweise haben neuere Forschungen gezeigt, dass der Zusammenbau des ATPase-Hexamers durch DNA stimuliert wird (Cabezon & de la Cruz 2006, Tato et al. 2005).
Der nächste Schritt in diesem vorgeschlagenen Szenario war die Entwicklung einer Proteintranslokase aus der RNA-Translokase. In diesem Stadium könnten weitere membrangebundene Untereinheiten rekrutiert worden sein, die das Andocken erleichtern und elastische Verbindungen zwischen dem katalytischen Hexamer und dem Membranteil herstellt. In der primitiven Zelle könnten die beiden Teile der Translokase getrennt voneinander existiert haben, bis die Translokation erforderlich war; ein Merkmal, das bei eukaryotischen ATP-Synthasen vom V-Typ erhalten geblieben ist (Beyenbach & Wieczorek 2006, Drory & Nelson 2006). Die Entwicklung einer Ionen-übertragenden ATP-Synthase aus der hypothetischen Proteintranslokase könnte auf eine Reihe von Aminosäure-Ersetzungen zurückzuführen sein, die die Hydrophobizität des inneren Raums erhöhten. Dies hätte die Proteintranslokation behindert und die Wahrscheinlichkeit erhöht, dass ein transportiertes Protein in der Translokase gefangen wird. Weil das Protein nun in der Translokase gefangen ist, würde die Drehung des Hexamers bei der Spaltung von ATP eine Drehung des gesamten Translokase verursachen, inklusive der membrangebundenen Teile. Diese Drehung könnte durch die in die Membran eingebetteten geladenen Aminosäureseitenketten, die die Membranuntereinheiten zusammenhielten, mit dem Ionentransport durch die Membran gekoppelt worden sein. Angesichts des energetischen Nutzens eines Ionengradienten wurden aus vielen möglichen Rotationsmodi diejenigen ausgewählt, die die Speicherung der Energie der ATP-Hydrolyse in Form eines transmembranen Ionengradienten mit dem richtigen Vorzeichen ermöglichten. Der Übergang von einer Proteintranslokase zu einer ATP-gesteuerten Ionentranslokase könnte durch die permanente Rekrutierung von Untereinheiten durch den Einbau der entsprechenden Gene vollzogen worden sein.
Die Frage bleibt aber nun offen: Welchen Typ hatte der gemeinsame Vorfahre der ATP-Synthasen? Drei Möglichkeiten stehen zur Auswahl:
Eine Möglichkeit ist, dass der gemeinsame Vorfahre eine Proteintranslokase war. In diesem Szenario wird die zentrale Drehachse sowohl in der bakteriellen als auch in der archaeischen Linie unabhängig von der Proteintranslokase rekrutiert, wodurch die ATP-Synthasen vom F- bzw. V-Typ entstehen; der Übergang von einer Proteintranslokase zu einer Ionen-übertragenden ATP-Synthase hätte jedoch ebenfalls unabhängig erfolgen müssen. Weiterhin besteht das Problem, dass LUCA, wie wir in vorherigen Beiträgen festgestellt haben, über eine ATP-Synthase verfügen musste. Es ist ziemlich unwahrscheinlich, dass solch ein komplexes Molekül, mit vielen homologen Strukturen, konvergent entstanden ist.
Eine andere Möglichkeit ist, dass der Übergang von der Proteintranslokase zur Ionen-übertragenden ATP-Synthase bereits im gemeinsamen Vorfahren von Archaeen und Bakterien stattgefunden hat, aber die zentrale Drehachse (deren strukturellen Anforderungen flexibel sind) wurde in einer der Linien durch ein nicht verwandtes Protein ersetzt.
Ein drittes Szenario, das am plausibelsten erscheint, postuliert, dass eine Ionen-übertragende ATP-Synthase, die ein gefangenes translokiertes Protein in der Rolle der zentralen Drehachse enthielt, ein evolutionäres Zwischenglied zwischen der Proteintranslokase und einer echten Membran-ATP-Synthase war und der gemeinsame Vorfahre sowohl des F- als auch der V-Typs- ist. In diesem Szenario wurde die Rolle der zentralen Drehachse im gemeinsamen Vorfahren von verschiedenen Proteinen zu verschiedenen Zeiten wahrgenommen, bis sich sowohl in Archaeen als auch in Bakterien durch den unabhängigen Einbau der Gene, die jetzt in den ATPase-Operonen vom F- bzw. V-Typ vorhanden sind, dauerhafte und unterschiedliche Komponenten entwickelten. Das heißt die gefangenen Proteine waren nicht spezifisch und erst mit der Entstehung der beiden Domänen kam es zur Spezialisierung. Dieses Szenario erfordert keine Ad-hoc-Hypothesen über die unabhängige Evolution von Ionen-übertragenden Maschinen sowohl in Bakterien als auch Archaeen. Der einzige bemerkenswerte Unterschied besteht in dem Stadium, in dem die Evolutionspfade der ATP-Synthasen vom F- und V-Typ divergieren.
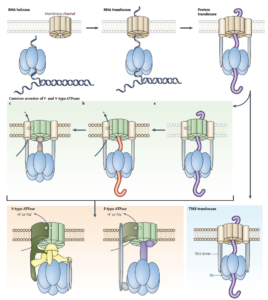
Abb. 8: Evolution der ATP-Synthase. Hypothetische, halbdurchlässige Membranen aus der Vorzeit sind durch leere Kreise und leichte gebogene Linien dargestellt, während moderne, ionendichte Membranen durch gefüllte Kreise und dichte gebogene Linien dargestellt sind. Das Vorhandensein von zwei peripheren Stielen in der primordialen Proteintranslozase und den Flagellarmotor- und Typ-III-Sekretionssystemen (T3SSs) beruht auf der Annahme, dass ein Translokationssystem mit nur einem peripheren Stiel in Abwesenheit des translozierten Substrats instabil sein würde. Die Beteiligung von zwei FliH-Untereinheiten in jedem peripheren Stiel beruht auf der Fähigkeit von FliH-Dimeren, einen Komplex mit einer FliI-Untereinheit zu bilden. Die hellgrüne Tafel in der Mitte zeigt die drei aufeinanderfolgenden evolutionären Zwischenstufen (a-c), von denen jede als gemeinsamer Vorfahre der F- und V-Typ-ATPasen in den drei im Haupttext beschriebenen alternativen evolutionären Szenarien betrachtet wird. Die violetten Röhren bezeichnen die translozierten, teilweise entfalteten Proteine. Die rote Röhre kennzeichnet ein transloziertes Protein, das im Kanal der Membrantranslozase gefangen ist. Die Namen der Untereinheiten sind nur für den Flagellarmotor und die T3SS-Maschinen angegeben.
Die vorgeschlagene evolutionäre Beziehung zwischen RNA-Helikasen, Proteintranslokasen und ATP-Synthasen vom F- und V/A-Typ scheint mit der Topologie des phylogenetischen Baums der RecA-Superfamilie der P-Loop-ATPasen vereinbar zu sein. Die phylogenetische Analyse vielfältiger, alter Proteinfamilien ist anfällig für lange Verzweigungen und andere Artefakte und sollte mit Vorsicht interpretiert werden (Philippe & Laurent 1998, Gribaldo & Philippe 2002, Yu & Egelman 1997, Iyer et al. 2004); dennoch wird diese spezielle Baumtopologie durch Bootstrap-Replikationen stark unterstützt (Abb. 9).
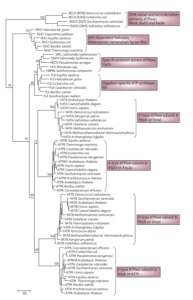
Abb. 9: Phylogenetischer Baum der katalytischen Untereinheiten der F- und V-Typ-ATPasen und verwandter P-Schleifen-ATPasen.
Ursprünge der Membran und der Zellen
Die Vorstellung, dass der gemeinsame Vorfahre der ATP-Synthasen vom F- und V-Typ eine andere Funktion hatte, z. B. die Translokation von Nukleinsäuren oder Proteinen, steht im Einklang mit den Unterschieden in der Membranbiogenese (Martin & Russel 2003, Pereto et al. 2004) und den DNA-Replikationssystemen (Mushegian & Koonin 1996, Edgell & Doolittle 1997, Leipe et al. 1999) zwischen Archaeen und Bakterien. Diese Beobachtungen führten zu radikalen Vorschlägen über die Natur des letzten gemeinsamen Vorfahren (Last Universal Common Ancestor, LUCA), nämlich dass er weder über eine Membranorganisation (Martin & Russel 2003) noch über eine DNA-Replikation (Leipe et al. 1999) verfügte und folglich keine typische Zelle war (Koonin & Martin 2005). Darüber hinaus scheint der Ursprung der Zellmembran selbst ein Dilemma zu sein: Damit eine Membran in einer Zelle funktionieren kann, muss sie zumindest mit einem minimalen Repertoire an Transportsystemen ausgestattet sein, aber es ist unklar, wie sich solche Systeme in Abwesenheit einer Membran entwickeln konnten. Das Modell eines nicht-membrangebundenen LUCA stößt jedoch auf erhebliche Schwierigkeiten. Das Hauptproblem ist die ubiquitäre Erhaltung mehrerer Membranproteine und komplexer, membranassoziierter molekularer Maschinen wie des SRP, der Kernproteine des Sec-Systems und der F- und V-Typ-ATPasen selbst (Koonin & Martin 2005, Jekely 2006). Die primitive Funktion einer RNA- und Proteintranslokase, die oben für einen Vorfahren der ATPasen vom F- und V-Typ vorgeschlagen wurde, könnte eine mögliche Lösung für das Rätsel der Urmembran darstellen. Den modernen ionenundurchlässigen Membranen könnten ursprüngliche, ionendurchlässige Proto-Membranen vorausgegangen sein, die in der Lage waren, RNA und Proteine einzuschließen (Deamer 1997, Ourisson & Nakatani 1994, Gotoh et al. 2006). Dieses Szenario lernten wir in der letzten Episode kennen. Diese Strukturen hätten das Potenzial gehabt, die ersten Membranenzyme zu beherbergen – zunächst Translokasen von Makromolekülen und später ATP-Synthasen und Transporter für kleine Moleküle. Es ist vielfach argumentiert worden, dass der weit verbreitete horizontale Genaustausch zwischen den ursprünglichen genetischen Systemen sowohl ein wesentliches Merkmal der frühen, präzellulären Evolution als auch eine notwendige Voraussetzung für die Evolution immer komplexerer Einheiten war (Koonin & Martin 2005, Woese 2002, Vestigian et al. 2006). Damit dies in Verbindung mit der Evolution von biologischen Membranen geschehen konnte, scheinen Nukleinsäure-Translokatoren eine wesentliche Voraussetzung zu sein. Diese ersten Nukleinsäuretranslokasen vermittelten den Import und Export von RNA-Molekülen in virusähnlichen Gebilden, die mehrere RNA-Segmente, eine primitive Membran und möglicherweise eine kapsidartige Struktur enthielten (Koonin et al. 2006). Zumindest konzeptionell könnten solche primitiven Translokasen analog zur hexameren P4-ATPase gewesen sein, die in modernen, lipidhaltigen dsRNA-Bakteriophagen nachgewiesen wurde (Kainov et al. 2004, 2006). Dieses Szenario für die Entstehung der ATP-Synthasen vom F- und V-Typ beschreibt eine Abfolge von Ereignissen, die von einer löslichen Helikase und einem Membrankanal über RNA- und Proteintranslokasen bis hin zu den Ionen-übertragenden ATP-Synthasen reicht. Die Divergenz dieser Szenarien auf der vorletzten Stufe, die zu mehreren Alternativen hinsichtlich der Art des gemeinsamen Vorfahren der ATP-Synthasen vom F- und V-Typ führt, hat jedoch ganz andere Auswirkungen auf den Status der Membranen in LUCA. Eine Proteintranslokase als gemeinsamer Vorfahre impliziert primitive, ionendurchlässige Membranen bei LUCA.
Dieses Szenario gibt Hinweise darauf, dass Stoffwechsel, Chemiosmose und die Gewinnung von ATP wichtige Aspekte bei der Entstehung des Lebens sind. Diese können aber nicht unabhängig von weiteren wichtigen Aspekten des Lebens bestehen bleiben: der Rolle der Nukleinsäuren. Die chemisch-physikalischen Bedingungen der Urerde ermöglichten spontane Stoffwechselreaktionen. Doch ohne die Existenz von komplexeren Proteineinheiten können komplexere Stoffwechselreaktionen nicht stattfinden. Für solche Proteine braucht es aber den genetischen Code, welche die Informationen für den Aufbau der Proteine speichert. Dafür braucht es die Nukleinsäuren: RNA und DNA. Wie wir in vorherigen teilen kennengelernt haben können auch diese unter den Bedingungen der Urerde entstanden sein – allen voran die RNA. Um diese RNA-Welt und der daraus sich entwickelnden DNA-Welt und den genetischen Code soll es in den nächsten Episoden gehen. Wichtig ist anzumerken: die RNA-Welt steht nicht in Konkurrenz zu der in den vorherigen Abschnitten beschriebenen Szenarien der Stoffwechsel-zuerst-Welt (Metabolism first world). Wichtiger als die Frage, was zuerst da war: RNA oder Stoffwechsel (beide entstanden eigentlich zeitgleich und parallel) ist es zu verstehen, wie beide Welten sich vereinigen und zur komplexen Maschinerie wurden, die wir Leben nennen.
Literatur
Adelman, J. L. et al. (2006): Mechanochemistry of transcription termination factor Rho. Mol. Cell 22, 611–621.
Aizawa, S. I. (2001): Bacterial flagella and type III secretion systems. FEMS Microbiol. Lett. 202, 157–164.
Aussel, L. et al. (2002): FtsK is a DNA motor protein that activates chromosome dimer resolution by switching the catalytic state of the XerC and XerD recombinases. Cell 108, 195–205.
Beyenbach, K. W. & Wieczorek, H. (2006): The V-type H+ ATPase: molecular structure and function, physiological roles and regulation. J. Exp. Biol. 209, 577–589.
Blocker, A., Komoriya, K. & Aizawa, S. (2003): Type III secretion systems and bacterial flagella: insights into their function from structural similarities. Proc. Natl Acad. Sci. USA 100, 3027–3030.
Cabezon, E. & de la Cruz, F. TrwB: (2006): an F1-ATPase-like molecular motor involved in DNA transport during bacterial conjugation. Res. Microbiol. 157, 299–305.
Cornelis, G. (2006): The type III secretion injectisome. Nat Rev Microbiol 4, 811–825.
Cross, R. L. & Müller, V. (2004): The evolution of A-, F-, and V-type ATP synthases and ATPases: reversals in function and changes in the H /ATP coupling ratio. FEBS Lett. 576, 1–4.
Deamer, D. W. (1997): The first living systems: a bioenergetic perspective. Microbiol. Mol. Biol. Rev. 61, 239–261.
Deng, W., Marshall, N., Rowland, J. et al. (2017): Assembly, structure, function and regulation of type III secretion systems. Nat Rev Microbiol 15, 323–337.
Diepold A, Armitage JP. (2015): Type III secretion systems: the bacterial flagellum and the injectisome. Philos Trans R Soc Lond B Biol Sci. 5;370(1679):20150020.
Driessen, A. J., Fekkes, P., & van der Wolk, J. P. (1998). The Sec system. Current Opinion in Microbiology, 1(2), 216–222.
Drory, O. & Nelson, N. (2006): The emerging structure of vacuolar ATPases. Physiology (Bethesda) 21, 317–325.
Edgell, D. R. & Doolittle, W. F. (1997): Archaea and the origin(s) of DNA replication proteins. Cell 89, 995–998.
Emma B, Forest O, Barry B (1997): Mutations of pma-1, the Gene Encoding the Plasma Membrane H+ATPase of Neurospora crassa, Suppress Inhibition of Growth by Concanamycin A, a Specific Inhibitor of Vacuolar ATPases. The Journal of Biological Chemistry. 272 (23): 14776–14786.
Gibbons, C., Montgomery, M. G., Leslie, A. G. & Walker, J. E. (2000): The structure of the central stalk in bovine F1-ATPase at 2.4 Å resolution. Nature Struct. Biol. 7, 1055–1061.
Gocheva V, Le Gall A, Boudvillain M, Margeat E, Nollmann M. (2015): Direct observation of the translocation mechanism of transcription termination factor Rho. Nucleic Acids Res. 27;43(4):2367-77.
Gogarten, J. P. & Taiz, L. (1992): Evolution of proton pumping ATPases: rooting the tree of life. Photosynth. Res. 33, 137–146.
Gomis-Ruth, F. X. et al. (2001): The bacterial conjugation protein TrwB resembles ring helicases and F1-ATPase. Nature 409, 637–641.
Gotoh, M. et al. (2006): Membrane properties of branched polyprenyl phosphates, postulated as primitive membrane constituents. Chem. Biodivers. 3, 434–455.
Gribaldo, S. & Philippe, H. (2002): Ancient phylogenetic relationships. Theor. Popul. Biol. 61, 391–408.
Hilario, E. & Gogarten, J. P. (1993): Horizontal transfer of ATPase genes — the tree of life becomes a net of life. Biosystems 31, 111–119.
Iyer, L. M., Leipe, D. D., Koonin, E. V. & Aravind, L. (2004): Evolutionary history and higher order classification of AAA+ ATPases. J. Struct. Biol. 146, 11–31.
Iyer, L. M., Makarova, K. S., Koonin, E. V. & Aravind, L. (2004): Comparative genomics of the FtsK-HerA superfamily of pumping ATPases: implications for the origins of chromosome segregation, cell division and viral capsid packaging. Nucleic Acids Res. 32, 5260–5279.
Izumi H, Torigoe T, Ishiguchi H, Uramoto H, Yoshida Y, Tanabe M, Ise T, Murakami T, Yoshida T, Nomoto M, Kohno K (2003): Cellular pH regulators: potentially promising molecular targets for cancer chemotherapy. Cancer Treatment Reviews. 29 (6): 541–9.
Jekely, G. (2006): Did the last common ancestor have a biological membrane? Biol. Direct 1, 35.
Juuti, J. T., Bamford, D. H., Tuma, R. & Thomas, G. J. Jr. (1998): Structure and NTPase activity of the RNA-translocating protein (P4) of bacteriophage phi 6. J. Mol. Biol. 279, 347–359.
Kainov, D. E. et al. (2003): RNA packaging device of double-stranded RNA bacteriophages, possibly as simple as hexamer of P4 protein. J. Biol. Chem. 278, 48084–48091.
Kainov, D. E., Lisal, J., Bamford, D. H. & Tuma, R. (2004): Packaging motor from double-stranded RNA bacteriophage phi12 acts as an obligatory passive conduit during transcription. Nucleic Acids Res. 32, 3515–3521.
Kainov, D. E., Tuma, R. & Mancini, E. J. (2006): Hexameric molecular motors: P4 packaging ATPase unravels the mechanism. Cell. Mol. Life Sci. 63, 1095–1105.
Kaplan DL, O’Donnell M. (2003): Rho factor: transcription termination in four steps. Curr Biol. 13 (18): R714-6.
Kawano, M., Igarashi, K., Yamato, I. & Kakinuma, Y. (2002): Arginine residue at position 573 in Enterococcus hirae vacuolar-type ATPase NtpI subunit plays a crucial role in Na+ translocation. J. Biol. Chem. 277, 24405–24410.
Kawasaki-Nishi, S., Nishi, T. & Forgac, M. (2001): Arg-735 of the 100-kDa subunit a of the yeast V-ATPase is essential for proton translocation. Proc. Natl Acad. Sci. USA 98, 12397–12402.
Keramisanou, D. et al. (2006): Disorder-order folding transitions underlie catalysis in the helicase motor of SecA. Nature Struct. Mol. Biol. 13, 594–602.
Koonin, E. V. & Gorbalenya, A. E. (1992): Autogenous translation regulation by Escherichia coli ATPase SecA may be mediated by an intrinsic RNA helicase activity of this protein. FEBS Lett. 298, 6–8.
Koonin, E. V. & Martin, W. (2005): On the origin of genomes and cells within inorganic compartments. Trends Genet. 21, 647–654.
Koonin, E. V., Senkevich, T. G. & Dolja, V. V. (2006): The ancient virus world and evolution of cells. Biol. Direct 1, 29.
Kühlbrandt, W. (2019): Structure and mechanisms of F-Type ATP synthases. Annu. Rev. Biochem. 88, 515–549.
Laskey, R. A. & Madine, M. A. (2003): A rotary pumping model for helicase function of MCM proteins at a distance from replication forks. EMBO Rep. 4, 26–30.
Lee, J. Y. & Yang, W. (2006): UvrD helicase unwinds DNA one base pair at a time by a two-part power stroke. Cell 127, 1349–1360.
Leipe, D. D., Aravind, L. & Koonin, E. V. (1999): Did DNA replication evolve twice independently? Nucleic Acids Res. 27, 3389–3401.
Lolkema, J. S., Chaban, Y. & Boekema, E. J. (2003): Subunit composition, structure, and distribution of bacterial V-type ATPases. J. Bioenerg. Biomembr. 35, 323–335.
Mahendrarajah, T.A., Moody, E.R.R., Schrempf, D. et al. (2023): ATP synthase evolution on a cross-braced dated tree of life. Nat Commun 14, 7456.
Martin, W. & Russell, M. J. (2003): On the origins of cells: a hypothesis for the evolutionary transitions from abiotic geochemistry to chemoautotrophic prokaryotes, and from prokaryotes to nucleated cells. Phil. Trans. R. Soc. Lond. B 358, 59–85.
Merz, A. J., So, M. & Sheetz, M. P. (2000): Pilus retraction powers bacterial twitching motility. Nature 407, 98–102.
Minamino T, Kinoshita M, Namba K. (2022): Insight Into Distinct Functional Roles of the Flagellar ATPase Complex for Flagellar Assembly in Salmonella. Front Microbiol. 4;13:864178.
Molodtsov, V., Wang, C., Firlar, E. et al. (2023): Structural basis of Rho-dependent transcription termination. Nature 614, 367–374.
Mori H, Ito K. (2001): The Sec protein-translocation pathway. Trends Microbiol. 9(10):494-500.
Mulkidjanian, A. Y., Galperin, M. Y., Makarova, K. S., Wolf, Y. I. & Koonin, E. V. (2008): Evolutionary primacy of sodium bioenergetics. Biol. Direct 3, 13.
Mulkidjanian, A. Y., Makarova, K. S., Galperin, M. Y. & Koonin, E. V. (2007): Inventing the dynamo machine: the evolution of the F-type and V-type ATPases. Nat. Rev. Microbiol. 5, 892–899.
Müller, V. & Grüber, G. (2003): ATP synthases: structure, function and evolution of unique energy converters. Cell. Mol. Life Sci. 60, 474–494.
Mushegian, A. R. & Koonin, E. V. (1996): A minimal gene set for cellular life derived by comparison of complete bacterial genomes. Proc. Natl Acad. Sci. USA 93, 10268–10273.
Nelson N, Perzov N, Cohen A, Hagai K, Padler V, Nelson H (2000): The cellular biology of proton-motive force generation by V-ATPases. The Journal of Experimental Biology. 203 (Pt 1): 89–95.
Nelson-Sathi, S. et al. (2015): Origins of major archaeal clades correspond to gene acquisitions from bacteria. Nature 517, 77–80.
Ohya Y, Umemoto N, Tanida I, Ohta A, Iida H, Anraku Y (1991): Calcium-sensitive cls mutants of Saccharomyces cerevisiae showing a Pet- phenotype are ascribable to defects of vacuolar membrane H(+)-ATPase activity. The Journal of Biological Chemistry. 266 (21): 13971–7.
Ourisson, G. & Nakatani, Y. (1994): The terpenoid theory of the origin of cellular life: the evolution of terpenoids to cholesterol. Chem. Biol. 1, 11–23.
Pallen, M. J., Bailey, C. M. & Beatson, S. A. (2006): Evolutionary links between FliH/YscL-like proteins from bacterial type III secretion systems and second-stalk components of the F0F1 and vacuolar ATPases. Protein Sci. 15, 935–941.
Patel, S. S. & Picha, K. M. (2000): Structure and function of hexameric helicases. Annu. Rev. Biochem. 69, 651–697.
Pereto, J., Lopez-Garcia, P. & Moreira, D. (2004): Ancestral lipid biosynthesis and early membrane evolution. Trends Biochem. Sci. 29, 469–477.
Perzov, N., Padler-Karavani, V., Nelson, H. & Nelson, N. (2001): Features of V-ATPases that distinguish them from F-ATPases. FEBS Lett. 504, 223–228.
Philippe, H. & Laurent, J. (1998): How good are deep phylogenetic trees? Curr. Opin. Genet. Dev. 8, 616–623.
Pirttimaa, M. J., Paatero, A. O., Frilander, M. J. & Bamford, D. H. (2002): Nonspecific nucleoside triphosphatase P4 of double-stranded RNA bacteriophage phi6 is required for single-stranded RNA packaging and transcription. J. Virol. 76, 10122–10127.
Pohlschroder, M., Hartmann, E., Hand, N. J., Dilks, K. & Haddad, A. (2005): Diversity and evolution of protein translocation. Annu. Rev. Microbiol. 59, 91–111.
Schafer, I. B. et al. (2006): Crystal structure of the archaeal A1A0 ATP synthase subunit B from Methanosarcina mazei Go1: implications of nucleotide-binding differences in the major A1A0 subunits A and B. J. Mol. Biol. 358, 725–740.
Skordalakes, E. & Berger, J. M. (2003): Structure of the Rho transcription terminator: mechanism of mRNA recognition and helicase loading. Cell 114, 135–146.
Skordalakes, E. & Berger, J. M. (2006): Structural insights into RNA-dependent ring closure and ATPase activation by the Rho termination factor. Cell 127, 553–564.
Song CF, Papachristos K, Rawson S, Huss M, Wieczorek H, Paci E, et al. (2013): Flexibility within the Rotor and Stators of the Vacuolar H+-ATPase . PLoS ONE 8(12): e82207.
Stewart, A. G., Laming, E. M., Sobti, M. & Stock, D. (2014): Rotary ATPases—dynamic molecular machines. Curr. Opin. Struct Biol. 25, 40–48.
Supekova, L., Supek, F. & Nelson, N. (1995): The Saccharomyces cerevisiae VMA10 is an intron-containing gene encoding a novel 13-kDa subunit of vacuolar H+-ATPase. J. Biol. Chem. 270, 13726–13732.
Tato, I., Zunzunegui, S., de la Cruz, F. & Cabezon, E. (2005): TrwB, the coupling protein involved in DNA transport during bacterial conjugation, is a DNA-dependent ATPase. Proc. Natl Acad. Sci. USA 102, 8156–8161.
Vetsigian, K., Woese, C. & Goldenfeld, N. (2006): Collective evolution and the genetic code. Proc. Natl Acad. Sci. USA 103, 10696–10701.
Vogler, A. P., Homma, M., Irikura, V. M. & Macnab, R. M. (1991): Salmonella typhimurium mutants defective in flagellar filament regrowth and sequence similarity of FliI to F0F1, vacuolar, and archaebacterial ATPase subunits. J. Bacteriol. 173, 3564–3572.
Walker, J. E. (1998): ATP synthesis by rotary catalysis (Nobel lecture). Angew. Chem. Int. Ed. Engl. 37, 2309–2319.
Wall, D. & Kaiser, D. (1999): Type IV pili and cell motility. Mol. Microbiol. 32, 1–10.
Wienisch M, Klingauf J (2006): Vesicular proteins exocytosed and subsequently retrieved by compensatory endocytosis are nonidentical. Nature Neuroscience. 9 (8): 1019–27.
Wilharm, G., Dittmann, S., Schmid, A. & Heesemann, J. (2007): On the role of specific chaperones, the specific ATPase, and the proton motive force in type III secretion. Int. J. Med. Microbiol. 297, 27–36.
Wilharm, G., Lehmann, V., Neumayer, W., Trcek, J. & Heesemann, J. (2004): Yersinia enterocolitica type III secretion: evidence for the ability to transport proteins that are folded prior to secretion. BMC Microbiol. 4, 27.
Williams, T. A. et al. (2017): Integrative modeling of gene and genome evolution roots the archaeal tree of life. Proc. Natl Acad. Sci. USA 114, E4602–E4611.
Woese, C. R. (2002): On the evolution of cells. Proc. Natl Acad. Sci. USA 99, 8742–8747.
Yu, X. & Egelman, E. H. (1997): The RecA hexamer is a structural homologue of ring helicases. Nature Struct. Biol. 4, 101–104.